Procedimientos para generar genotecas muy diversas.
Un procedimiento para generar una genoteca de ácidos nucleicos,
comprendiendo dicho procedimiento:
(a) proporcionar una población de moldes de ácidos nucleicos monocatenarios, comprendiendo cada uno de dichos moldes una secuencia codificante y una secuencia promotora unida operativamente;
(b) hibridar con dicha población de moldes de ácidos nucleicos monocatenarios una mezcla de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios, siendo dichos fragmentos de una longitud más corta que dichos moldes de ácidos nucleicos;
(c) poner en contacto cada uno de los productos de hibridación de la etapa (b) tanto con una polimerasa de ADN que carece de actividad de desplazamiento de hebra como con una ligasa de ADN en unas condiciones en las que dichos fragmentos hibridados aleatoriamente actúen como cebadores para completar unas segundas hebras de ácidos nucleicos que son sustancialmente complementarias a dichos moldes de ácidos nucleicos, produciendo así una población de hebras codificantes con mutaciones que codifican para hebras que difieren entre sí en número, posición e identidad de dichas mutaciones; y
(d) poner en contacto los productos de la etapa (c) con una polimerasa de ARN para generar una genoteca de ARN, siendo transcrita dicha genoteca a partir de dichas segundas hebras de ácidos nucleicos.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US1999/014776.
Solicitante: BRISTOL-MYERS SQUIBB COMPANY.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: ROUTE 206 AND PROVINCE LINE ROAD PRINCETON, NJ 08543-4000 ESTADOS UNIDOS DE AMERICA.
Inventor/es: WAGNER, RICHARD, WRIGHT, MARTIN, C., KREIDER,Brent.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/09 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Tecnología del ADN recombinante.
- C12N15/10 C12N 15/00 […] › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C12N15/74 C12N 15/00 […] › Vectores o sistemas de expresión especialmente adaptados a huéspedes procariotas distintos a E. coli, p. ej. Lactobacillus, Micromonospora.
- C12P19/34 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
- C12P21/06 C12P […] › C12P 21/00 Preparación de péptidos o de proteínas (proteína monocelular C12N 1/00). › preparados por hidrólisis de un enlace peptídico, p. ej. hidrolizados.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2381824_T3.pdf
Fragmento de la descripción:
Procedimientos para generar genotecas muy diversas
Antecedentes de la invención En general, esta invención se refiere a procedimientos para generar y alterar genotecas recombinantes.
La capacidad de aislar una secuencia de ácidos nucleicos de aminoácidos deseada requiere la disponibilidad de genotecas recombinantes con un número y diversidad suficientes, de forma que una especie en particular esté representada en la genoteca y pueda ser identificada mediante una o más técnicas de cribado. Dichas genotecas facilitan el aislamiento de compuestos útiles, incluyendo terapéuticos, diagnósticos de investigación y reactivos agriculturales, así como sus secuencias codificantes.
Además, las genotecas deseables pueden diseñarse específicamente para que contengan grandes cifras de posibles variantes de un único compuesto. Este tipo de genotecas pueden usarse para cribar versiones mejoradas del compuesto, por ejemplo, de una variante de un compuesto con una eficacia terapéutica optimizada.
Para estas o cualquier otra aplicación, las metodologías generales para incrementar la diversidad de las genotecas son muy útiles y representan un foco importante en la industria de diseño de proteínas.
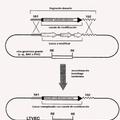
Sumario de la invención En general, la presente invención presenta un procedimiento para generar una genoteca de ácidos nucleicos, implicando el procedimiento: (a) proporcionar una población de moldes de ácidos nucleicos monocatenarios, incluyendo cada uno de los moldes una secuencia codificante y una secuencia promotora unida operativamente; (b) hibridar con la población de moldes de ácidos nucleicos monocatenarios una mezcla de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios, siendo los fragmentos de una longitud más corta que el molde de ácido nucleico; (c) poner en contacto cada uno de los productos de hibridación de la etapa (b) tanto con una polimerasa de ADN que carezca de actividad de desplazamiento de hebra como con una ligasa de ADN en unas condiciones en las que los fragmentos actúen como cebadores para completar una segunda hebra de ácido nucleico que es sustancialmente complementaria del molde de ácido nucleico; y (d) poner en contacto los productos de la etapa (c) con una polimerasa de ARN para generar una genoteca de ARN, siendo transcrita la genoteca a partir de la segunda hebra de ácido nucleico.
En las formas de realización preferidas, el procedimiento se usa para introducir una o más mutaciones en la genoteca; la mezcla de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios se genera escindiendo una molécula de ácido nucleico bicatenario; la mezcla de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios se genera mediante la síntesis de oligonucleótidos aleatorios; el molde del ácido nucleico monocatenario se genera usando un fago M13 portador del ácido nucleico, mediante la digestión de una hebra de un molde de ácido nucleico bicatenario usando exonucleasa del gen VI o exonucleasa lambda, capturando una hebra simple de ácido nucleico biotinilada usando estreptavidina, o mediante una transcripción inversa del ARN; la mezcla de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios incluye al menos 100 especies diferentes de fragmentos de ácidos nucleicos; la etapa (b) se lleva a cabo usando entre 1 y aproximadamente 1.000 fragmentos por molde de ácido nucleico monocatenario; se usa una hebra simple del producto de la etapa (c) como molde de ácido nucleico y se repiten las etapas (b) y (c) ; las etapas (b) y (c) se repiten usando, en cada ronda, el producto de la etapa (c) como molde de ácido nucleico; el procedimiento implica adicionalmente proporcionar uno o más fragmentos de ácidos nucleicos monocatenarios que forman homodúplex con el molde de ácido nucleico monocatenario y llevar a cabo la etapa (b) en presencia de los fragmentos formadores del homodúplex; el promotor es un promotor T7; la polimerasa de ADN es la polimerasa de ADN T4; el procedimiento implica además amplificar el producto de la etapa (c) antes de dicha etapa de contacto (d) ; el procedimiento implica además la etapa de: (e) traducir la genoteca de ARN para generar una genoteca de proteínas; el procedimiento implica además la etapa de: (e) unir al extremo 3' de la secuencia codificante de cada uno de sustancialmente todos los miembros de la genoteca de ARN a una molécula aceptora de aminoácidos; y el procedimiento implica además la etapa de (f) traducir la genoteca de ARN para generar una genoteca de fusión de ARN-proteínas.
En un segundo aspecto, la invención presenta un procedimiento para reducir la variación en la secuencia en una población de moléculas de ácidos nucleicos, implicando el procedimiento: (a) proporcionar una primera población de moldes de ácidos nucleicos monocatenarios de secuencia variable, incluyendo cada uno de sustancialmente todos los moldes una secuencia codificante y una secuencia promotora unida operativamente; (b) hibridar los miembros de la primera población con una segunda población de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios, siendo los fragmentos de una longitud más corta que el molde de ácido nucleico y teniendo los fragmentos una secuencia sustancialmente idéntica; (c) poner en contacto los productos de hibridación de la etapa (b) tanto con una polimerasa de ADN que carece de actividad de desplazamiento de hebra como con una ligasa de ADN en unas condiciones en las que los fragmentos actúen como cebadores para completar una segunda hebra de ácido nucleico que es sustancialmente complementaria al molde de ácido nucleico; y (d) poner en contacto los productos de la etapa (c) con una polimerasa de ARN para generar una población de moléculas de ARN, siendo transcrita la población de moléculas de ARN a partir de la segunda hebra de ácido nucleico y teniendo una variación reducida en la secuencia con respecto a la primera población de moldes de ácidos nucleicos monocatenarios.
En formas de realización preferidas, el procedimiento se usa para eliminar una o más mutaciones de la primera población de moldes de ácidos nucleicos monocatenarios; la etapa (b) implica la hibridación de la primera población de moldes de ácidos nucleicos monocatenarios con dos o más poblaciones diferentes de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios; la segunda población de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios se genera escindiendo una molécula de ácido nucleico bicatenario; la segunda población de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios se genera mediante la síntesis de oligonucleótidos aleatorios; el molde de ácido nucleico monocatenario se genera usando un fago M13 portador del ácido nucleico, mediante la digestión de una hebra de un molde de ácido nucleico bicatenario usando exonucleasa del gen VI o exonucleasa lambda, capturando una hebra simple de ácido nucleico biotinilada usando estreptavidina, o mediante una transcripción inversa del ARN; la etapa (b) se lleva a cabo usando entre 1 y aproximadamente 1.000 fragmentos de ácidos nucleicos monocatenarios por molde de ácido nucleico monocatenario; se usa una hebra simple del producto de la etapa (c) como molde de ácido nucleico y se repiten las etapas (b) y (c) ; las etapas (b) y (c) se repiten usando, en cada ronda, el producto de la etapa (c) como molde de ácido nucleico; el promotor es un promotor T7; la polimerasa de ADN es la polimerasa de ADN T4; el procedimiento implica además amplificar el producto de la etapa (c) antes de dicha etapa de contacto (d) ; el procedimiento implica además la etapa de: (e) traducir la población de moléculas de ARN para generar una genoteca de proteínas; el procedimiento implica además la etapa de: (e) unir al extremo 3' de la secuencia codificante de cada uno de sustancialmente todos los miembros de la población de moléculas de ARN a una molécula aceptora de aminoácidos; y el procedimiento implica además la etapa de (f) traducir la población de moléculas de ARN para generar una genoteca de fusión de ARN-proteínas.
En un tercer aspecto, la invención presenta un procedimiento para generar una genoteca de ácidos nucleicos, implicando el procedimiento: (a) proporcionar una población de moldes de ácidos nucleicos monocatenarios, incluyendo cada molde una secuencia codificante; (b) proporcionar una población de moléculas de ácidos nucleicos monocatenarios de secuencia variable, siendo la población de moldes de ácidos nucleicos monocatenarios y la población de moléculas de ácidos nucleicos monocatenarios... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento para generar una genoteca de ácidos nucleicos, comprendiendo dicho procedimiento:
(a) proporcionar una población de moldes de ácidos nucleicos monocatenarios, comprendiendo cada uno de dichos moldes una secuencia codificante y una secuencia promotora unida operativamente;
(b) hibridar con dicha población de moldes de ácidos nucleicos monocatenarios una mezcla de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios, siendo dichos fragmentos de una longitud más corta que dichos moldes de ácidos nucleicos;
(c) poner en contacto cada uno de los productos de hibridación de la etapa (b) tanto con una polimerasa de ADN que carece de actividad de desplazamiento de hebra como con una ligasa de ADN en unas condiciones en las que dichos fragmentos hibridados aleatoriamente actúen como cebadores para completar unas segundas hebras de ácidos nucleicos que son sustancialmente complementarias a dichos moldes de ácidos nucleicos, produciendo así una población de hebras codificantes con mutaciones que codifican para hebras que difieren entre sí en número, posición e identidad de dichas mutaciones; y
(d) poner en contacto los productos de la etapa (c) con una polimerasa de ARN para generar una genoteca de ARN, siendo transcrita dicha genoteca a partir de dichas segundas hebras de ácidos nucleicos.
2. El procedimiento de la reivindicación 1, en el que dicho procedimiento se usa para introducir una o más mutaciones en dicha genoteca.
3. Un procedimiento para reducir la variación en la secuencia en una población de moléculas de ácidos nucleicos, comprendiendo dicho procedimiento:
(a) proporcionar una primera población de moldes de ácidos nucleicos monocatenarios de secuencia variable, comprendiendo cada uno de sustancialmente todos de dichos moldes una secuencia codificante y una secuencia promotora unida operativamente;
(b) hibridar con los miembros de dicha primera población una segunda población de fragmentos de ácidos nucleicos monocatenarios, siendo dichos fragmentos de una longitud más corta que dichos moldes de ácidos nucleicos y siendo dichos fragmentos sustancialmente complementarios de dichos moldes de ácidos nucleicos, en los que una subpoblación de dicha segunda población comprende una secuencia sustancialmente idéntica e hibrida con una región de secuencia variable;
(c) poner en contacto los productos de hibridación de la etapa (b) tanto con una polimerasa de ADN que carece de actividad de desplazamiento de hebra como con una ligasa de ADN en unas condiciones en las que dichos fragmentos actúen como cebadores para completar unas segundas hebras de ácidos nucleicos que son sustancialmente complementarias de dichos moldes de ácidos nucleicos; y
(d) poner en contacto los productos de la etapa (c) con una polimerasa de ARN para generar una tercera población de moléculas de ARN, siendo dicha tercera población de moléculas de ARN transcrita a partir de dichas segundas hebras de ácidos nucleicos y teniendo una variación reducida en la secuencia con respecto a dicha primera población de moldes de ácidos nucleicos monocatenarios.
4. El procedimiento de la reivindicación 3, en el que dicho procedimiento se usa para eliminar una o más mutaciones de dicha primera población de moldes de ácidos nucleicos monocatenarios.
5. El procedimiento de la reivindicación 1 ó 3, en el que dicha mezcla de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios se genera escindiendo moléculas de ácidos nucleicos bicatenarios o mediante la síntesis de oligonucleótidos aleatorios.
6. El procedimiento de la reivindicación 1 ó 3, en el que dicha mezcla de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios comprende al menos aproximadamente 100 especies diferentes de fragmentos de ácidos nucleicos.
7. El procedimiento de la reivindicación 1 ó 3, que comprende además la generación de hebras complementarias de dichas segundas hebras de ácidos nucleicos, proporcionando dichas hebras complementarias como dicha población
o dicha primera población de moldes de ácidos nucleicos monocatenarios, y repitiendo las etapas (b) y (c) durante una o más rondas, y repitiendo después la etapa (d) .
8. El procedimiento de la reivindicación 1, en el que dicho procedimiento comprende además proporcionar uno o más fragmentos de ácidos nucleicos monocatenarios que forman un homodúplex con dichos moldes de ácidos nucleicos monocatenarios y llevar a cabo la etapa (b) en presencia de dichos fragmentos formadores de homodúplex.
9. El procedimiento de la reivindicación 1 ó 3, en el que dicho promotor es un promotor T7.
10. El procedimiento de la reivindicación 1 ó 3, en el que dicha polimerasa de ADN es polimerasa de ADN T4.
11. El procedimiento de la reivindicación 1 ó 3, en el que dicho procedimiento comprende además amplificar dicho producto de la etapa (c) antes de dicha etapa de contacto (d) .
12. El procedimiento de la reivindicación 1 ó 3, que comprende además un procedimiento para generar una genoteca de proteínas, en donde dicho procedimiento comprende la etapa de:
(e) traducir dicha genoteca de ARN para generar una genoteca de proteínas.
13. El procedimiento de la reivindicación 1 ó 3, que comprende además un procedimiento para generar una genoteca de fusión de ARN-proteínas, en donde dicho procedimiento comprende las etapas de:
(e) unir una molécula aceptora de aminoácidos al extremo 3' de dicha secuencia codificante de cada uno de sustancialmente todos los miembros de dicha genoteca de ARN; y opcionalmente
(f) traducir dicha genoteca de ARN para generar una genoteca de fusión de ARN-proteínas.
14. Un procedimiento para generar una genoteca de ácidos nucleicos, comprendiendo dicho procedimiento:
(a) proporcionar una primera población de moldes de ácidos nucleicos monocatenarios, comprendiendo cada uno de dichos moldes una secuencia codificante;
(b) proporcionar una segunda población de moléculas de ácidos nucleicos monocatenarios de secuencia variable, siendo dicha primera población de moldes de ácidos nucleicos monocatenarios y dicha segunda población de moléculas de ácidos nucleicos monocatenarios de secuencia variable sustancialmente complementarias;
(c) hibridar dicha primera población de moldes de ácidos nucleicos monocatenarios con dicha segunda población de moléculas de ácidos nucleicos monocatenarios de secuencia variable en unas condiciones suficientes para formar dúplex; y
(d) poner en contacto dichos dúplex con una o más enzimas de escisión/reparación en unas condiciones que permitan a dichas enzimas corregir los pares de bases mal apareados en dichos dúplex.
15. El procedimiento de la reivindicación 14, en donde dicho procedimiento comprende además, después de la etapa (d) , proporcionar una nueva población de moldes de ácidos nucleicos monocatenarios que comprenden una secuencia codificante, derivando dichos moldes del producto de la etapa (d) , sustituyendo dicha nueva población de moldes por dicha primera población de moldes, y repitiendo las etapas (c) , y (d) durante una o más rondas.
16. Un procedimiento para generar una genoteca de ácidos nucleicos, comprendiendo dicho procedimiento:
(a) proporcionar una población de moldes de ácidos nucleicos monocatenarios, comprendiendo cada uno de dichos moldes una secuencia codificante;
(b) hibridar con dicha población de moldes de ácidos nucleicos monocatenarios una mezcla de fragmentos de ácidos nucleicos monocatenarios sustancialmente complementarios, siendo dichos fragmentos de una longitud más corta que dichos moldes de ácidos nucleicos;
(c) poner en contacto cada uno de los productos de hibridación de la etapa (b) tanto con una polimerasa de ADN que carece de actividad de desplazamiento de hebra como con una ligasa de ADN en unas condiciones en las que dichos fragmentos actúen como cebadores para completar una segunda hebra de ácidos nucleicos que es sustancialmente complementaria de dichos moldes de ácidos nucleicos; y
(d) poner en contacto los productos de la etapa (c) con una o más enzimas de escisión/reparación en unas condiciones que permitan a dichas enzimas corregir los pares de bases mal apareados en dichos productos.
17. El procedimiento de la reivindicación 16, en donde dicho procedimiento comprende además las etapas de: proporcionar una nueva población de moldes de ácidos nucleicos monocatenarios que comprenden una secuencia codificante y que derivan del producto de la etapa (d) , sustituir dicha nueva población de moldes por dicha población de moldes de ácidos nucleicos monocatenarios y repetir las etapas (b) - (d) durante una o más rondas.
18. El procedimiento de la reivindicación 14 ó 16 en el que dicho contacto con dichas enzimas de escisión/reparación se lleva a cabo in vitro.
19. El procedimiento de la reivindicación 14 ó 16, en el que dicho contacto con dichas enzimas de escisión/reparación se lleva a cabo en una célula bacteriana.
20. El procedimiento de la reivindicación 1, 3, 14 ó 16 en el que dichos moldes de ácidos nucleicos monocatenarios se generan usando un fago M13 portador de dichos moldes de ácidos nucleicos, mediante digestión de una hebra de los moldes de ácidos nucleicos bicatenarios usando exonucleasa del gen VI o exonucleasa lambda, mediante la captura de hebras simples de ácidos nucleicos biotiniladas usando estreptavidina, o mediante una transcripción inversa del ARN.
21. El procedimiento de la reivindicación 1, 3, 14 ó 16, en el que la etapa (b) se lleva a cabo usando entre 1 y aproximadamente 1.000 moléculas de ácidos nucleicos monocatenarios de secuencia variable o fragmentos de ácidos nucleicos monocatenarios por cada uno de dichos moldes de ácidos nucleicos monocatenarios.
22. El procedimiento de la reivindicación 14 ó 16, en donde dicho procedimiento comprende además la etapa de:
(e) amplificar dicho producto de la etapa (d) .
23. El procedimiento de la reivindicación 14 ó 16, en el que cada una de dichas secuencias codificantes está unida operativamente a una secuencia promotora.
24. El procedimiento de la reivindicación 23, en el que dicho procedimiento comprende además la etapa de:
(e) transcribir los productos de la etapa (d) para generar una genoteca de ARN.
25. El procedimiento de la reivindicación 24, que comprende además un procedimiento para generar una genoteca de proteínas, en donde dicho procedimiento comprende la etapa de:
(f) traducir dicha genoteca de ARN para generar una genoteca de proteínas.
26. El procedimiento de la reivindicación 24, que comprende además un procedimiento para generar una genoteca de fusión de ARN-proteínas, en donde dicho procedimiento comprende las etapas de:
(f) unir una molécula aceptora de aminoácidos al extremo 3' de dicha secuencia codificante de cada uno de sustancialmente todos los miembros de dicha genoteca de ARN; y opcionalmente (g) traducir dicha genoteca de ARN para generar una genoteca de fusión de ARN-proteínas.
Patentes similares o relacionadas:
Sistema de expresión bacteriano cistrónico doble, del 25 de Marzo de 2020, de LUPIN LIMITED: Un procedimiento para la producción de un anticuerpo o un fragmento del mismo que comprende las etapas de: (i) transformar una célula hospedadora bacteriana […]
Partículas de transducción no replicativas y sistemas indicadores basados en partículas de transducción, del 15 de Enero de 2020, de Geneweave Biosciences Inc: Un sistema de empaquetamiento de células bacterianas para empaquetar una molécula de ácido nucleico indicadora en una partícula de transducción no replicativa (NRTP) para su introducción […]
Método de construcción de biblioteca de péptidos y vectores relacionados, del 1 de Enero de 2020, de Hunan Zonsen Peplib Biotech Co., Ltd: Método integrado de construcción de una biblioteca de péptidos completa que contiene todos los péptidos posibles para el tamaño específico, comprendiendo el método: (i) […]
 Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Transporte de proteínas basado en bacterias, del 14 de Agosto de 2019, de Universität Basel: Un vector que comprende, en la dirección 5' a 3': un promotor; una primera secuencia de ADN que codifica una señal de transporte procedente […]
Sistema de expresión y secreción, del 26 de Junio de 2019, de F. HOFFMANN-LA ROCHE AG: Una molécula de ácido nucleico que codifica un primer polipéptido y un segundo polipéptido, enlazada de forma funcional a una secuencia señal que codifica […]
 Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Síntesis microbiana de aldehídos y alcoholes correspondientes, del 17 de Junio de 2019, de Easel Biotechnologies, LLC: Un método para producir un alcohol, que comprende: cultivar una pluralidad de células microbianas en un medio de fermentación, en donde las […]