Procedimiento de ajuste de resultados de un instrumento de reacción en cadena de la polimerasa (PCR).
Procedimiento para la gestión de resultados de un instrumento de reacción en cadena de la polimerasa (PCR) entiempo real,
comprendiendo el procedimiento:
calcular, a partir de resultados del instrumento de PCR en tiempo real, una señal de fluorescencia de una muestradurante un período basal del instrumento de PCR en tiempo real;
determinar si la señal de fluorescencia durante el período basal aumenta o no en, como mínimo, un ciertoporcentaje; y
marcar la muestra como una muestra de titulación potencialmente elevada, cuando la señal de fluorescenciaaumenta en, como mínimo, el porcentaje determinado.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2008/083113.
Solicitante: Grifols Therapeutics Inc.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 4101 Research Commons, 79 TW Alexander Drive Research Triangle Park, NC 27709 ESTADOS UNIDOS DE AMERICA.
Inventor/es: PAUL,JUSTIN M, BEAMES,BURTON D.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12M1/00 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12M EQUIPOS PARA ENZIMOLOGIA O MICROBIOLOGIA (instalaciones para la fermentación de estiércoles A01C 3/02; conservación de partes vivas de cuerpos humanos o animales A01N 1/02; aparatos de cervecería C12C; equipos para la fermentación del vino C12G; aparatos para preparar el vinagre C12J 1/10). › Equipos para enzimología o microbiología.
- C12M1/36 C12M […] › C12M 1/00 Equipos para enzimología o microbiología. › que incorporan un mecanismo de control en función del tiempo o de las condiciones del medio, p. ej. fermentadores controlados automáticamente.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2424759_T3.pdf
Fragmento de la descripción:
Procedimiento de ajuste de resultados de un instrumento de reacción en cadena de la polimerasa (PCR)
SECTOR TECNICO
En general, la presente descripción se refiere a instrumentos de la reacción en cadena de la polimerasa (PCR) . Más particularmente, la invención se refiere a procedimientos para la verificación o el ajuste de los resultados del software de detección de secuencias (SDS) utilizados en los instrumentos de PCR.
TECNICA ANTERIOR
Los sistemas de reacción en cadena de la polimerasa en tiempo real (PCR) se utilizan para la amplificación de una muestra, tal como una cadena de ADN, con el fin de que sea más fácil detectar o identificar la secuencia de la muestra. Los dispositivos de PCR en tiempo real utilizan programas informáticos o software de detección de secuencias (SDS) para analizar y procesar los resultados de las pruebas de PCR. Sin embargo, en algunos casos, el SDS no es capaz de detectar muestras con precisión, lo que puede dar como resultado la identificación incorrecta de la existencia de ciertas secuencias o de la concentración de las mismas. Por ejemplo, cuando una muestra de plasma sanguíneo se analiza utilizando ciertos sistemas de PCR de Applied Biosystems, o en otros sistemas que utilizan un SDS relacionado, a veces los sistemas pueden no detectar una concentración elevada de un virus, tal como, por ejemplo, el parvovirus B19. Por consiguiente, los sistemas actuales pueden malinterpretar una muestra de plasma que contiene una titulación elevada de estos virus, que contaminarían una masa de plasma, provocando una pérdida significativa de recursos de plasma.
Por lo tanto, existe una necesidad en el estado de la técnica actual de hacer frente a estas y otras deficiencias e insuficiencias de los sistemas y procedimientos convencionales. Las mejoras en los sistemas y procedimientos convencionales, tal como se describe en la presente memoria descriptiva, son capaces de superar estas y otras deficiencias de la técnica anterior.
El documento EP1518935 da a conocer una corrección de la línea de base para mejorar la cuantificación de los ácidos nucleicos en una muestra CARACERiSTICAS DE LA INVENCION
La presente invención da a conocer procedimientos para la gestión de los resultados de un instrumento de reacción en cadena de la polimerasa en tiempo real (PCR) . Según una entre muchas realizaciones, un procedimiento dado a conocer en la presente memoria descriptiva comprende el cálculo, a partir de resultados del instrumento de PCR en tiempo real, de una señal de fluorescencia de una muestra durante el período basal del instrumento de PCR en tiempo real. El procedimiento incluye además la determinación de si la señal de fluorescencia aumenta en, como mínimo, un cierto porcentaje durante el período basal o no aumenta. Además, el procedimiento incluye el marcaje de la muestra como una muestra de titulación potencialmente elevada, cuando la señal de fluorescencia aumenta en, como mínimo, el porcentaje determinado.
Según otra forma de realización, se describe un procedimiento para analizar una muestra de sangre y comprende la cuantificación de ácido nucleico viral en la muestra de sangre utilizando un instrumento de reacción en cadena de la polimerasa en tiempo real (rtPCR) mediante la modificación de la interpretación de los datos de modo que las muestras de alta titulación no se malinterpretan. Una señal de fluorescencia se calcula a partir de los resultados de PCR proporcionados durante un período basal. Además, se determina si la señal de fluorescencia aumenta en, como mínimo, un porcentaje predeterminado durante el período basal. El procedimiento incluye además marcar la muestra de sangre como una muestra de titulación potencialmente elevada, cuando la señal de fluorescencia aumenta en, como mínimo, el porcentaje predeterminado.
La presente invención da a conocer además software o firmware informático, programas informáticos, secuencias de código de software, y similares, instalados en un medio legible por ordenador y ejecutables mediante un dispositivo de procesamiento. Según una forma de realización, una secuencia de código de software comprende lógica configurada para calcular una señal de fluorescencia de una muestra durante un período basal de ciclos de ensayo de un instrumento de reacción en cadena de la polimerasa (PCR) . La secuencia de código de software incluye además lógica configurada para determinar si la señal de fluorescencia durante el período de referencia aumenta en, como mínimo, un cierto porcentaje o no aumenta. Además, la secuencia de código de software incluye una lógica configurada para marcar la muestra como una muestra de titulación potencialmente elevada, cuando la señal de fluorescencia aumenta en, como mínimo, el porcentaje determinado.
Otras características, ventajas y realizaciones de la presente descripción, que no se describen expresamente en la presente memoria descriptiva, se harán evidentes para un técnico en la materia tras el examen de la siguiente descripción detallada y los dibujos adjuntos. Se pretende que tales realizaciones implícitas de la presente descripción queden incluidas en la presente memoria descriptiva.
BREVE DESCRIPCION DE LOS DIBUJOS
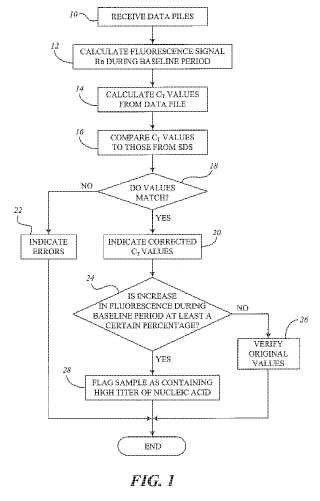
La figura 1 es un diagrama de flujo que describe un proceso para el ajuste de los resultados de los datos a partir de un dispositivo de PCR según una realización.
La figura 2 es un diagrama de flujo que describe un proceso para el cálculo del ciclo de umbral según una realización.
DESCRIPCION DETALLADA
Los dispositivos/sistemas utilizados actualmente para procesar e interpretar los datos de la reacción en cadena de polimerasa (PCR) , pueden identificar incorrectamente la concentración de algunos ácidos nucleicos en las muestras. Este problema puede ser provocado por cómo se procesan los resultados de las pruebas de PCR en el software de detección de secuencias (SDS) asociado con el análisis de PCR. Por ejemplo, varios sistemas de PCR, tales como muchos de los sistemas de PCR en tiempo real de Applied Biosystems, utilizan SDS que puede fallar para detectar con precisión muestras de plasma sanguíneo que contienen altas concentraciones de virus, por ejemplo, el parvovirus B19. Debido a la manera en la que se detecta la fluorescencia, es posible que una muestra de plasma sanguíneo que contiene una titulación elevada de virus pudiera ser pasada por alto como limpia. En particular, se ha observado que los niveles de fluorescencia durante un período basal son elevados, de modo que cuando se comparan los niveles de fluorescencia de la muestra con los niveles basales, no parece haber ningún aumento significativo por encima de un nivel de umbral que indique una concentración de un virus. Por lo tanto, es necesaria una modificación para superar las deficiencias del software y anular los resultados erróneos, para identificar correctamente la concentración de un virus, si lo hubiera, en una muestra de plasma.
La presente invención describe procedimientos de detección de altas concentraciones de ácido nucleico que se amplifican utilizando instrumentos convencionales de PCR. Los ácidos nucleicos pueden ser detectados, por ejemplo, utilizando una sonda fluorogénica, tal como una sonda TaqMan®. En la presencia de la secuencia de ácido nucleico diana, la sonda se hidroliza durante cada ciclo de PCR y se mide un aumento en la fluorescencia. El SDS puede utilizar un algoritmo para separar la fluorescencia en las componentes de longitudes de onda específicas para los colorantes fluorescentes. Los primeros ciclos de PCR, por ejemplo, los ciclos 3º a 15º, se pueden configurar específicamente por un usuario para establecer los valores a partir de los cuales se determina un nivel de fluorescencia basal. El usuario define además un valor de umbral asignado para cada ciclo que refleja un cierto nivel de fluorescencia mayor que el nivel basal calculado. El ciclo umbral (CT) indica el número del ciclo de PCR durante el cual la fluorescencia generada dentro de una reacción supera el umbral e indica además la detección del ácido nucleico. Sin embargo, debe señalarse que las muestras con alta concentración de ácidos nucleicos tienden a amplificarse durante el período basal. De este modo, el SDS de algunos sistemas de PCR convencionales lee esta fluorescencia como fondo e interpreta incorrectamente estas muestras de alta concentración como negativas para el ácido nucleico diana, cuando en realidad puede haber una concentración elevada de la secuencia diana. Los procedimientos de la presente descripción compensan estos resultados inexactos proporcionando una lectura más precisa, si es necesario, o verificando la exactitud de los resultados anteriores.
La lógica descrita en la... [Seguir leyendo]
Reivindicaciones:
1. Procedimiento para la gestión de resultados de un instrumento de reacción en cadena de la polimerasa (PCR) en tiempo real, comprendiendo el procedimiento:
calcular, a partir de resultados del instrumento de PCR en tiempo real, una señal de fluorescencia de una muestra durante un período basal del instrumento de PCR en tiempo real;
determinar si la señal de fluorescencia durante el período basal aumenta o no en, como mínimo, un cierto porcentaje; y
marcar la muestra como una muestra de titulación potencialmente elevada, cuando la señal de fluorescencia aumenta en, como mínimo, el porcentaje determinado.
2. Procedimiento, según la reivindicación 1, en el que el porcentaje es del 20%.
3. Procedimiento, según la reivindicación 1, que comprende además:
verificar que los valores de fluorescencia originales detectados por el software de detección de secuencias (SDS) asociado con la PCR son sustancialmente precisos cuando se determina que la señal de fluorescencia durante el período basal no aumenta, como mínimo, el porcentaje determinado.
4. Procedimiento, según la reivindicación 1, que comprende además:
calcular el ciclo umbral (CT) a partir de los valores de fluorescencia originales detectado por el software de detección de secuencias (SDS) asociado con la PCR, y comparar el CT con los valores de ciclo umbral detectados por el SDS para determinar si hay coincidencia.
5. Procedimiento, según la reivindicación 4, que comprende además:
indicar el CT calculado a partir de los valores de fluorescencia originales como los valores correctos cuando hay coincidencia, e indicar la aparición de error cuando no hay coincidencia.
6. Procedimiento, según la reivindicación 4, en el que el cálculo de CT comprende además: calcular una señal de fluorescencia como una proporción de FAM / ROX; calcular una fluorescencia basal utilizando un procedimiento de regresión lineal, calcular una señal diferencial entre la señal de fluorescencia y la fluorescencia basal en cada ciclo; calcular la pendiente y la intersección de la señal diferencial entre cada ciclo y el ciclo anterior, y calcular la CT como el ciclo fraccional en el que la señal diferencial cruza un umbral.
7. Procedimiento para analizar una muestra de sangre, procedimiento que comprende:
cuantificar una muestra de sangre utilizando un instrumento de reacción en cadena de la polimerasa en tiempo real (PCR) para proporcionar resultados de PCR; calcular una señal de fluorescencia a partir de los resultados de PCR proporcionados durante un período basal; determinar si la señal de fluorescencia aumenta, como mínimo, un porcentaje predeterminado durante el período
basal, y marcar la muestra de sangre como una muestra de titulación potencialmente elevada, cuando la señal de fluorescencia aumenta en, como mínimo, el porcentaje predeterminado.
8. Procedimiento, según la reivindicación 7, que comprende además:
verificar que los resultados de la PCR son sustancialmente precisos cuando se determina que la señal de fluorescencia durante el período basal no aumenta, como mínimo, en el porcentaje predeterminado.
9. Procedimiento, según la reivindicación 7, que comprende además:
calcular el ciclo umbral (CT) a partir de los resultados de la PCR, y
comparar el CT con los valores de ciclo umbral detectados por el software de detección de secuencias (SDS) asociado con la PCR para determinar si hay coincidencia.
10. Procedimiento, según la reivindicación 9, que comprende además: 10 indicar el CT calculado a partir de los resultados de PCR como valores correctos cuando hay coincidencia; e indicar la aparición de error cuando no hay coincidencia. 15 11. Procedimiento, según la reivindicación 9, en el que el cálculo del CT comprende además: calcular una señal de fluorescencia; calcular una fluorescencia basal utilizando un procedimiento de regresión lineal; 20 calcular una señal diferencial entre la señal de fluorescencia y la fluorescencia basal en cada ciclo;
calcular la pendiente y la intersección de la señal diferencial entre cada ciclo y el ciclo anterior, y 25 calcular CT como el ciclo fraccional en el que la señal diferencial cruza un umbral.
12. Medio legible por ordenador que incorpora una secuencia de código de software que es ejecutable por un dispositivo de procesamiento, secuencia de código de software que comprende:
una lógica configurada para calcular una señal de fluorescencia de una muestra durante un período basal de ciclos de ensayo de un instrumento de reacción en cadena de la polimerasa (PCR) ;
una lógica configurada para determinar si la señal de fluorescencia durante el período basal aumenta o no en, como mínimo, un cierto porcentaje; y
una lógica configurada para marcar la muestra como una muestra de titulación potencialmente elevada, cuando la señal de fluorescencia aumenta en, como mínimo, el porcentaje determinado.
13. Medio legible por ordenador, según la reivindicación 12, en el que el porcentaje es del 20%. 40
14. Medio legible por ordenador, según la reivindicación 12, que comprende además:
una lógica configurada para verificar que los valores de fluorescencia originales detectados por el software de detección de secuencias (SDS) asociado con la PCR son sustancialmente precisos cuando se determina que la 45 señal de fluorescencia durante el período basal no aumenta en, como mínimo, el porcentaje determinado.
15. Medio legible por ordenador, según la reivindicación 12, que comprende además: una lógica configurada para calcular el ciclo umbral (CT) a partir de los valores de fluorescencia originales detectados 50 por el software de detección de secuencias (SDS) asociado con la PCR; y una lógica configurada para comparar el CT con los valores de ciclo umbral detectados por el SDS para determinar si hay coincidencia. 55 16. Medio legible por ordenador, según la reivindicación 15, que comprende además: una lógica configurada para indicar el CT calculado a partir de los valores de fluorescencia originales como los valores correctos cuando hay coincidencia; y 60 una lógica configurada para indicar la aparición de error cuando no hay coincidencia.
17. Medio legible por ordenador, según la reivindicación 15, en el que la lógica configurada para calcular CT está configurada además para:
calcular una señal de fluorescencia;
calcular la fluorescencia basal utilizando un procedimiento de regresión lineal; calcular una señal diferencial entre la señal de fluorescencia y la fluorescencia basal en cada ciclo; 5 calcular la pendiente y la intersección de la señal diferencial entre cada ciclo y el ciclo anterior; y calcular el CT como el ciclo fraccional en el que la señal diferencial cruza un umbral.
18. Medio legible por ordenador, según la reivindicación 12, en el que la secuencia de código de software está 10 incorporada en el software de detección de secuencias.
19. Medio legible por ordenador, según la reivindicación 12, en el que la secuencia de código de software está incorporada en un dispositivo independiente configurado para recibir resultados de la PCR a partir de un software de detección de secuencias asociado con la PCR.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]