Polipéptidos con actividad celulasa.
Polipéptidos con actividad celulasa.
La presente invención se refiere principalmente a un polipéptido con actividad celulasa para su uso en procesos de degradación de biomasa.
La invención también se refiere a fragmentos funcionales del polipéptido, así como a los polinucleótidos que codifican dichos polipéptidos y/o fragmentos.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201430313.
Solicitante: ABENGOA RESEARCH, S.L.
Nacionalidad solicitante: España.
Inventor/es: SANTERO SANTURINO,EDUARDO, TERRÓN GONZÁLEZ,Laura, SAADEDDIN,Anas, GÓMEZ DEL PULGAR VILLANUEVA,Eva María.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N1/21 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 1/00 Microorganismos, p.ej. protozoos; Composiciones que los contienen (preparaciones de uso médico que contienen material de protozoos, bacterias o virus A61K 35/66, de algas A61K 36/02, de hongos A61K 36/06; preparación de composiciones de uso médico que contienen antígenos o anticuerpos bacterianos, p. ej. vacunas bacterianas, A61K 39/00 ); Procesos de cultivo o conservación de microorganismos, o de composiciones que los contienen; Procesos de preparación o aislamiento de una composición que contiene un microorganismo; Sus medios de cultivo. › modificados por la introducción de material genético extraño.
- C12N15/74 C12N […] › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Vectores o sistemas de expresión especialmente adaptados a huéspedes procariotas distintos a E. coli, p. ej. Lactobacillus, Micromonospora.
- C12N9/42 C12N […] › C12N 9/00 Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas. › actúan sobre los enlaces beta-glucosídicos-1,4, p. ej. celulasa.
PDF original: ES-2545161_A1.pdf

Fragmento de la descripción:
Polipéptidos con actividad celulasa CAMPO DE LA INVENCIÓN
La presente invención pertenece al campo de las enzimas con actividad celulasa para la degradación de biomasa, más concretamente, se refiere a polipéptidos con actividad celulolítica o a fragmentos funcionales o variantes de los mismos, así como a los polinucleótidos que codifican dichos polipéptidos o fragmentos. La presente invención también se relaciona con una construcción génica, un vector de expresión y una célula huésped que comprenden dichos polinucleótidos, así como con un método para producir los polipéptidos, variantes o fragmentos de la invención. Por último, la invención se relaciona con el uso de los polipéptidos de la invención, fragmentos funcionales o variantes de los mismos para la degradación de biomasa, tanto para su aplicación en procedimientos para la producción de azúcares fermentables como para la producción de bioproductos por transformación química de estos azúcares, preferiblemente por fermentación microbiana de los mismos. La presente invención también incluye una composición con capacidad de degradar la biomasa que comprende al menos un polipéptido, al menos un fragmento funcional del mismo o al menos una variante del mismo de acuerdo con la presente invención y al menos algún elemento adicional que favorezca la degradación de biomasa.
ANTECEDENTES DE LA INVENCIÓN
Las fracciones fermentables de la biomasa incluyen la celulosa y hemicelulosa. La celulosa consiste en largas cadenas insolubles de glucosa unidas covalentemente mediante enlaces beta-1,4. La hemicelulosa es una fracción heterogénea de biomasa compuesta de xilosa y azúcares menores de 5 y 6 carbonos. Aunque es un biopolímero abundante, la celulosa es altamente cristalina, insoluble en agua y muy resistente a la despolimerización. La degradación enzimática completa de celulosa a glucosa, probablemente la materia prima más deseable para procesos de fermentación, se puede conseguir mediante la acción sinérgica de 3 clases distintas de enzimas: endoglucanasas que catalizan la endohidrólisis de los enlaces (1,4)-b-D-glicosídicos de la celulosa; las exoglucanasas que llevan a cabo la hidrólisis de enlaces (1,4) en (1,4)-b-D glicanos para liberar celobiosa de los términos no reductores; y b-glucosidasas que liberan b-D-glucosa de la celobiosa (ver figura 1).
El desarrollo de un proceso económico de conversión de un producto de poco valor como la biomasa a bioetanol u otro tipo de bioproductos vía transformación química o fermentación,
requiere la optimización de varias etapas clave. En especial, una de gran importancia es la de la producción de celulasas. La utilización práctica de celulosa mediante hidrólisis con celulasas para producir glucosa requiere grandes cantidades de celulasas para despolimerizar por completo la celulosa. Además, la producción de celulasas y la hidrólisis enzimática son en proporción las etapas más costosas en este proceso.
Todas estas dificultades técnicas evidencian la necesidad de desarrollar nuevas enzimas que optimicen las tecnologías existentes para la producción de biofuel y otros bioproductos hacia sistemas más competitivos, económicos y que produzcan un menor impacto medioambiental al minimizar las emisiones de dióxido de carbono.
El hábitat del suelo es un ambiente enormemente diverso. Un gramo de suelo puede contener varios millones de bacterias pertenecientes a más de 4000 a 7000 especies diferentes. Sin embargo, solo 1% de la flora bacteriana del suelo se puede cultivar bajo condiciones estándar de laboratorio.
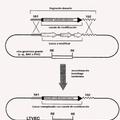
La metagenómica que consiste en la extracción, clonado y análisis de la totalidad del material genético de un hábitat determinado, ha emergido para explorar el potencial de la información genética de microorganismos no cultivables. Las librerías metagenómicas son una herramienta poderosa para el descubrimiento de nuevas actividades biológicas y ya han sido utilizadas con éxito para aislar nuevas enzimas hidrolíticas como amilasas, celulasas y xilanasas. Por ejemplo, llmberger N. ("Metagenomic cellullases highly tolerant towards the presence of ionic liquids-linking thermostability and halotolerance" Appl. Microbiol. Biotechnol. (2012)95: 135-146) describe la identificación y el aislamiento de enzimas con actividad celulasa a partir de librerías metagenómicas construidas a partir de diferentes ambientes en los que la actividad celulasa está muy presente como, por ejemplo, extractos del gusano Teredo navalis, muestras provenientes de una planta de biogás o heces de elefante.
El rastreo de librerías metagenómicas, que es un paso crítico en el aislamiento exitoso de actividades biológicas nuevas y mejoradas, se puede llevar a cabo mediante ensayos de actividad. Las librerías metagenómicas se construyen en vectores de expresión para permitir el aislamiento de actividades biológicas de secuencias génicas enteramente nuevas cuando estas se expresan con éxito en un determinado hospedador. Los ensayos cromogénicos de actividad son usados comúnmente para la detección de hidrolasas a partir de librerías metagenómicas. La figura 2 representa esquemáticamente el proceso de identificación y
caracterización de actividades enzimáticas mediante la generación de librerías metagenómicas.
Los autores de la presente invención han identificado y aislado a partir de una librería metagenómica de microorganismos del suelo, un polipéptido que muestra una alta actividad celulasa, especialmente endoglucanasa, y que posee unas características funcionales que la hacen muy apropiada para su uso en procesos industriales de degradación de biomasa y producción de bioetanol y otros bioproductos de alto valor añadido.
OBJETO DE LA INVENCIÓN
El objeto principal de la invención es un polipéptido aislado con actividad celulolítica (en adelante el polipéptido de la invención) que comprende la secuencia SEQ ID N01 o que posee un grado de homología o de identidad de al menos un 80% con la SEQ ID N01 o que está codificado por un polinucleótido que híbrida bajo condiciones de baja, media, medio- alta, alta o muy alta astringencia con la secuencia SEQ ID NO 6 que codifica el polipéptido maduro.
Son asimismo un objeto de la invención, los fragmentos funcionales o las variantes del polipéptido de la invención.
Otro objeto de la invención son las secuencias polinucleotídicas que codifican el polipéptido de la invención o los fragmentos funcionales o las variantes del mismo (en adelante polinucleótido de la invención).
Otro objeto de la invención viene representado por una construcción génica que comprende la secuencia polinucleotídica antes mencionada operativamente unida a una o más secuencias de control que dirigen la expresión de polipéptidos en sistemas de expresión.
Lógicamente, también es objeto de la invención un vector de expresión que comprende el polinucleótido de la invención o dicha construcción génica y una célula huésped que comprende dicho vector de expresión y que es capaz de producir el polipéptido de la invención o los fragmentos funcionales o las variantes de este.
Un objeto adicional de la invención es un método para producir el polipéptido de la invención que comprende:
a) Cultivar la célula huésped antes mencionada en las condiciones necesarias para la producción del polipéptido,
b) Recuperar el polipéptido producido.
También es objeto de la presente invención una composición celulolítica que comprende al menos un polipéptido de acuerdo con la invención o al menos un fragmento funcional o una variante del mismo y, opcionalmente, al menos un elemento que favorezca o facilite la degradación de biomasa (en adelante composición de la invención).
Otro objeto fundamental de la invención es el uso del polipéptido de la invención, de los fragmentos funcionales o las variantes del mismo o de la composición de la invención para la degradación de biomasa.
De este objeto general se derivan los siguientes más particulares.
El primero es un procedimiento para producir azúcares fermentables que comprende incubar biomasa con el polipéptido de la invención, con fragmentos funcionales o variantes del mismo o con la composición de la invención.
El segundo objeto que se deriva del uso antes mencionado es un procedimiento para producir un bioproducto a partir de biomasa que comprende:
a) producir azúcares fermentables incubando biomasa con el polipéptido de la invención, con fragmentos funcionales del mismo o con la composición de la
invención
b) llevar a cabo una transformación química de los azúcares obtenidos en la etapa a) para obtener al menos un bioproducto,
... [Seguir leyendo]
Reivindicaciones:
1. Un polipéptido aislado con actividad celulasa que comprende la secuencia SEQ ID N01 o que posee un grado de homología o identidad de al menos un 80% con la SEQ ID N01 o que está codificado por un polinucleótido que híbrida bajo condiciones de baja, media, media-alta, alta o muy alta astringencia con la secuencia SEQ ID NO 6 que codifica el polipéptido maduro.
2. Un polipéptido de acuerdo con la reivindicación 1 que posee un grado de homología o identidad de al menos un 85%, preferiblemente de al menos un 90%, más preferiblemente de al menos un 95% y aún más preferiblemente de al menos un 99%.
3. Un polipéptido de acuerdo con cualquiera de las reivindicaciones anteriores que es activo a temperaturas de entre 30 y 65°C.
4. Un fragmento funcional o una variante de un polipéptido de acuerdo con cualquiera de las reivindicaciones anteriores.
5. Un fragmento de acuerdo con la reivindicación 4 que comprende al menos los aminoácidos 41 a 378 de la SEQ ID NO 1.
6. Un fragmento de acuerdo con la reivindicación 4 o 5 seleccionado entre SEQ ID NO 3, SEQ ID NO 4 o SEQ ID NO 5.
7. Una secuencia polinucleotídica que codifica un polipéptido de acuerdo con cualquiera de las reivindicaciones 1 a 3 o un fragmento o una variante de acuerdo con cualquiera de las reivindicaciones 4 a 6.
8. Un secuencia polinucleotídica de acuerdo con la reivindicación 7 cuya se secuencia se selecciona de entre SEQ ID NO 6, SEQ ID NO 8, SEQ ID NO 9 o SEQ ID NO 10.
9. Una construcción génica que comprende una secuencia polinucleotídica de acuerdo con la reivindicación 7 u 8 operativamente unida a una o más secuencias de control que dirigen la expresión de polipéptidos en sistemas de expresión.
10. Un vector de expresión que comprende una construcción génica de acuerdo con la reivindicación 9.
11. Una célula huésped que comprende una construcción génica de acuerdo con la reivindicación 9 o un vector de acuerdo con la reivindicación 10 y que expresa el polipéptido de acuerdo con cualquiera de las reivindicaciones 1 a 3 o un fragmento o una variante de acuerdo con cualquiera de las reivindicaciones 4-6.
12. Una célula huésped de acuerdo con la reivindicación 11 donde la célula es un organismo unicelular procariota o eucariota.
13. Una célula huésped de acuerdo con cualquiera de las reivindicaciones 11 o 12 donde la célula procariota se selecciona del grupo de bacterias de los géneros Bacillus, Clostridium, Esherichia, Klebsiella, Pseudomonas, Streptomyces, Thermoanaerobacterium o Zymomonas y la célula eucariota se selecciona del grupo de hongos de las especies Aspergillus awamori, Aspergillus foetidus, Aspergillus fumigatus, Aspergillus japonicus, Aspergillus nidulans, Aspergillus niger, Aspergillus oryzae, Bjerkandera adusta, Ceriporiopsis aneirina, Ceriporiopsis caregiea, Ceriporiopsis rivulosa, Ceriporiopsis sufrufa, Ceriporiopsis subvermispora, Coprinus cinereus, Coriolus hirsutus, Fusarium bactridioides, Fusarium cereales, Fusarium culmorum, Fusarium graminearum, Fusarium graminum, Fusarium heterosporum, Fusarium negundi, Fusarium oxysporum, Fusarium reticulatum, Fusarium roseum, Fusarium sambucinum, Fusarium sarcochroum, Fusarium sporotrichioides, Fusarium sulphureum, Fusarium torulosum, Fusarium trichothecioides, Fusarium venenatum, Humicola lanuginosa, Mucor miehei, Myceliophthora thermophila, Neurospora crassa, Penicillium purpurogenum, Phanerochaete chrysosporium, Phlebia radiata, Pleurotus eryngii, Saccharomyces calsbergensis, Saccharomyces cerevisiae, Saccharomyces diastaticus, Saccharomyces douglasii, Saccharomyces kluyveri, Saccharomyces rtorvensis, Saccharomyces oviformis, Thielavia terrestres, Trametes villosa, Trametes versicolor, Trichoderma harzianum, Trichoderma koningii, Trichoderma longibrachiatum, Trichoderma reseii, Trichoderma viride o Yarrowia lipolytica..
14. Un método para producir un polipéptido con actividad celulolítica que comprende:
a) Cultivar una célula huésped de acuerdo con cualquiera de las reivindicaciones 11 a 13 en las condiciones necesarias para la producción del polipéptido,
b) Recuperar el polipéptido producido.
15. Una composición celulolítica que comprende al menos un polipéptido de acuerdo con cualquiera de las reivindicaciones 1 a 3 y/o al menos un fragmento o una vahante de
acuerdo con cualquiera de las reivindicaciones 4 a 6 y, opcionalmente, al menos un elemento que favorezca o facilite la degradación de biomasa.
16. Una composición de acuerdo con la reivindicación 15 que comprende al menos un elemento adicional que se selecciona de entre una o más endoglucanasas, exoglucanasas, celebiohidrolasas, beta-glucosidasas, glicosil-hidrolasas, hemicelulasas, xylanasas, enzimas ligninolíticas o mezclas de las mismas.
17. Uso del polipéptido de acuerdo con cualquiera de las reivindicaciones 1 a 3, del fragmento o de la variante de acuerdo con cualquiera de las reivindicaciones 4 a 6 o de la composición de acuerdo con cualquiera de las reivindicaciones 15 a 16 para la degradación de biomasa.
18. Uso de acuerdo con la reivindicación 17 donde la degradación de biomasa se lleva a cabo en un procedimiento para la producción de bioproductos.
19. Un procedimiento para producir azúcares fermentables que comprende incubar biomasa con el polipéptido de acuerdo con cualquiera de las reivindicaciones 1 a 3, con el fragmento o una variante de acuerdo con cualquiera de las reivindicaciones 4 a 6 o con la composición de acuerdo con cualquiera de las reivindicaciones 15 a 16.
20. Un procedimiento de acuerdo con la reivindicación 19 que comprende recuperar los azúcares obtenidos después de la incubación con el polipéptido de acuerdo con cualquiera de las reivindicaciones 1 a 3, con el fragmento o una variante de acuerdo con cualquiera de las reivindicaciones 4 a 6 o con la composición de acuerdo con cualquiera de las reivindicaciones 15 a 16.
21. Un procedimiento para producir un bioproducto a partir de biomasa que comprende:
a) producir azúcares fermentables de acuerdo con el procedimiento de la reivindicación 19,
b) llevar a cabo una transformación química de los azúcares obtenidos en la etapa a) para producir al menos un bioproducto,
c) Recuperar el al menos un bioproducto obtenido en la etapa b).
22. Un procedimiento de acuerdo con la reivindicación 21 donde la transformación de los azúcares comprende la fermentación con al menos un microorganismo fermentante.
23. Un procedimiento de acuerdo con la reivindicación 22 donde el al menos un microorganismo fermentante se selecciona entre bacterias como Bacillus thermoglucosidaisus, Clostridium butyricum, Corynebacterium glutamicum, Enterobacter aerogenes, Escherichia coli, Geobacillus themoglucosidasius, Klebsiella oxytoca, Lactobacillus sp. Leunoscoc mesenteroides, Thermoanaerobacter BG1L1, Thermoanaerobacter ethanolicus, Thermoanaerobacter mathranii, Thermoanaerobacter thermosaccharolyticum, Zymobacter palmae, Zymomonas mobilis u hongos como Candida arabinofermentans, Candida boidinii, Candida diddensis, Candida fermentans, Chrysosporium lucknowense, Candida pastoris, Candida shehatae, Candida sonorensis, Candida tropicalis, Hansenula anómala, Hansenula polymorpha, Kluyveromyces fragilis, Kluyveromyces marxianus, Pichia pastoris, Pichia stipitis, Saccharomyces cerevisiae, Saccharomyces bulderi, Saccharomyces barnetti, Saccharomyces exiguus, Saccharomyces diastaticus, Saccharomyces uvarum o Schizosaccharomyces pombe o una combinación de alguno de estos microorganismos.
24. Un procedimiento de acuerdo con cualquiera de las reivindicaciones 21 a 23 donde el bioproducto obtenido se selecciona entre alcoholes como etanol, metanol, butanol, hexanol, octanol, decanol, dodecanol, 1,3-butanediol(1,3-diol), 1-alcohol; ácidos orgánicos como ácido cítrico, ácido acético, ácido itacónico, ácido láctico, ácido glutámico, ácido succínico, beta-cetoácido, beta-cetoalcohol, beta-hidroxiácido; cetonas como la acetona; gases como hidrógeno o dióxido de carbono; hidrocarburos como alcanos, alquenos o alquinos; sustancias nitrogenadas como aminas, amida, nitrocompuestos o nitrilos; haluros; aminoácidos como ácido glutámico; antibióticos como penicilina o tetraciclinas; vitamina como riboflavina, vitamina B12 o betacaroteno; ácidos grasos como ácido dodecanoico, ácidos grasos trans-A2 o ácido palmítico; y otros productos como etileno, glicerol, 1,3- propano-diol, betalactano, cefalosporinas, ácidos grasos trans o furano.
Patentes similares o relacionadas:
Sistema de expresión bacteriano cistrónico doble, del 25 de Marzo de 2020, de LUPIN LIMITED: Un procedimiento para la producción de un anticuerpo o un fragmento del mismo que comprende las etapas de: (i) transformar una célula hospedadora bacteriana […]
Partículas de transducción no replicativas y sistemas indicadores basados en partículas de transducción, del 15 de Enero de 2020, de Geneweave Biosciences Inc: Un sistema de empaquetamiento de células bacterianas para empaquetar una molécula de ácido nucleico indicadora en una partícula de transducción no replicativa (NRTP) para su introducción […]
Método de construcción de biblioteca de péptidos y vectores relacionados, del 1 de Enero de 2020, de Hunan Zonsen Peplib Biotech Co., Ltd: Método integrado de construcción de una biblioteca de péptidos completa que contiene todos los péptidos posibles para el tamaño específico, comprendiendo el método: (i) […]
 Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Transporte de proteínas basado en bacterias, del 14 de Agosto de 2019, de Universität Basel: Un vector que comprende, en la dirección 5' a 3': un promotor; una primera secuencia de ADN que codifica una señal de transporte procedente […]
Sistema de expresión y secreción, del 26 de Junio de 2019, de F. HOFFMANN-LA ROCHE AG: Una molécula de ácido nucleico que codifica un primer polipéptido y un segundo polipéptido, enlazada de forma funcional a una secuencia señal que codifica […]
 Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Síntesis microbiana de aldehídos y alcoholes correspondientes, del 17 de Junio de 2019, de Easel Biotechnologies, LLC: Un método para producir un alcohol, que comprende: cultivar una pluralidad de células microbianas en un medio de fermentación, en donde las […]