PCR multiplex monocromática cuantitativa.
Un método para determinar el número de copias de un primer ácido nucleico diana y un segundo ácido nucleico diana,
que comprende:
a) poner en contacto un primer ácido nucleico diana con un primer conjunto de cebadores y un segundo ácido nucleico diana con un segundo conjunto de cebadores y añadir un único marcador de detección para formar una mezcla de reacción en un sistema homogéneo, en el que el número de copias de la primera secuencia de ácido nucleico diana es mayor que el número de copias de la segunda secuencia de ácido nucleico diana,
b) amplificar por la reacción en cadena de la polimerasa el primer ácido nucleico diana con el primer conjunto de cebadores para formar un primer amplicón que tiene una primera temperatura de fusión (Tm) y amplificar por la reacción en cadena de la polimerasa el segundo ácido nucleico diana con el segundo conjunto de cebadores para formar un segundo amplicón que tiene una segunda Tm, en el que dicha segunda Tm es mayor que dicha primera Tm;
c) durante la reacción en cadena de la polimerasa, determinar la cantidad del marcador de detección a una primera temperatura de adquisición, en el que dicha primera temperatura de adquisición de la señal está por debajo de dicha primera Tm;
d) incrementar la temperatura de la mezcla de reacción hasta una segunda temperatura de adquisición de la señal y determinar la cantidad del marcador de detección, en el que dicha segunda temperatura de adquisición de la señal está por encima de dicha primera Tm y por debajo de dicha segunda Tm;
e) repetir las etapas (b) a (d) al menos una vez; y
f) determinar el número de copias relativo de dicho primer y dicho segundo ácidos nucleicos diana.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2009/069243.
Solicitante: UNIVERSITY OF UTAH RESEARCH FOUNDATION.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 615 ARAPEEN DRIVE, SUITE 310 SALT LAKE CITY, UTAH 84108 ESTADOS UNIDOS DE AMERICA.
Inventor/es: CAWTHON,RICHARD M.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2433718_T3.pdf
Fragmento de la descripción:
PCR multiplex monocromática cuantitativa
Campo técnico
Se describen métodos para determinar el número de copias relativo y absoluto de una primera secuencia de ácido nucleico diana comparado con una segunda secuencia de ácido nucleico diana en una única reacción con un único marcador de detección y kits útiles en la práctica de los métodos.
Antecedentes de la invención La reacción en cadena de la polimerasa cuantitativa en tiempo real (QPCR) determina, para cada pocillo de reacción, el Ct, es decir el número de ciclos fraccionado en el que el incremento de la fluorescencia del pocillo (proporcional a la formación del producto) supera un umbral determinado que es varias desviaciones estándar mayor que la fluorescencia en la línea base (Higuchi, R., Fockler, C., Dollinger, G. y Watson, R. (1993) Kinetic PCR analysis: real-time monitoring of DNA amplification reactions. Biotechnology (NY) , 11, 1026-1030) . La representación Ct frente a log (cantidad de ADN diana introducido) es lineal, lo que permite la cuantificación relativa de desconocidos por comparación con una curva estándar derivada de amplificar, en la misma placa, diluciones seriadas de una muestra de ADN de referencia.
Para muchas aplicaciones de QPCR, el investigador desea normalizar la señal de una secuencia diana (T) respecto a la señal de una secuencia de referencia (R) . Los estudios iniciales midieron T y R en reacciones separadas (monoplex) con un colorante que fluoresce después de la intercalación en cualquier ADN bicatenario, por ejemplo, bromuro de etidio o SYBR Green I, y esta estrategia continúa. Estudios más recientes han medido T y R en el mismo recipiente de reacción, en una QPCR multiplex multicolor, usando colorantes fluorescentes separados con distintos espectros de excitación/emisión para cada una de las secuencias de ADN que se estaban cuantificando (Wittwer, C.T., Hermann, M.G., Gundr y , C.N. y Elenitoba-Johnson, K.S. (2001) Real-time multiplex PCR assays. Methods, 25, 430-442) . La medida de las proporciones T/R por QPCR multiplex disminuye a la mitad el número de reacciones de PCR separadas que deben correrse. Además, como tanto las señales T como R se recogen en cada recipiente de reacción, la variación en la cantidad de una muestra de ADN dado que se pipetea para reacciones replicadas ya no genera variación en las proporciones T/R, como lo hace cuando T y R se miden en pocillos separados en una QPCR monoplex. WO0166799 (DUPONT, 2001-09-13) describe una QPCR multiplex con un colorante intercalante.
La desventaja principal de la QPCR multiplex multicolor es el coste relativamente alto de las sondas fluorescentes y el alto coste de los instrumentos de QPCR especializados que están equipados para leer dos o más colores fluorescentes. En las estrategias tradicionales de PCR multiplex (ya sea la PCR cuantitativa o no) , también resulta a veces excesivamente largo identificar conjuntos de cebadores y concentraciones de cebadores que eviten que la amplificación temprana de un molde con número de copias más alto por una pareja de cebadores inhiba la amplificación tardía de un molde diferente con menor número de copias por una segunda pareja de cebadores.
Resumen de la invención La presente invención proporciona métodos para determinar el número de copias de dos o más secuencias de ácido nucleico diana en una única reacción con un único marcador de detección. También se describen métodos para determinar el número de copias de secuencias teloméricas. Estos datos pueden usarse para asociar la longitud del telómero medida con un riesgo de mortalidad o probabilidad de ocurrencia de enfermedad que se corresponde con una longitud de telómero observada en una población.
En la presente memoria se describen métodos y composiciones para determinar el número de copias de un primer ácido nucleico diana comparado con el número de copias de un segundo ácido nucleico diana por una PCR multiplex monocromática cuantitativa (MMQPCR) en un único pocillo, un sistema homogéneo, con un único marcador de detección.
Las ventajas adicionales de los métodos y composiciones descritos se mostrarán en parte en la descripción siguiente y, en parte, se entenderán a partir de la descripción o pueden aprenderse por la práctica de los métodos y composiciones descritos. Las ventajas de los métodos y composiciones descritos se averiguarán y conseguirán por medio de los elementos y combinaciones particularmente remarcadas out en las reivindicaciones adjuntas. Debe entenderse que tanto la descripción general anterior como la descripción detallada siguiente son sólo ejemplares y explicativas y no restrictivas de la invención según se reivindica.
Descripción breve de las figuras Los dibujos adjuntos, que se incorporan en y constituyen una parte de esta especificación, ilustran varias realizaciones de los métodos y composiciones descritos y junto con la descripción, sirven para explicar los principios de los métodos y composiciones descritos.
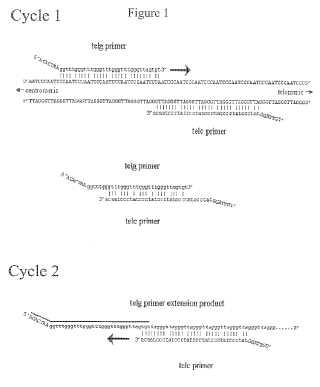
La Figura 1 muestra en Ciclo 1 el cebador telg que hibrida con secuencias teloméricas nativas y ceba la síntesis de ADN. El cebador telc hibrida con secuencias teloméricas nativas pero no puede cebar la síntesis de ADN, debido a su emparejamiento erróneo en 3'. Cuando se hibridan entre sí como se muestra, y en otras configuraciones no mostradas, telg y telc tienen múltiples emparejamientos erróneos, incluyendo en sus bases terminales 3', de manera que se inhibe la formación de dímeros de cebadores. Los extremos 3' de telg y telc pueden alinearse como una superposición de tres pb perfectamente complementaria, no es lo suficientemente estable como para permitir la formación eficaz de dímeros de cebadores. En el Ciclo 2, telc puede hibridar a lo largo de los productos de extensión del cebador telg que se sintetizaron en el Ciclo 1 pero sólo puede cebar la síntesis de ADN cuando hibrida en la configuración mostrada, ya que otras configuraciones producen un emparejamiento erróneo en la base terminal 3' de telc. En el producto de extensión de telg, la barra superior marca la secuencia del cebador telg en sí mismo, y las bases en itálica marcan la secuencia recién sintetizada en el Ciclo 1 de la PCR. Las secuencias en mayúsculas no de molde en los extremos 5' de los cebadores evitan que los extremos 3' del producto telomérico de PCR ceben la síntesis de ADN en la mitad de otras copias del producto telomérico de PCR.
La Figura 2 muestra las curvas de fusión después de 25 ciclos de amplificación (perfil térmico proporcionado en la sección de Materiales y Métodos) de 150 ng de ADN genómico humano sólo con cebadores teloméricos (círculos) , sólo cebadores de albúmina ("x" s) o ambos conjuntos de cebadores (triángulos) . Las curvas de fusión control sin molde están en negro sin símbolos. Después de la incubación final a 880C, las reacciones se enfriaron hasta 720C y la señal se adquirió de 720C a 950C, en pasos de 0, 50C, con un periodo de permanencia de 30 segundos por paso. Existe una diferencia de aproximadamente 110C en las temperaturas de fusión de los amplicones teloméricos y de albúmina.
La Figura 3 muestra PCR Múltiplex Monocromática Cuantitativa (MMQPCR) de 20 ng de cada una de las tres muestras de ADN humano de referencia que previamente se ha mostrado que tienen telómeros largos (círculos) , telómeros de longitud media ("x" s) o telómeros cortos (triángulos) . Las curvas de amplificación control sin molde se muestran con cuadrados negros. Panel superior: representación semi-logarítmica; panel inferior: representación lineal.
La Figura 4 muestra curvas estándar usadas para determinar las proporciones relativas T/S. Se prepararon cinco concentraciones de una muestra de ADN genómico humano estándar que abarcan un intervalo de 81 veces por diluciones seriadas de 3 veces (150 g, 50 ng, 16, 7 ng, 5, 55 ng y 1, 85 ng por pocillo) y se alicuotaron en duplicado en una placa de PCR de 96 pocillos. Las señales fluorescentes tanto de la diana como de referencia se recogieron de cada pocillo de reacción. Los círculos representan los datos del gen de albúmina de copia única, adquiridos a 880C; los triángulos los datos para las repeticiones teloméricas, adquiridos a 740C. El mismo ADN Estándar se usó para formar las reacciones de curva estándar en cada placa en el estudio.
La Figura 5 muestra la correlación de proporciones T/S relativas medidas por PCR Multiplex Monocromática Cuantitativa con albúmina como el gen de copia única, y longitudes medias de Fragmentos de Restricción terminal (TRF) determinadas por análisis de transferencia Southern, en muestras de ADN de sangre completa de 95 individuos. Cada valor T/S es la media de medidas en triplicado; cada longitud TRF media es el... [Seguir leyendo]
Reivindicaciones:
1. Un método para determinar el número de copias de un primer ácido nucleico diana y un segundo ácido nucleico diana, que comprende:
a) poner en contacto un primer ácido nucleico diana con un primer conjunto de cebadores y un segundo ácido nucleico diana con un segundo conjunto de cebadores y añadir un único marcador de detección para formar una mezcla de reacción en un sistema homogéneo, en el que el número de copias de la primera secuencia de ácido nucleico diana es mayor que el número de copias de la segunda secuencia de ácido nucleico diana,
b) amplificar por la reacción en cadena de la polimerasa el primer ácido nucleico diana con el primer conjunto de cebadores para formar un primer amplicón que tiene una primera temperatura de fusión (Tm) y amplificar por la reacción en cadena de la polimerasa el segundo ácido nucleico diana con el segundo conjunto de cebadores para formar un segundo amplicón que tiene una segunda Tm, en el que dicha segunda Tm es mayor que dicha primera Tm;
c) durante la reacción en cadena de la polimerasa, determinar la cantidad del marcador de detección a una primera temperatura de adquisición, en el que dicha primera temperatura de adquisición de la señal está por debajo de dicha primera Tm;
d) incrementar la temperatura de la mezcla de reacción hasta una segunda temperatura de adquisición de la señal y determinar la cantidad del marcador de detección, en el que dicha segunda temperatura de adquisición de la señal está por encima de dicha primera Tm y por debajo de dicha segunda Tm;
e) repetir las etapas (b) a (d) al menos una vez; y
f) determinar el número de copias relativo de dicho primer y dicho segundo ácidos nucleicos diana.
2. El método de la reivindicación 1, en el que la reacción en cadena de la polimerasa comprende al menos tres etapas consecutivas de ciclos, en el que la primera etapa de ciclos de la reacción en cadena de la polimerasa comprende una reacción en cadena de la polimerasa en el que la temperatura de hibridación de la reacción en cadena de la polimerasa es mayor que la temperatura de hibridación de la segunda etapa de ciclos, en el que la segunda etapa de ciclos de la reacción en cadena de la polimerasa comprende una reacción en cadena de la polimerasa en el que la temperatura de hibridación de la reacción en cadena de la polimerasa es menor que la temperatura de hibridación de la primera etapa de ciclos y en el que la tercera etapa de ciclos de la reacción en cadena de la polimerasa comprende una reacción en cadena de la polimerasa en la que la temperatura de hibridación de la reacción en cadena de la polimerasa es menor que la temperatura de hibridación de la primera etapa de ciclos y mayor que la temperatura de hibridación de la segunda etapa de ciclos.
3. El método de la reivindicación 2, en el que
a) sólo se forma el primer amplicón durante la primera etapa de ciclos de la reacción en cadena de la polimerasa,
b) sólo se forma el segundo amplicón durante la segunda etapa de ciclos de la reacción en cadena de la polimerasa, o c) tanto el primer como segundo amplicones se forman durante la tercera etapa de ciclos de la reacción en cadena de la polimerasa.
4. El método de la reivindicación 2, en el que la etapa de amplificación se repite hasta que se determina el marcador de detección a dicha segunda temperatura de adquisición de la señal.
5. El método de la reivindicación 1 ó 2, en el que la cantidad del marcador de detección se detecta a dicha primera y dicha segunda temperaturas de adquisición de la señal durante cada una de dichas etapas de amplificación.
6. El método de la reivindicación 1 ó 2, en el que la diferencia entre la primera Tm y la segunda Tm es al menos 4 grados Celsius.
7. El método de la reivindicación 1 ó 2, en el que
a) al menos uno de los cebadores en el segundo conjunto de cebadores comprende un anclaje GC en el extremo 5' del cebador, o b) al menos uno de los cebadores en dicho primer conjunto de cebadores comprende una secuencia 5' que incluye los nucleótidos A y T.
8. El método de la reivindicación 1 ó 2, en el que los extremos 3' de los cebadores del primer conjunto de cebadores son complementarios entre sí.
9. El método de la reivindicación 8, en el que un cebador del primer conjunto de cebadores es un cebador con emparejamiento erróneo que comprende al menos un nucleótido con emparejamiento erróneo adyacente al extremo 3' del cebador, en el que dicho nucleótido no es complementario al ácido nucleico diana, pero es complementario al nucleótido 3' terminal el otro cebador en el primer conjunto de cebadores.
10. El método de la reivindicación 9, en el que el producto de la extensión del cebador con emparejamiento erróneo del primer conjunto de cebadores es capaz de hibridar con el otro cebador en el primer conjunto de cebadores.
11. El método de la reivindicación 1 ó 2, en el que uno de los cebadores en el primer conjunto de cebadores está bloqueado para cebar el primer ácido nucleico diana.
12. El método de la reivindicación 11, en el que el cebador bloqueado para cebar el primer ácido nucleico diana 10 comprende una base con emparejamiento erróneo en su extremo 3'.
13. El método de la reivindicación 1 ó 2, en el que el marcador de detección es un colorante intercalante.
14. El método de la reivindicación 1 ó 2, en el que el número de copias de un primer y un segundo ácidos nucleicos diana mide la cantidad relativa del primer ácido nucleico comparado con el segundo ácido nucleico.
15. El método de la reivindicación 1 ó 2, en el que el primer ácido nucleico diana
a) comprende una secuencia de repetición en tándem, o b) se obtiene de una muestra.
16. El método de la reivindicación 15, en el que el número de copias del primer ácido nucleico diana determina el número de secuencias de repetición en tándem presente en la muestra.
17. El método de la reivindicación 1, en el que la cantidad de marcador de detección determinada durante la primera 20 y dicha segunda temperaturas de adquisición de la señal se comparan con un control.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]