Mutaciones del gen de PIK3CA en cánceres humanos.
Método de evaluación del cáncer en una muestra corporal de un ser humano que se sospecha que tienecáncer,
que comprende las etapas de:
determinar una mutación no sinónima, intragénica en una secuencia que codifica para PIK3CA en lamuestra corporal, en el que una secuencia que codifica para PIK3CA de tipo natural consiste en lasecuencia nt1-3204 de SEQ ID NO: 1; y la mutación provoca un cambio en un residuo de aminoácidoseleccionado del grupo que consiste en R38C, R38H, R88Q, P104R, G106V, R108P, delK111, G118D,G122D, P124T, N345K, D3 50H, C378R, C420R, E453Q, P539R, E542K, E542G, E542V, E545K, E545G,E545D, Q546K, Q546P, Q661K, H701P, C901F, F909L, S1008P, T1025A, T1025N, M1043I, H1047Y,H1047R, H1047L, G1049S; e
identificar que el ser humano probablemente tiene cáncer si se determina la mutación no sinónima,intragénica en la secuencia que codifica para PIK3CA en la muestra corporal.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2005/005193.
Solicitante: THE JOHNS HOPKINS UNIVERSITY.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 3400 NORTH CHARLES STREET BALTIMORE, MD 21218 ESTADOS UNIDOS DE AMERICA.
Inventor/es: VOGELSTEIN, BERT, KINZLER, KENNETH, W., SAMUELS,YARDENA, VELCULESCU,VICTOR.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2437590_T3.pdf
Fragmento de la descripción:
Mutaciones del gen de PIK3CA en cánceres humanos Esta solicitud se realizó usando fondos proporcionados por el gobierno de los Estados Unidos con las subvenciones n.os NIH-CA 62924 y NIH-CA 43460. El gobierno de los Estados Unidos conserva por tanto ciertos derechos en la invención.
CAMPO DE LA INVENCIÓN
La invención se refiere a los campos de pruebas de diagnóstico y métodos terapéuticos para el cáncer.
ANTECEDENTES DE LA INVENCIÓN
Las PI3K son lípido cinasas que funcionan como transductores de señales aguas abajo de receptores de la superficie celular y median rutas importantes para el crecimiento, la proliferación, la adhesión, la supervivencia y la motilidad celulares (1, 2) . Aunque se ha observado aumento de la actividad de PI3K en muchos tumores colorrectales y otros (3, 4) , no se ha identificado ninguna mutación intragénica de PI3K.
Se ha notificado anteriormente que miembros de la ruta de PIK3 están alterados en cánceres, por ejemplo, el gen supresor de tumores PTEN (15, 16) , cuya función es revertir la fosforilación mediada por PI3K (17, 18) . Se ha notificado la reduplicación o amplificación de las regiones cromosómicas que contienen PIK3CA y AKT2 en algunos cánceres humanos (2, 19, 20) , pero los genes que son las dianas de tales acontecimientos genéticos a gran escala no se han definido ni pueden definirse fácilmente.
Zhang et al., (Cancer Research, 63, 4225-4231, 2003) dan a conocer la sobreexpresión de PIK3CA en cáncer de ovario.
Ma et al., (Oncogene, 19, 2739-2744, 2000) dan a conocer que PIK3CA es un oncogén en cáncer de cuello uterino.
Shayesteh et al., (Nature Genetics, 21, 99-102, 1999) dan a conocer que PIK3CA es un oncogén en cáncer de cuello uterino.
Hill et al., (J. Biol. Chem., 275, 3741-3744, 2000) dan a conocer el uso de anticuerpos anti-p110a para bloquear las respuestas móviles estimuladas por EGF en células de cáncer de mama.
Dao et al., (Proceedings of the Annual Meeting of the American Association for Cancer Research, 43, 602-2002) dan a conocer que el bloqueo de la expresión de p110a con moléculas antisentido para PIK3CA anula el fenotipo de cáncer.
Philp et al, (Cancer Research, 61, 7426-7429, 2001) dan a conocer la presencia de una mutación somática en la subunidad reguladora P85a de PIK3R1 en tumores de ovario y de colon humanos.
BREVE SUMARIO DE LA INVENCIÓN
En una primera realización, se proporciona un método para evaluar el cáncer en una muestra corporal de un ser humano que se sospecha que tiene un cáncer, que comprende las etapas de determinar una mutación no sinónima, intragénica en una secuencia que codifica para PIK3CA en la muestra corporal, en el que una secuencia que codifica para PIK3CA de tipo natural comprende la secuencia mostrada en SEQ ID NO: 2; y la mutación provoca un cambio en un residuo de aminoácido seleccionado del grupo que consiste en R38C, R38H, R88Q, P104R, G106V, R108P, delK111, G118D, G122D, P124T, N345K, D350H, C378R, C420R, E453Q, P539R, E542K, E542G, E542V, E545K, E545G, E545D, Q546K, Q546P, Q661K, H701P, C901F, F909L, S1008P, T1025A, T1025N, M1043I, H1047Y, H1047R, H1047L, G1049S; e identificar que el ser humano probablemente tiene cáncer si se determina la mutación no sinónima, intragénica en la secuencia que codifica para PIK3CA en la muestra corporal.
BREVE DESCRIPCIÓN DE LOS DIBUJOS
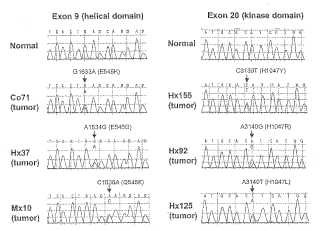
Figura 1. Detección de mutaciones en PIK3CA. Ejemplos representativos de mutaciones en los exones 9 y 20. En cada caso, se obtuvo el cromatograma de secuencia superior de tejido normal y los tres cromatogramas de secuencia inferiores de los tumores indicados. Las flechas indican la ubicación de las mutaciones de cambio de sentido. Las alteraciones de nucleótidos y aminoácidos se indican por encima de la flecha.
Figura 2. Distribución de mutaciones en PIK3CA. Las flechas indican la ubicación de mutaciones de cambio de sentido, y los recuadros representan dominios funcionales (p85BD, dominio de unión a p85; RBD, dominio de unión a Ras; dominio C2; dominio helicoidal; dominio cinasa) . El porcentaje de mutaciones detectado dentro de cada región en cánceres se indica a continuación.
Figuras 3A-3C. Aumento de la actividad lípido cinasa de p110a mutante. Se transfectaron células NIH3T3 con vector vacío o con constructos de vector que contenía o bien p110a de tipo natural o bien p110a mutante (H1047R) tal como se indica por encima de los carriles. Se realizaron inmunoprecipitaciones con o bien IgG control o bien anticuerpos policlonales anti-p85. (Figura 3A) La mitad de los inmunoprecipitados se sometieron a un ensayo de PI3cinasa usando fosfatidilinositol como sustrato. “PI3P” indica la posición de PI-3-fosfato determinada con marcadores de fosfatidilo patrón y “Ori” indica el origen. (Figura 3B) La otra mitad de los inmunoprecipitados se analizó mediante inmunotransferencia de tipo Western con anticuerpo anti-p110a. (Figura 3C) Los lisados celulares de células transfectadas contenían cantidades similares de proteína total tal como se determina mediante inmunotransferencia de tipo Western usando un anticuerpo anti-tubulina a. Se observaron resultados idénticos a los mostrados en esta figura en tres experimentos de transfección independientes.
DESCRIPCIÓN DETALLADA DE LA INVENCIÓN
El agrupamiento de mutaciones dentro de PIK3CA lo convierten en un excelente marcador para la detección temprana o para el seguimiento de la progresión de la enfermedad. Las pruebas centradas en las regiones agrupadas producirán la mayor parte de los alelos mutantes.
La secuencia que codifica para PIK3CA humana se notifica en la bibliografía y se muestra en SEQ ID NO: 1. Esta es la secuencia de un individuo particular en la población de seres humanos. Los seres humanos varían entre sí en sus secuencias génicas. Estas variaciones son muy mínimas, produciéndose algunas veces a una frecuencia de aproximadamente 1 a 10 nucleótidos por gen. Existen formas diferentes de cualquier gen particular dentro de la población humana. Estas formas diferentes se denominan variantes alélicas. Las variantes alélicas a menudo no cambian la secuencia de aminoácidos de la proteína codificada; tales variantes se denominan sinónimas. Incluso si cambian el aminoácido codificado (no sinónimas) , la función de la proteína no se ve afectada normalmente. Tales cambios son evolutiva o funcionalmente neutros. Cuando se hace referencia a PIK3CA humana en la presente solicitud, se pretende que todas las variantes alélicas se abarquen por el término. La secuencia de SEQ ID NO: 1 se proporciona meramente como ejemplo representativo de una secuencia humana de tipo natural. La invención no se limita a esta forma alélica individual de PIK3CA. Para los fines de determinar una mutación, pueden compararse las secuencias de PIK3CA determinadas en una muestra de prueba con una secuencia determinada en un tejido diferente del ser humano. Una diferencia en la secuencia en los dos tejidos indica una mutación somática. Alternativamente, puede compararse la secuencia determinada en un gen de PIK3CA en una muestra de prueba con la secuencia de SEQ ID NO: 1. Puede identificarse una diferencia entre la secuencia de muestra de prueba y SEQ ID NO: 1 como una mutación. Pueden someterse a prueba tejidos que se sospecha que son cancerosos, como también muestras corporales que puede esperarse que contengan células descamadas de tumores o células de cáncer. Las muestras corporales adecuadas para las pruebas incluyen sangre, suero, plasma, esputo, orina, heces, aspirado del pezón, saliva y líquido cefalorraquídeo.
Las mutaciones en PIK3CA se agrupan en los exones 9 (SEQ ID NO: 4) y 20 (SEQ ID NO: 5) . Se producen otras mutaciones, pero estos dos exones parecen ser los puntos activos para las mutaciones. Muchas mutaciones se producen en el dominio helicoidal de PIK3CA (nt 1567-2124 de SEQ ID NO: 2) y en su dominio cinasa (nt 2095-3096 de SEQ ID NO: 2) . Menos se producen en el dominio P85BD de PIK3CA (nt 103-335 de SEQ ID NO: 2) . Se han encontrado mutaciones en los exones 1, 2, 4, 5, 7, 9, 13, 18 y 20. Puede someterse a prueba cualquier combinación de estos exones, opcionalmente junto con pruebas en otros exones. Las pruebas para detectar mutaciones pueden realizarse a lo largo de la secuencia codificante completa o pueden centrarse en las zonas en la que se ha encontrado que se agrupan las mutaciones. Se producen zonas activas particulares de mutaciones en las posiciones de nucleótido 1624, 1633, 1636 y 3140 de la secuencia que codifica para PIK3CA.
Se han encontrado mutaciones de PIK3CA en una variedad de diferentes tipos de tumores. Por tanto, puede someterse a prueba cualquiera de una variedad de tumores para detectar... [Seguir leyendo]
Reivindicaciones:
1. Método de evaluación del cáncer en una muestra corporal de un ser humano que se sospecha que tiene cáncer, que comprende las etapas de:
determinar una mutación no sinónima, intragénica en una secuencia que codifica para PIK3CA en la muestra corporal, en el que una secuencia que codifica para PIK3CA de tipo natural consiste en la secuencia nt1-3204 de SEQ ID NO: 1; y la mutación provoca un cambio en un residuo de aminoácido seleccionado del grupo que consiste en R38C, R38H, R88Q, P104R, G106V, R108P, delK111, G118D, G122D, P124T, N345K, D350H, C378R, C420R, E453Q, P539R, E542K, E542G, E542V, E545K, E545G, E545D, Q546K, Q546P, Q661K, H701P, C901F, F909L, S1008P, T1025A, T1025N, M1043I, H1047Y, H1047R, H1047L, G1049S; e identificar que el ser humano probablemente tiene cáncer si se determina la mutación no sinónima, intragénica en la secuencia que codifica para PIK3CA en la muestra corporal.
2. Método según la reivindicación 1, en el que la muestra corporal es un primer tejido que se sospecha que es neoplásico, y el método comprende además las etapas de:
someter a prueba un segundo tejido que no se sospecha que es neoplásico para detectar la presencia de la mutación no sinónima, intragénica, en el que los tejídos primero y segundo se aíslan del ser humano; e identificar la mutación no sinónima, intragénica como somática si dicha mutación está ausente en el segundo tejido.
3. Método según la reivindicación 1, en el que la mutación no sinónima, intragénica se selecciona del grupo que consiste en CH2T, G113A, G263A, C311G, G317T, G323C, del332-334, G353A, G365A, C370A, T1035A, G1048C, T1132C, T1258C, G1357C, G1616G, G1624A, A1625G, A1625T, G1633A, A1634G, G1635T, C1636A, A1637C, C1981A, A2102C, G2702T, T2725C, T3022C, A3073G, C3074A, G3129T, C3139T, A3140G, A3140T, G3145A.
4. Método según la reivindicación 1, en el que la mutación no sinónima, intragénica es en el dominio helicoidal de PIK3CA (nt 1567-2124 de SEQ ID NO: 2) .
5. Método según la reivindicación 1, en el que la mutación no sinónima, intragénica en el dominio cinasa de PIK3CA (nt 2095-3096 de SEQ ID NO: 2) .
6. Método según la reivindicación 1, en el que la mutación no sinónima, intragénica es en el dominio P85BD de PIK3CA (n.
10. 335 de SEQ ID NO: 2) .
7. Método según la reivindicación 1, en el que la muestra corporal es tejido colorrectal.
8. Método según la reivindicación 1, en el que la muestra corporal es tejido cerebral.
9. Método según la reivindicación 1, en el que la muestra corporal es tejido gástrico.
10. Método según la reivindicación 1, en el que la muestra corporal es tejido de mama.
11. Método según la reivindicación 1, en el que la muestra corporal es tejido pulmonar.
12. Método según la reivindicación 1, en el que la muestra corporal es sangre, suero o plasma.
13. Método según la reivindicación 1, en el que la muestra corporal es esputo.
14. Método según la reivindicación 1, en el que la muestra corporal es saliva.
15. Método según la reivindicación 1, en el que la muestra corporal es orina.
16. Método según la reivindicación 1, en el que la muestra corporal son heces.
17. Método según la reivindicación 1, en el que la muestra corporal es aspirado del pezón.
18. Método según la reivindicación 1, en el que la mutación no sinónima, intragénica es G1624A.
19. Método según la reivindicación 1, en el que la mutación no sinónima, intragénica es G1633A.
20. Método según la reivindicación 1, en el que la mutación no sinónima, intragénica es C1636A.
21. Método según la reivindicación 1, en el que la mutación no sinónima, intragénica es A3140G.
22. Método según la reivindicación 1, en el que la muestra corporal se somete a prueba para detectar mutaciones en las posiciones de nucleótido 1624, 1633, 1636 y 3140 de la secuencia que codifica para PIK3CA.
23. Método según la reivindicación 1, en el que la muestra corporal se somete a prueba para detectar las 10 mutaciones G1624A, G1633A, C1636A y A3140G.
24. Método según la reivindicación 23, en el que la muestra corporal se somete a prueba adicionalmente para detectar las mutaciones G113A, T1258C, G3129T y C3139T.
25. Método según la reivindicación 23, en el que la muestra corporal se somete a prueba adicionalmente para detectar la mutación G2702T.
26. Método según la reivindicación 1, en el que la mutación no sinónima, intragénica es una mutación por
deleción. 20
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]