Motivos estructurales de polipéptidos asociados con la actividad de señalización celular.
Un polipéptido de aminoacil-ARNt sintetasa (AARS) aislado que consiste en 60-300 residuos de aminoácido,
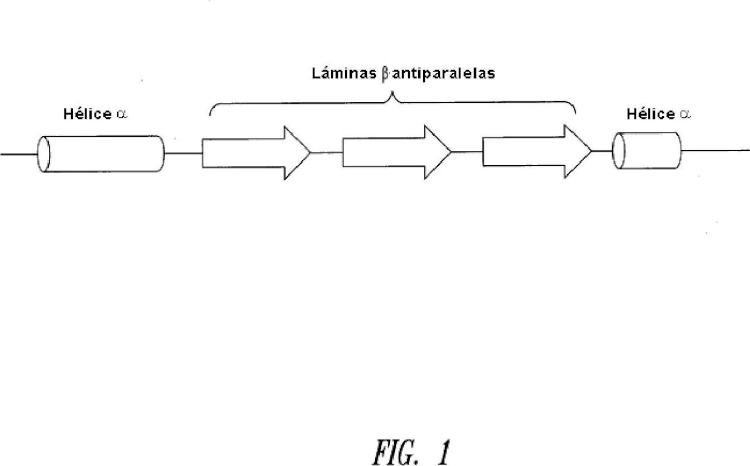
en donde el polipéptido de AARS comprende tres hebras ß antiparalelas flanqueadas en cada extremo por una hélice α y muestra una actividad de señalización celular, en donde el polipéptido comprende los residuos 325-410 del SEQ ID NO: 6 o un fragmento activo o variante que muestra una identidad de al menos 90% con los residuos 325-410 del SEQ ID NO: 6, y en donde el polipéptido tiene actividad quimioquina.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2010/025642.
Solicitante: Atyr Pharma, Inc.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 3565 General Atomics Court, Suite 103 San Diego, CA 92121 ESTADOS UNIDOS DE AMERICA.
Inventor/es: GREENE,LESLIE ANN.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- A61K38/00 NECESIDADES CORRIENTES DE LA VIDA. › A61 CIENCIAS MEDICAS O VETERINARIAS; HIGIENE. › A61K PREPARACIONES DE USO MEDICO, DENTAL O PARA EL ASEO (dispositivos o métodos especialmente concebidos para conferir a los productos farmacéuticos una forma física o de administración particular A61J 3/00; aspectos químicos o utilización de substancias químicas para, la desodorización del aire, la desinfección o la esterilización, vendas, apósitos, almohadillas absorbentes o de los artículos para su realización A61L; composiciones a base de jabón C11D). › Preparaciones medicinales que contienen péptidos (péptidos que contienen ciclos beta-lactama A61K 31/00; dipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina 2,5-dionas, A61K 31/00; péptidos basados en la ergolina A61K 31/48; que contienen compuestos macromoleculares que tienen unidades aminoácido repartidas estadísticamente A61K 31/74; preparaciones medicinales que contienen antígenos o anticuerpos A61K 39/00; preparaciones medicinales caracterizadas por los ingredientes no activos, p. ej. péptidos como soportes de fármacos, A61K 47/00).
- C07K1/113 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07K PEPTIDOS (péptidos que contienen β -anillos lactamas C07D; ipéptidos cíclicos que no tienen en su molécula ningún otro enlace peptídico más que los que forman su ciclo, p. ej. piperazina diones-2,5, C07D; alcaloides del cornezuelo del centeno de tipo péptido cíclico C07D 519/02; proteínas monocelulares, enzimas C12N; procedimientos de obtención de péptidos por ingeniería genética C12N 15/00). › C07K 1/00 Procedimientos generales de preparación de péptidos. › sin cambio de la estructura primaria.
- C12N15/63 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Introducción de material genético extraño utilizando vectores; Vectores; Utilización de huéspedes para ello; Regulación de la expresión.
- G01N33/15 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › preparaciones medicinales.
- G01N33/68 G01N 33/00 […] › en los que intervienen proteínas, péptidos o aminoácidos.
PDF original: ES-2552773_T3.pdf




Patentes similares o relacionadas:
Métodos y composiciones para el diagnóstico y pronóstico de lesión renal e insuficiencia renal, del 29 de Julio de 2020, de Astute Medical, Inc: Un método para evaluar el estado renal en un sujeto, que comprende: realizar una pluralidad de ensayos configurados para detectar una […]
Neuregulina para tratar la insuficiencia cardíaca, del 29 de Julio de 2020, de Zensun (Shanghai) Science & Technology, Co., Ltd: Neuregulina para usar en un método para tratar la insuficiencia cardíaca crónica en un paciente, donde el paciente tiene un nivel plasmático de NT-proBNP […]
Inmunomoduladores, del 29 de Julio de 2020, de BRISTOL-MYERS SQUIBB COMPANY: Un compuesto de la fórmula (I) **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo, en donde: A se selecciona de **(Ver fórmula)** en donde: […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Método para llevar a cabo el seguimiento de la enfermedad de Gaucher, del 15 de Julio de 2020, de Centogene GmbH: Un método para determinar la evolución de la enfermedad de Gaucher en un sujeto, que comprende la etapa de determinar en varios puntos en el […]
Procedimiento para evaluación de la función hepática y el flujo sanguíneo portal, del 15 de Julio de 2020, de The Regents of the University of Colorado, a body corporate: Procedimiento in vitro para la estimación del flujo sanguíneo portal en un individuo a partir de una única muestra de sangre o suero, comprendiendo el procedimiento: […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos de monitorización terapéutica de profármacos de ácido fenilacético, del 24 de Junio de 2020, de Immedica Pharma AB: Glicerilo tri-[4-fenilbutirato] (HPN-100) para su uso en un método para tratar un trastorno del ciclo de la urea en un sujeto que tiene discapacidad […]