Moléculas de ADN y métodos.
Una molécula de ADN que comprende: un intrón de Grupo II modificado que no expresa la transcriptasa inversa codificada por el intrón,
pero que contiene un gen de marcador seleccionable modificado en la orientación inversa respecto del intrón de Grupo II modificado, donde el gen de marcador seleccionable comprende una región codificadora de marcador seleccionable y un promotor ligado operativamente a dicha región, siendo dicho promotor capaz de generar la expresión del marcador seleccionable codificado por una única copia del gen de marcador seleccionable en una cantidad suficiente para que el marcador seleccionable altere el fenotipo de una célula bacteriana de la clase Clostridia, de manera tal que pueda ser distinguida de la célula bacteriana de la clase Clostridia carente del gen de marcador seleccionable; y un promotor para la transcripción del intrón de Grupo II modificado, estando dicho promotor ligado operativamente a dicho intrón de Grupo II modificado; donde el gen de marcador seleccionable modificado contiene un intrón de Grupo I situado en orientación hacia adelante respecto del intrón de Grupo II modificado, a fin de alterar la expresión del marcador seleccionable; donde la molécula de ADN permite la remoción del intrón de Grupo I del transcripto de ARN del intrón de Grupo II modificado, a fin de dejar una región codificadora del marcador seleccionable, y permite la inserción de dicho transcripto de ARN o una de sus copias de ADN en un sitio de una molécula de ADN en una célula bacteriana de la clase Clostridia; y donde el marcador seleccionable confiere resistencia a eritromicina a la célula bacteriana de la clase Clostridia.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/GB2007/002308.
Solicitante: MORVUS TECHNOLOGY LTD.
Nacionalidad solicitante: Reino Unido.
Dirección: Llanvetherine Court Abergavenny NP7 8NL REINO UNIDO.
Inventor/es: MINTON, NIGEL, PETER, HEAP,JOHN TIMOTHY.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07H21/04 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07H AZUCARES; SUS DERIVADOS; NUCLEOSIDOS; NUCLEOTIDOS; ACIDOS NUCLEICOS (derivados de ácidos aldónicos o sacáricos C07C, C07D; ácidos aldónicos, ácidos sacáricos C07C 59/105, C07C 59/285; cianohidrinas C07C 255/16; glicales C07D; compuestos de constitución indeterminada C07G; polisacáridos, sus derivados C08B; ADN o ARN concerniente a la ingeniería genética, vectores, p. ej. plásmidos o su aislamiento, preparación o purificación C12N 15/00; industria del azúcar C13). › C07H 21/00 Compuestos que contienen al menos dos unidades mononucleótido que tienen cada una grupos fosfato o polifosfato distintos unidos a los radicales sacárido de los grupos nucleósido, p. ej. ácidos nucleicos. › con desoxirribosilo como radical sacárido.
- C12N15/74 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Vectores o sistemas de expresión especialmente adaptados a huéspedes procariotas distintos a E. coli, p. ej. Lactobacillus, Micromonospora.
- C12P19/34 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
PDF original: ES-2546648_T3.pdf
Fragmento de la descripción:
Moléculas de ADN y métodos
La presente descripción se refiere a moléculas de ADN y métodos que usan las moléculas para Introducir mutaciones en el ADN de una célula bacteriana grampositiva, en particular una célula de la clase Clostridia.
La clase Clostridia incluye los órdenes Clostridiales, Halanaerobiales y Thermoanaerobacteriales. El orden Clostridiales incluye la familia Clostridiaceae, que incluye el género Clostridium.
El género Clostridium es uno de los géneros bacterianos más grandes. Está compuesto por formadores de esporas grampositivos anaerobios obligados. Ciertos miembros se pueden emplear en escala industrial para la producción de combustibles químicos, p.ej., Clostridium thermocellum y Clostridium acetobutylicum. También se ha demostrado que esta última especie de Clostridia, junto con otros representantes benignos, tiene potencial como vehículo de entrega de agentes terapéuticos dirigidos contra el cáncer. No obstante, el género ha logrado mayor notoriedad como consecuencia de los miembros que causan enfermedades en humanos y animales domésticos, p.ej., Clostridium difficile, Clostridium botulinum y Clostridium perfringens.
A pesar de la destacada importancia comercial y médica del género, el progreso hacia su explotación efectiva o sobre el desarrollo de enfoques racionales contra las enfermedades que causan se ha visto gravemente obstaculizado por la falta de un conocimiento básico de la biología de los organismos a nivel molecular. En gran medida, esto es consecuencia de la ausencia de herramientas genéticas efectivas.
En años recientes, se han determinado las secuencias completas del genoma de al menos una cepa representativa de las especies principales, incluso de C. acetobutylicum, C. difñcile, C. botulinum y C. perfringens. En otras especies bacterianas, este conocimiento puede servir como trampolín para un manejo más efectivo de las enfermedades o para la generación de cepas con propiedades de proceso mejoradas. Una herramienta fundamental para estos proyectos es la capacidad para integrar racionalmente el ADN al genoma. Dicha tecnología se puede emplear: (i) para generar mutantes específicos como medio para asignar funciones a genes individuales y conjuntos de genes, como paso inicial esencial hacia la comprensión de la fisiología y la patogenia; (ii) para inactivar genes regulatorios o estructurales por inserción, como medio para mejorar la producción de mercaderías de interés, y (iii) para introducir en forma estable información genética codificadora de factores no previstos. Sin embargo, en la actualidad no existen vectores de integración efectivos para estudios de mutación en ningún Clostridium sp. y la capacidad para inactivar genes por inserción en el género continúa siendo lamentablemente inadecuada.
Los intentos previos tendientes a preparar mutantes de Clostridium sp. dependieron de la recombinación homologa entre un vector de integración y el cromosoma del huésped. En la cepa 13 de C. perfringens, NCIMB 852 de C. beijerinckii, ATCC 824 de C. acetobutylicum y CD37 de C. difñcile, se ha demostrado que los plásmidos con origen de replicación negativo portadores de regiones del cromosoma del huésped se integran al genoma por recombinación homologa (Shimizu et al. (1994) J. Bacteriol. 176: 1616-23; Wilkinson y Young (1994) Microbiol. 14: 89-95; Green et al. (1996) Microbiol. 142: 279-286; Liyanage et al. (21) Appl. Environ. Microbiol. 67: 24-21). En el caso de C. beijerinckii y C. difficile, se movilizaron vectores de donantes de E. coli. En C. perfringens y C. acetobutylicum, se introdujeron plásmidos por transformación. En C. beijerinckii, surgieron integrantes con frecuencias de 1'6 a 1'7 por receptor, lo cual representa unos dos órdenes de magnitud inferiores a la frecuencia de transferencia observada (1'4 a 1'5) con plásmidos competentes para la replicación (Wilkinson y Young, 1994, supra). En el caso de C. difñcile, no se informó indicación de las frecuencias obtenidas (Liyanage et al., 21, supra). En C. acetobutylicum, surgieron integrantes con una frecuencia de ,8 a ,9 "colonias" por mg de ADN (Green ef al., 1996, supra). En los integrantes mencionados, las secuencias de plásmido en el sitio objetivo estaban flanqueadas por dos copias directamente repetidas del segmento de ADN director de la integración. Como consecuencia, eran inestables en la segregación, p.ej., con pérdidas por 3 generaciones de entre 1,8 a 3, X 1'3 para C. acetobutylicum (Green et al., 1996, supra) y entre ,37 a 1,3 X 1'3 para C. beijerinckii (Wilkinson y Young, 1994, supra).
Por lo tanto, se prefieren los integrantes obtenidos por intercambio alélico. En consecuencia, se buscaron y obtuvieron mutantes con cruzamiento doble en C. perfringens (Awad et al. (1995) Mol. Microbiol. 15: 191-22; Bannam et al. (1995) Mol. Microbiol. 16: 535-551. Sin embargo, el intercambio alélico solo fue posible mediante la inclusión de regiones de homología relativamente largas (3, kb) en cada extremo del gen de resistencia a antibióticos empleado para inactivar el gen objetivo. Además, incluso con esta precaución, el aislamiento de los mutantes demostró ser muy variable (es decir, solo se obtuvieron mutantes pie en 2 de 1 experimentos independientes) y los aislamientos de muchos mutantes pueden tardar hasta 6 meses, mientras que otros nunca se aíslan. Se pudieron detectar eventos de integración infrecuentes en C. perfringens como consecuencia de la alta frecuencia con la cual se puede transformar ADN en este microorganismo. Los intentos tendientes a generar mutantes de cruzamiento dobles en otras especies de Clostridia fueron infructuosos.
Hasta la fecha, la generación de mutantes en cierto rango de especies de Clostridia distintas de C. perfringens, mediante el uso de recombinación homologa clásica, ha demostrado ser difícil. Así, solo se han realizado cinco mutaciones en C. acetobutylicum. Cuatro (butK, CAC375; pta, CAC1742; aad, CACP162 y soIR, CACP61) fueron
preparados mediante integración por cruzamiento simple de un plásmido deficiente en replicación (Green et al., 1996, supra] Green y Bennett (1996) Appl. Biochem. Biotechnol. 213, 57-58; Harris et al. (22) J. Bactehol. 184, 3586-3597) mientras que un quinto en spoOA (CAC271) fue aislado mediante una estrategia que intentó sin éxito la generación de un mutante por intercambio recíproco que usó un plásmido de replicación defectuosa (Nair ef al. (1999) J. Bacteriol. 181, 319-33). De modo similar, solo se ha informado la generación de tres mutantes dirigidas en C. difficile. Un mutante (gldA, CD274) fue generado mediante un plásmido de replicación deficiente (Liyanage ef al., 21, supra) si bien este evento pareció ser letal y las células mutantes no pudieron propagarse. Los otros dos genes inactivados (rgaR, CD3255 y rgbR, CD189) surgieron después de la introducción de un plásmido de replicación defectuosa portador de fragmentos internos de los dos genes estructurales (OConnor et al. (26) Mol. Microbiol. 61, 1335-1351). Al parecer, estos últimos plásmidos fueron introducidos con "cierta dificultad" y si bien se aislaron integrantes, no se reportaron frecuencias de aislamiento. De hecho, es difícil hacer una evaluación de las eficacias de los procedimientos de mutagénesis usados previamente en ambos microorganismos, dado que generalmente no se presentan indicaciones de la frecuencia de generación de tales mutantes. En el caso de C. acetobutylicum (Thomas et al., (25) Metabolic engineering of soventogenic clostridia. En: Dürre, P. Handbook on Clostridia, CRC Press, pp 813-83) se reconoce que hay "menos de un transformante por mg de ADN de plásmido". Además, dado que la mayoría de estos mutantes se preparan por inserción de cruzamiento simple, son inestables debido a la escisión del plásmido. Por ejemplo, la prueba Southern blotting de mutante rgaR de C. difficile reveló la presencia de "remoción por bucle", con plásmidos de replicación independiente en algunas células de la población (OConnor et al., 26, supra).
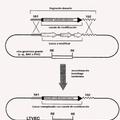
Se ha observado un aumento en la cantidad de diseños de tecnología que sacan provecho de los sistemas que incluyen elementos genéticos móviles para lograr una modificación más efectiva de los genomas bacterianos. El intrón de Grupo II LI.LtrB de Lactococcus lactis es un elemento que media su propia movilidad a través de la acción de una transcriptasa inversa codificada por el intrón (LtrA) y el bucle de ARN removido. Además, puede ser redirigido a virtualmente cualquier secuencia de ADN deseada a través de la modificación de del ARN del intrón (Guo et al. (2) Science 289: 452-457; Mohr et al. (2) Genes Dev. 14: 559-573). En consecuencia, mediante la mutación adecuada de bases individuales en la región de 15 bp del intrón involucrado en el objetivo, Karberg et al.... [Seguir leyendo]
Reivindicaciones:
1. Una molécula de ADN que comprende: un intrón de Grupo II modificado que no expresa la transcriptasa inversa codificada por el intrón, pero que contiene un gen de marcador seleccionable modificado en la orientación inversa respecto del intrón de Grupo II modificado, donde el gen de marcador seleccionable comprende una región codificadora de marcador seleccionable y un promotor ligado operativamente a dicha región, siendo dicho promotor capaz de generar la expresión del marcador seleccionable codificado por una única copia del gen de marcador seleccionable en una cantidad suficiente para que el marcador seleccionable altere el fenotipo de una célula bacteriana de la clase Clostridia, de manera tal que pueda ser distinguida de la célula bacteriana de la clase Clostridia carente del gen de marcador seleccionable; y un promotor para la transcripción del intrón de Grupo II modificado, estando dicho promotor ligado operativamente a dicho intrón de Grupo II modificado; donde el gen de marcador seleccionable modificado contiene un intrón de Grupo I situado en orientación hacia adelante respecto del intrón de Grupo II modificado, a fin de alterar la expresión del marcador seleccionable; donde la molécula de ADN permite la remoción del intrón de Grupo I del transcripto de ARN del intrón de Grupo II modificado, a fin de dejar una región codificadora del marcador seleccionable, y permite la inserción de dicho transcripto de ARN o una de sus copias de ADN en un sitio de una molécula de ADN en una célula bacteriana de la clase Clostridia', y donde el marcador seleccionable confiere resistencia a eritromicina a la célula bacteriana de la clase Clostridia.
2. La molécula de ADN de la Reivindicación 1, donde el intrón de Grupo II modificado está flanqueado por exones, donde los exones permiten el ensamblado de un transcripto de ARN del intrón de Grupo II.
3. La molécula de ADN de una cualquiera de las Reivindicaciones 1 o 2, donde el intrón de Grupo II modificado comprende porciones directoras.
4. La molécula de ADN de la Reivindicación 3, donde las porciones directoras permiten la inserción del transcripto de ARN del intrón de Grupo II modificado en un sitio dentro de una molécula de ADN en la célula bacteriana de la clase Clostridia.
5. La molécula de ADN de una cualquiera de las reivindicaciones anteriores, donde la molécula de ADN es un plásmido, por ejemplo un plásmido que es un vector de lanzadera de Escherichia coli - Clostridia.
6. La molécula de ADN de una cualquiera de las reivindicaciones anteriores, que además comprende un gen codificador de una proteína que permite la transferencia conjugativa desde Escherichia coli a una célula bacteriana de la clase
Clostridia.
7. La molécula de ADN de una cualquiera de las reivindicaciones anteriores, donde el promotor ligado operativamente a la región codificadora del marcador seleccionable es el promotor de los genes thl o ptb o adc de C. acetobutylicum, o los genes fdx o cpe de C. perfringens.
8. La molécula de ADN de una cualquiera de las reivindicaciones anteriores, donde el intrón de Grupo I está situado dentro o secuencia arriba de la región codificadora del marcador seleccionable.
9. La molécula de ADN de una cualquiera de las reivindicaciones anteriores, donde el promotor ligado operativamente al intrón de Grupo II modificado es un promotor inducible, tal como un promotor inducible por IPTG o xilosa.
1. Un método para introducir una molécula de ácido nucleico en un sitio de una molécula de ADN en una célula bacteriana de la clase Clostridia, donde el método comprende las etapas de:
(i) proporcionar una célula bacteriana de la clase Clostridia con la molécula de ADN de una cualquiera de las Reivindicaciones 1 a 9 y una molécula de ADN capaz de expresar una transcriptasa inversa de Grupo II codificada por el intrón; y
(ii) cultivar la célula bacteriana en condiciones que permitan la remoción del intrón de Grupo I del transcripto de ARN del intrón de Grupo II modificado y la inserción de dicho transcripto de ARN que contiene el gen de marcador seleccionable o una de sus copias de ADN en dicho sitio;
donde el sitio está de preferencia situado dentro de un gen o dentro de una porción de ADN que afecte la expresión de un gen y/o está situado dentro del genoma bacteriano; y donde, en la etapa (i), la molécula de ADN capaz de expresar una transcriptasa inversa de Grupo II codificada por el intrón es opcionalmente la misma que la molécula de ADN de una cualquiera de las Reivindicaciones 1 a 9.
11. El método de la Reivindicación 1 que además comprende cultivar la célula bacteriana de la clase Clostridia en condiciones que permiten la expresión del marcador seleccionable; y que además, de preferencia también comprende seleccionar la célula bacteriana de la clase Clostridia basada sobre un fenotipo alterado, conferido por el marcador seleccionable; y que además, de preferencia también comprende la etapa de aislar un único clon de células derivado de la célula obtenida en la Reivindicación 1.
12. La molécula de ADN de una cualquiera de las Reivindicaciones 1 a 9 o el método de las Reivindicaciones 1 u 11, donde la célula bacteriana de la clase Clostridia es del género Clostridium, tal como C. thermocellum o C. acetobutylicum o C. difíicile o C. botulinum o C. perfringens o C. beijerinckii o C. tetani o C. cellulyticum o C. septicum.
13. Un método para dirigir una molécula de ácido nucleico a un sitio seleccionado de una molécula de ADN en una célula bacteriana de la clase Clostridia, donde el método comprende:
(i) proporcionar una célula bacteriana de la clase Clostridia con la molécula de ADN de la Reivindicación 3 y una molécula de ADN capaz de expresar una transcriptasa inversa de Grupo II codificada por el intrón; y
(ii) cultivar la célula bacteriana en condiciones que permiten remover el intrón de Grupo I del transcripto de ARN del intrón de Grupo II modificado e insertar dicho transcripto de ARN, que contiene el gen de marcador seleccionable o una de sus copias de ADN en dicho sitio seleccionado; donde el sitio seleccionado es de preferencia un gen o una porción de ADN que afecta la expresión de un gen.
14. Una célula bacteriana muíante de la clase Clostridia que comprende una molécula de ADN que comprende un sitio que comprende un ácido nucleico, donde el ácido nucleico comprende:
un intrón de Grupo II modificado que no expresa la transcriptasa inversa codificada por el intrón, pero que contiene un gen de marcador seleccionable en la orientación inversa respecto del intrón de Grupo II modificado, donde el gen de marcador seleccionable comprende una región codificadora de un marcador seleccionable y un promotor ligado operativamente a dicha región, donde el promotor es capaz de generar la expresión del marcador seleccionable codificado por una única copia del gen del marcador seleccionable en una cantidad suficiente para que el marcador seleccionable altere el fenotipo de la célula bacteriana de la clase Clostridia de manera tal que pueda ser distinguida de la célula bacteriana de la clase Clostridia carente del gen de marcador seleccionable, donde el marcador seleccionable confiere resistencia a eritromicina a la célula bacteriana de la clase Clostridia;
donde el sitio está situado de preferencia dentro de un gen o dentro de una porción de ADN que afecte la expresión de un gen y/o está situado dentro del genoma bacteriano.
15. Una molécula de ADN, tal como un plásmido, que comprende un gen de resistencia modificado que contiene un intrón de Grupo I situado en orientación inversa respecto del gen de resistencia modificado que altera la expresión de la resistencia, donde cuando se transcribe el intrón de Grupo I, es capaz de autoextraerse del transcripto de ARN, y donde la resistencia es resistencia a eritromicina.
16. La molécula de ADN de la Reivindicación 15, donde el gen de resistencia modificado comprende un promotor que es capaz de provocar la expresión de resistencia codificado por una única copia del gen de resistencia modificado en una cantidad suficiente para que la resistencia altere el fenotipo de una célula bacteriana de la clase Clostridia de manera tal que pueda ser distinguida de la célula bacteriana de la clase Clostridia carente del gen de resistencia modificado.
17. Uso de una molécula de ADN de conformidad con una cualquiera de las Reivindicaciones 1 a 9 o 15 o 16 en la preparación de una célula bacteriana muíante de la clase Clostridia.
Patentes similares o relacionadas:
Sistema de expresión bacteriano cistrónico doble, del 25 de Marzo de 2020, de LUPIN LIMITED: Un procedimiento para la producción de un anticuerpo o un fragmento del mismo que comprende las etapas de: (i) transformar una célula hospedadora bacteriana […]
Partículas de transducción no replicativas y sistemas indicadores basados en partículas de transducción, del 15 de Enero de 2020, de Geneweave Biosciences Inc: Un sistema de empaquetamiento de células bacterianas para empaquetar una molécula de ácido nucleico indicadora en una partícula de transducción no replicativa (NRTP) para su introducción […]
Método de construcción de biblioteca de péptidos y vectores relacionados, del 1 de Enero de 2020, de Hunan Zonsen Peplib Biotech Co., Ltd: Método integrado de construcción de una biblioteca de péptidos completa que contiene todos los péptidos posibles para el tamaño específico, comprendiendo el método: (i) […]
 Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Transporte de proteínas basado en bacterias, del 14 de Agosto de 2019, de Universität Basel: Un vector que comprende, en la dirección 5' a 3': un promotor; una primera secuencia de ADN que codifica una señal de transporte procedente […]
Sistema de expresión y secreción, del 26 de Junio de 2019, de F. HOFFMANN-LA ROCHE AG: Una molécula de ácido nucleico que codifica un primer polipéptido y un segundo polipéptido, enlazada de forma funcional a una secuencia señal que codifica […]
 Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Síntesis microbiana de aldehídos y alcoholes correspondientes, del 17 de Junio de 2019, de Easel Biotechnologies, LLC: Un método para producir un alcohol, que comprende: cultivar una pluralidad de células microbianas en un medio de fermentación, en donde las […]