Métodos y composiciones.
Colección de polipéptidos codificados genéticamente de complejos que comprenden un ácido nucleico que codifica un polipéptido,
en donde:
(i) el polipéptido codificado por el ácido nucleico está unido al ácido nucleico;
(ii) un compuesto conector unido a dicho polipéptido;
(iii) el compuesto conector está unido al polipéptido mediante al menos tres enlaces covalentes independientes; y
(iv) la colección es una genoteca exposición de ARNm, exposición de ADN, exposición en levadura, exposición en ribosomas o exposición en bacterias.
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E12151953.
Solicitante: Bicycle Therapeutics Limited.
Nacionalidad solicitante: Reino Unido.
Dirección: Meditrina, Babraham Research Campus Cambridge CB22 3AT REINO UNIDO.
Inventor/es: HEINIS,CHRISTIAN, WINTER,GREGORY.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- A61K47/48
- C12N15/10 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Procedimientos para el aislamiento, la preparación o la purificación de ADN o ARN (preparación química de ADN o ARN C07H 21/00; preparación de polinucleótidos no estructurales a partir de microorganismos o con la ayuda de enzimas C12P 19/34).
- C40B30/04 C […] › C40 TECNOLOGIA COMBINATORIA. › C40B QUIMICA COMBINATORIA; BIBLIOTECAS, p. ej. QUIMIOTECAS (bibliotecas combinatorias in silico de ácidos nucleicos, proteínas o péptidos G16B 35/00; química combinatoria in silico G16C 20/60). › C40B 30/00 Procedimientos de selección de bibliotecas. › midiendo la capacidad para unirse específicamente a una molécula diana, p. ej. unión anticuerpo-antígeno, unión receptor-ligando.
- C40B40/02 C40B […] › C40B 40/00 Bibliotecas per se , p. ej. arrays, mezclas. › Bibliotecas contenidas en o exhibidas por microorganismos, p. ej. bacterias o células animales; Bibliotecas contenidas en o exhibidas por vectores, p. ej. plásmidos; Bibliotecas que únicamente contienen microorganismos o vectores.
- G01N33/68 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › en los que intervienen proteínas, péptidos o aminoácidos.
PDF original: ES-2509959_T3.pdf


Fragmento de la descripción:
Métodos y composiciones Campo de la invención
La invención se refiere a la modificación y al constreñimiento de polipéptidos, en particular a polipéptidos codificados genéticamente en complejos con el ácido nucleico que los codifica.
Antecedentes de la invención
La generación de las moléculas con afinidad y especificidad elevadas por las dianas biológicas es un problema central en la química, en la biología y en las ciencias farmacéuticas. En particular, los ligandos de fijación son importantes para la creación de fármacos que son capaces de interferir con los procesos biológicos. La creación de ligandos que se fijan a un ligando diana elegido suele implicar un proceso de generación de una colección de posibles moléculas de fijación y el análisis de las propiedades de fijación de dichas moléculas.
Mientras que las técnicas biológicas de selección in vitro se utilizaron con eficacia para aislar grandes estructuras biopoliméricas, tales como anticuerpos, hasta la fecha eran menos practicables para el aislamiento de fármacos que fueran moléculas pequeñas. Las técnicas biológicas de selección in vitro se limitan por lo general a polímeros biológicos tales como polipéptidos, ARN o ADN. Los biopolímeros pequeños como, por ejemplo, los péptidos, también se pueden fijar a dianas biológicas, pero pueden carecer de flexibilidad conformacional y pueden ser propensos a la degradación proteolítica en los líquidos corporales. Además está la debilidad de la afinidad de unión de los péptidos lineales pequeños. Se conocen diferentes estrategias de circularización para constreñir las colecciones de péptidos pequeños codificados genéticamente. Se sabe que los repertorios de péptidos expuestos en fagos, por ejemplo, se circularizan mediante la oxidación de dos restos de cisteína flanqueantes. Se sabe que las colecciones de péptidos cíclicos codificados por ARNm se generan al formar un enlace entre la amina aminoterminal y un resto de lisina del péptido con un reactivo químico para reticulación. Esta estrategia se utilizó para aislar macrociclos insensibles a la óxido-reducción que se fijan a la proteína de señalización Gai1 (Millward, S. W. et al., ACS Chem. Biol., 2007). También se conocen diferentes estrategias para ser usadas en la incorporación de bloques de construcción no naturales en colecciones de polipéptidos codificados genéticamente para expandir la diversidad de las colecciones o para insertar propiedades que no pueden proporcionar los aminoácidos naturales. Sin embargo, las estrategias permitieron sólo la adición de un número limitado de añadidos orgánicos pequeños a los polipéptidos lineales codificados genéticamente. Frankel, A. et al., por ejemplo, habían incorporado aminoácidos no naturales a polipéptidos naturales que estaban codificados mediante exposición de ARNm (Frankel, A et al., Chem. Biol., 2003). Jespers L. et al. habían conectado químicamente una molécula indicadora fluorescente a un lazo hipervariable de un repertorio de anticuerpos expuestos en fagos, y seleccionaron del repertorio los que se fijaban a antígenos (Jespers, L., et al., Prot. Eng., 2004). Dwyer, M. A. et al. habían juntado péptidos sintéticos a un repertorio de péptidos expuestos en fagos mediante la ligación química nativa para la generación de una colección de inhibidores de proteasas que contenían un aminoácido no natural (Dwyer, M. A. et al., Chemistry & Biology, 2000). También se han conectado moléculas orgánicas pequeñas a repertorios combinatorios de péptidos codificados por ARNm. El equipo de investigación de Roberts, R. W. había pegado un resto de penicilina a una posición fija de una colección de péptidos por exposición de ARNm para seleccionar los inhibidores de la proteína 2a de fijación a la penicilina en Staphylococcus aureus (Li, S. y Roberts, W. R., Chem & Biol., 2003).
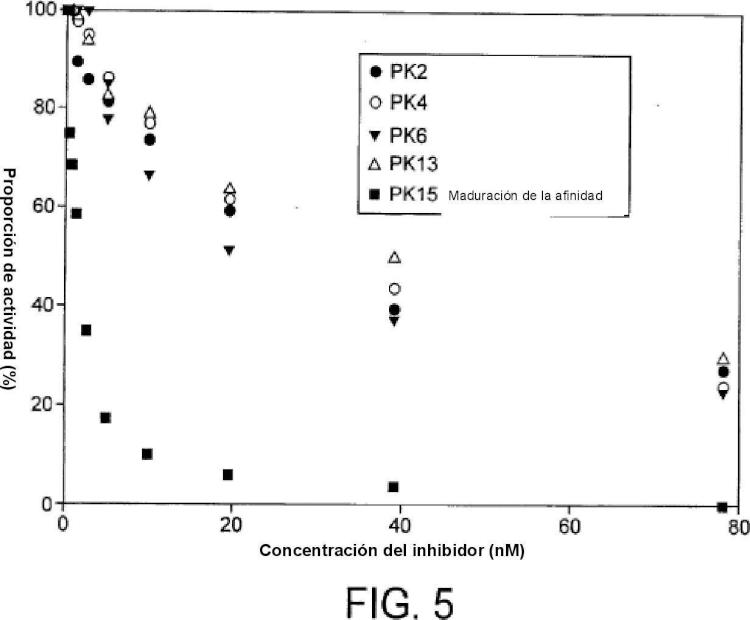
Se han propuesto varias metodologías para aplicar la selección in vitro a colecciones combinatorias de compuestos que tienen arquitecturas moleculares más diversas (p. ej., moléculas ramificadas) y que están formadas por bloques de construcción no naturales. A diferencia de los procedimientos biológicos de selección in vitro, estas metodologías utilizan estrategias químicas para pegar etiquetas de ADN a las moléculas orgánicas pequeñas. Brenner S. y Lerner R. A. habían propuesto un proceso de síntesis combinatoria paralela para codificar cada miembro de una gran quimioteca con secuencias nucleotídicas únicas en perlas (Brenner, S. y Lerner, R. A., PNAS, 1992). Después de que la entidad química se fija a la diana, el código genético se descodifica por secuenciación de la etiqueta nucleotídica. Liu D. R. y colaboradores habían conjugado una pequeña colección de moléculas orgánicas a oligonucleótidos de ADN y realizaron selecciones por afinidad con antígenos diferentes (Doyon, J. B. et al., JACS, 2003). Neri D. y colaboradores habían generado grandes repertorios de parejas moleculares mediante el autoensamblaje de subquimiotecas más pequeñas codificadas por ADN mediante la hibridación de dos hebras de ADN (Melkko, S. et al., Nature Biotechnol., 2004). La metodología se utilizó con éxito para la maduración de la afinidad de los ligandos que son moléculas pequeñas. Halpin D. R. y Harris P. B. desarrollaron una estrategia para la evolución in vitro de quimiotecas combinatorias que implica la amplificación de determinados compuestos para realizar varios ciclos de selección (Halpin, D. R. y Harbury, P. B., PLOS Biology, 2004). Woiwode T. F. et al. pegaron colecciones de compuestos sintéticos a las proteínas de la cubierta de las partículas de bacteriófago de tal forma que la identidad de la estructura química está especificada en el genoma del fago (Woiwode, T. F., Chem. & Biol., 2003). Todas estas estrategias que emplean entidades químicas especificadas por ADN han resultado ser eficientes en experimentos modelo y algunos incluso han producido nuevas moléculas pequeñas aglutinantes. Sin embargo, quedó muy claro que la codificación de colecciones grandes de compuestos y la amplificación de los compuestos
seleccionados es mucho más exigente que los procedimientos equivalentes en los sistemas biológicos de selección.
Jespers et al. (2004, «Protein engineering design and selection», volumen 17, n.° 10, páginas 709-713) describen la selección de biosensores ópticos a partir de colecciones de anticuerpos quimiosintéticos. Este documento trata de la unión de una molécula indicadora fluorescente a través del lazo hipervariable de un repertorio de anticuerpos expuestos en fagos. En particular, este documento describe la conexión de una molécula indicadora fluorescente a un lazo hipervariable (región determinante de la complementariedad o CDR) de un repertorio de anticuerpos sintéticos. La molécula indicadora fluorescente está conectada mediante un enlace covalente único a un resto de cisterna introducido artificialmente en el lazo hipervariable. Se realiza la unión del uno al otro. Los restos cisteína en las partículas de fagos se redujeron con DTT y se retiró el exceso del agente reductor mediante precipitación convencional con polietilenglicol (PEG) como se conoce bien en la técnica.
Dwyer et al. describen la exposición en fagos biosintéticos, que describe una nueva herramienta de ingeniería de proteínas que combina la diversidad química y genética. Dwyer et al. (Chem. Biol. 2000, volumen 7, n.° 4, páginas 263-274) describen la ligación química de un péptido sintético que tiene un aminoácido no natural a una colección de péptidos sintéticos que comprende los principales restos estructurales de una proteína de interés. La motivación para realizar esto fue para generar un amplio abanico de secuencias de proteasas, y que cada una contenga un segmento constante que incorpora un aminoácido no natural. El péptido sintético que comprende el aminoácido no natural se pegó simplemente mediante ligación química nativa, lo que dio lugar al acoplamiento de los dos fragmentos peptídicos. No se describe ningún compuesto conector. No se describe la adhesión de ninguna molécula pequeña. No se consiguió ninguna constricción ni restricción conformacional del péptido resultante. No se describe ninguna formación de enlaces covalentes entre determinados restos y la cadena polipeptídica.
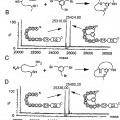
Diferentes equipos de investigación han sujetado previamente polipéptidos con restos de cisteína a una estructura molecular sintética (Kemp, D. S y McNamara, P. E., J. Org. Chem. 1985; Timmerman, P. et al., ChemBioChem, 2005). Meloen y colaboradores habían utilizado tris(bromometil)benceno y moléculas relacionadas para la ciclación cuantitativa y rápida de varios lazos peptídicos sobre armazones sintéticos... [Seguir leyendo]
Reivindicaciones:
1. Colección de polipéptidos codificados genéticamente de complejos que comprenden un ácido nucleico que codifica un polipéptido, en donde:
(i) el polipéptido codificado por el ácido nucleico está unido al ácido nucleico;
(ii) un compuesto conector unido a dicho polipéptido;
(Ni) el compuesto conector está unido al polipéptido mediante al menos tres enlaces covalentes independientes; y
(iv) la colección es una genoteca exposición de ARNm, exposición de ADN, exposición en levadura, exposición en ribosomas o exposición en bacterias.
2. Colección de acuerdo con la reivindicación 1, en donde el compuesto conector tiene una simetría molecular que corresponde al número de enlaces covalentes mediante los cuales se une al polipéptido.
3. Colección de acuerdo con la reivindicación 2, en el que el compuesto conector posee simetría molecular ternaria y el compuesto conector está unido al polipéptido mediante tres enlaces covalentes.
4. Colección de acuerdo con cualquiera de las reivindicaciones anteriores, en donde el compuesto conector comprende un grupo químico estructuralmente rígido.
5. Colección de acuerdo con la reivindicación 4, en donde el compuesto conector comprende tris(bromometil)benceno (TBMB).
6. Colección de acuerdo con cualquiera de las reivindicaciones anteriores, en donde dicho polipéptido comprende un resto de cisteína, y en donde al menos uno de dichos tres enlaces covalentes independientes para la unión de dicho compuesto conector al polipéptido comprende un enlace con dicho resto de cisteína.
7. Procedimiento para fabricar un complejo, en donde dicho procedimiento comprende
(i) proporcionar el polipéptido codificado por un ácido nucleico, en donde el polipéptido está unido al ácido
nucleico mediante exposición de ARNm, exposición de ADN, exposición en levadura, exposición en ribosomas o exposición en bacterias;
(ii) proporcionar un compuesto conector; y
(iii) unir dicho compuesto conector a dicho polipéptido mediante la formación de al menos tres enlaces covalentes entre dicho compuesto conector y el polipéptido.
8. Procedimiento de acuerdo con la reivindicación 7, en donde dicho procedimiento comprende la etapa adicional de
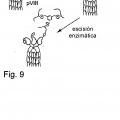
(iv) escindir uno o más enlaces de la cadena polipeptídica.
9. Procedimiento de acuerdo con la reivindicación 8, en donde dicha etapa de escisión comprende poner en contacto dicho polipéptido con una proteasa.
10. Complejo obtenido mediante el procedimiento de cualquiera de las reivindicaciones 7 a 9.
11. Procedimiento para identificar un complejo de acuerdo con cualquiera de las reivindicaciones anteriores, que es capaz de fijarse a un ligando, en donde el procedimiento comprende:
(i) proporcionar un complejo de acuerdo con cualquiera de las reivindicaciones anteriores
(ii) poner en contacto dicho complejo con el ligando, y
(iii) seleccionar los complejos que se fijan a dicho ligando.
12. Procedimiento de acuerdo con la reivindicación 11, que además comprende determinar la secuencia del ácido nucleico de dicho complejo.
13. Procedimiento de acuerdo con la reivindicación 11, que además comprende la etapa de fabricar el complejo aislado capaz de fijarse a dicho ligando.
14. Procedimiento de acuerdo con la reivindicación 11, que además comprende la etapa de fabricar un conjugado polipéptido-compuesto conector aislado o identificado mediante un procedimiento de la invención, en donde dicha fabricación comprende la unión del compuesto conector al polipéptido, en donde dicho polipéptido se expresa de
manera recombinante o se sintetiza por procedimientos químicos.
15. Procedimiento de acuerdo con la reivindicación 14, que además comprende la etapa de extender el polipéptido en uno o más de los extremos amino o carboxilo del polipéptido.
16. Procedimiento de acuerdo con cualquiera de las reivindicaciones 14 o 15, que además comprende la etapa de 5 conjugar a otro polipéptido dicho conjugado polipéptido-compuesto conector.
17. Procedimiento de acuerdo con la reivindicación 16, en donde dicha conjugación se realiza
(i) añadiendo otra cisterna más al polipéptido después de unir covalentemente el compuesto conector, y
(ii) conjugar dicho polipéptido a dicho otro polipéptido mediante un enlace disulfuro con dicha cisterna adicional.
Patentes similares o relacionadas:
Inmunomoduladores, del 29 de Julio de 2020, de BRISTOL-MYERS SQUIBB COMPANY: Un compuesto de la fórmula (I) **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo, en donde: A se selecciona de **(Ver fórmula)** en donde: […]
Métodos y composiciones para el diagnóstico y pronóstico de lesión renal e insuficiencia renal, del 29 de Julio de 2020, de Astute Medical, Inc: Un método para evaluar el estado renal en un sujeto, que comprende: realizar una pluralidad de ensayos configurados para detectar una […]
Neuregulina para tratar la insuficiencia cardíaca, del 29 de Julio de 2020, de Zensun (Shanghai) Science & Technology, Co., Ltd: Neuregulina para usar en un método para tratar la insuficiencia cardíaca crónica en un paciente, donde el paciente tiene un nivel plasmático de NT-proBNP […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Método para llevar a cabo el seguimiento de la enfermedad de Gaucher, del 15 de Julio de 2020, de Centogene GmbH: Un método para determinar la evolución de la enfermedad de Gaucher en un sujeto, que comprende la etapa de determinar en varios puntos en el […]
Procedimiento para evaluación de la función hepática y el flujo sanguíneo portal, del 15 de Julio de 2020, de The Regents of the University of Colorado, a body corporate: Procedimiento in vitro para la estimación del flujo sanguíneo portal en un individuo a partir de una única muestra de sangre o suero, comprendiendo el procedimiento: […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos de monitorización terapéutica de profármacos de ácido fenilacético, del 24 de Junio de 2020, de Immedica Pharma AB: Glicerilo tri-[4-fenilbutirato] (HPN-100) para su uso en un método para tratar un trastorno del ciclo de la urea en un sujeto que tiene discapacidad […]