Método para la producción de hidroxiectoína.
Método para la producción de hidroxiectoína.
La presente invención se refiere a un polinucleótido aislado que comprende los genes de producción de ectoína e hidroxiectoína,
preferiblemente con al menos dos copias del gen que codifica para la enzima ectoína hidroxilasa, y cuya expresión está dirigida por al menos un promotor activo en condiciones de baja salinidad y temperatura. Además, la presente invención se refiere al vector y la célula que comprende dicho polinucleótido, y a un método de producción de ectoína e hidroxiectoína basado en dicha célula. Preferiblemente, dicha célula es de la especie Chromohalobacter salexigens y es incapaz de expresar los genes ectA, ectB, ectC y ectD nativos.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201200263.
Solicitante: UNIVERSIDAD DE SEVILLA.
Nacionalidad solicitante: España.
Inventor/es: RODRÍGUEZ DE MOYA VERA,Javier, ARGANDOÑA BERTRÁN,Montserrat, NIETO GUTIÉRREZ,Joaquín José, VARGAS MACÍAS,Carmen.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/52 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Genes que codifican enzimas o proenzimas.
- C12N15/62 C12N 15/00 […] › Secuencias de ADN que codifican proteínas de fusión.
- C12P17/12 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 17/00 Preparación de compuestos heterocíclicos que contienen O, N, S, Se o Te como únicos heteroátomos del ciclo (C12P 13/04 - C12P 13/24 tienen prioridad). › que contienen un ciclo de seis miembros.
Fragmento de la descripción:
MÉTODO PARA LA PRODUCCIÓN DE HIDROXIECTOÍNA
La presente invención se refiere a un polinucleótido aislado que comprende los
genes de producción de ectoína e hidroxiectoína, preferiblemente con al menos
5 dos copias del gen que codifica para la enzima ectoína hidroxilasa, y cuya
expresión está dirigida por al menos un promotor activo en condiciones de baja
salinidad y temperatura. Además, la presente invención se refiere al vector y la
célula que comprende dicho polinucleótido, y a un método de producción de
ectoína e hidroxiectoína basado en dicha célula. Por tanto, la invención se podría
1 O encuadrar en el campo de la Biotecnología.
ESTADO DE LA TÉCNICA
Los microorganismos halófilos y halotolerantes se adaptan a las altas
15 concentraciones salinas mediante cambios en la composición lipídica de sus
membranas y la acumulación en su citoplasma de solutos con bajo peso
molecular. Así, las arqueas aerobias halófilas extremas de la familia
Halobacteriaceae, la bacteria halófila extrema Salinibacter ruber, así como serie
de bacterias halófilas moderadas Gram positivas, acetogénicas y sulfato
20 reductoras, del orden Ha/anaerobia/es acumulan concentraciones muy elevadas
de iones inorgánicos, principalmente K+ y cr, hasta niveles que se asemejan a los
de la concentración salina del medio externo. Sin embargo, el resto de
microorganismos halófilos, en especial aquellos con mayor adaptación a las
concentraciones salinas elevadas, acumulan en su citoplasma grandes
25 cantidades de compuestos orgánicos específicos, solubles, que actúan como
osmolitos para equilibrar la presión osmótica externa. Se trata de moléculas
orgánicas de bajo peso molecular, muy solubles y generalmente de carga neutra.
Se han denominado quot;solutos compatiblesquot; porque no interfieren con el
metabolismo celular, y pueden acumularse hasta alcanzar concentraciones muy
30 elevadas, del orden de 1 mol/kg de agua. Además de su función como
osmoprotectores, los solutos compatibles pueden ejercer otras funciones de
protección, por lo que poseen un gran potencial desde el punto de vista aplicado.
Es de gran interés la aplicación industrial de estos compuestos en la tecnología
de enzimas (biosensores, PCR, entre otros) , en la fabricación y diseño de nuevos
fármacos útiles en cosmética y dermofarmacia, en agricultura, en medicina y, en
general, como agentes que favorecen la conservación de biomoléculas, células
completas y tejidos.
5
Entre los solutos compatibles que se han investigado, las ectoínas son las que
han mostrado las mayores propiedades estabilizantes. Las aplicaciones de las
ectoínas han sido ampliamente revisadas por (Pastor y col., 2010 Biotechnology
Advances 6:782-801 ) , entre otras, se ha demostrado que pueden proteger
1 O enzimas, ADN y la estructura de las proteínas frente a distintos tipos de estrés.
Así mismo, se han postulado como potenciales moléculas protectoras frente a
diferentes enfermedades por su capacidad estabilizadora de las proteínas. Por
último, son ampliamente empleadas en preparaciones dermatológicas pos su
capacidad protectora de la piel.
15
El término ectoínas engloba a dos compuestos tetrahidropirimidínicos cíclicos: la
ectoína (ácido 1 , 4, 5, 6-tetrahidro-2-metil-4-pirimidinocarboxílico) y su derivado
hidroxilado, la hidroxiectoína (ácido 1 , 4, 5, 6-tetrahidro-5-hidroxi-2-metil-4-
pirimidinocarboxílico) . Ambas, ectoína e hidroxiectoína, son junto con la betaína,
20 los solutos compatibles mayoritariamente sintetizados por bacterias halófilas
Gram negativas heterótrofas y también por un amplio número de bacterias Gram
positivas halófilas moderadas y halotolerantes. La ruta biosintética de la ectoína
se elucidó a nivel bioquímico por primera vez en Halomonas elongata y consta de
tres etapas enzimáticas que se originan a partir del aspartato semialdehído,
25 intermediario en el metabolismo de aminoácidos, siendo el diaminobutirato (DA) y
el Ny-acetildiaminobutirato (NADA) compuestos intermediarios de la ruta. El
aspartato semialdehído proviene de la fosforilación del L-aspartato y su posterior
reducción a aspartato semialdehído. El aspartato semialdehído es convertido en
ácido diaminobutírico (DA) mediante la enzima diaminobutirato transaminasa. A
30 continuación, este compuesto es acetilado hasta Ny-acetildiaminobutirato (NADA) ,
interviniendo la enzima diaminobutirato acetiltransferasa. Finalmente, mediante la
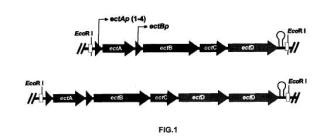
condensación del NADA por acción de la ectoína sintasa, se obtiene la ectoína.La región genética implicada en la síntesis de ectoína comprende tres genes, ectA, ectB y ectC, que codifican las enzimas ácido diaminobutírico acetiltransferasa, ácido diaminobutírico transaminasa y ectoína sintasa, respectivamente. Estos tres genes se encuentran habitualmente en un mismo operón, aunque existen excepciones donde encontramos clústeres incompletos o incluso microorganismos que sólo poseen el gen ectC. La principal ruta biosintética para la hidroxiectoína implica la hidroxilación directa de la ectoína, mediante una reacción catalizada por la enzima ectoína hidroxilasa (EctD o ThpD) , que fue caracterizada en Chromohalobacter salexigens (García-Estepa y col., 2006 J. Bacteriol. 188:3774-84) y Streptomyces chr y somallus. Además, se ha sugerido una ruta secundaria para la síntesis de hidroxiectoína en C. salexigens que partiría del precursor NADA. Este soluto compatible es más común en bacterias halófilas Gram positivas como Brevibacterium linens o Marinococcus halophilus, aunque frecuentemente es sintetizado en cantidades menores junto a la ectoína en microorganismos productores de la misma. A pesar de que su estructura química es muy parecida a la de la ectoína, la hidroxiectoína confiere mayor protección frente a los choques térmicos (García-Estepa y col., 2006 J. Bacteriol. 188:3774-84) y la desecación. Estas propiedades adicionales han propiciado la aparición de estudios sobre procesos industriales orientados a la producción específica del derivado hidroxilado de la ectoína (Pastor y col., 2010 Biotechnology Advances 6:782-801) .
En el caso concreto de la producción de ectoínas por C. salexigens, las concentraciones de ectoína e hidroxiectoína alcanzan su máximo en la fase estacionaria de crecimiento y la acumulación de ectoína se produce principalmente en respuesta a una elevada salinidad (14, 5% (p/v) NaCI, 37 °C) , mientras que la de hidroxiectoína se produce frente a una elevada salinidad y temperatura (14, 5% (p/v) NaCI, 45 °C) (García-Estepa y col., 2006 J. Bacteriol. 188:3774-84) . La transcripción de los genes responsables de la síntesis de ectoína (ectABC) implica a múltiples promotores (PectA 1-4, PectB) que permiten que el sistema sea regulado por diversos factores ambientales como la salinidad, la temperatura, la presencia de osmoprotectores o el hierro. La expresión de fusiones transcripcionales PectA-IacZ y PectB-IacZ es máxima en faseestacionaria a elevada salinidad, aunque mantiene unos niveles mínimos a baja
salinidad (0, 75 M NaCI) , especialmente la fusión PectA-IacZ, lo que sugiere que el
sistema ectABC es semiconstitutivo.
5 El creciente interés comercial de las ectoínas ha posibilitado el desarrollo de
muchas estrategias para su producción biotecnológica. La principal ventaja de los
procesos de producción biológica de ectoínas frente a su síntesis química reside
en su mayor especificidad y su menor complicación al ser un proceso con menos
etapas de separación de los productos y con menor generación de productos
1O secundarios. Entre las posibles desventajas que ofrecen los procesos
biotecnológicos se encuentran la gran cantidad de nutrientes necesarios y el
control exhaustivo a que deben someterse las condiciones de fermentación (pH,
aireación, etc) . Además, la elevada concentración salina del medio de cultivo es
un problema importante en estos procesos, al ser la causante de la corrosión de
15 los equipos, además de limitar la tasa de crecimiento, provocando una
disminución en la producción de ectoínas. Aun así, los procesos de fermentación
son actualmente el método de elección para la producción de ectoínas (Pastor y
col., 2010 Biotechnology Advances 6:782-801) . El proceso más utilizado
actualmente para la producción de ectoína es conocido como quot;bacteria/ milkingquot;, e
20 implica el cultivo aerobio de H. elongata a 25°C en un medio salino con 15 % (p/v)
de NaCI (2, 57 M NaCI) hasta conseguir una gran densidad celular. Cuando el
cultivo...
Reivindicaciones:
1. Un polinucleótido aislado que comprende:
(a) la secuencia codificante del gen ectA, donde dicho gen codifica la enzima diaminobutirato acetiltransferasa;
(b) la secuencia codificante del gen ectB, donde dicho gen codifica la enzima diaminobutirato transaminasa;
(e) la secuencia codificante del gen ectC, donde dicho gen codifica la enzima ectoína sintasa; y
(d) la secuencia codificante del gen ectD, donde dicho gen codifica la enzima ectoína hidroxilasa.
2. El polinucleótido según la reivindicación 1, caracterizado porque comprende al menos dos copias de la secuencia codificante del gen ectD, donde dicho gen codifica la enzima ectoína hidroxilasa.
3. El polinucleótido según cualquiera de las reivindicaciones 1 o 2, caracterizado porque además comprende al menos un promotor.
4. El polinucleótido según la reivindicación 3, caracterizado porque el promotor permite la expresión en condiciones ambient~les de salinidad entre 2 y 6% peso/volumen.
5. El polinucleótido según la reivindicación 4, caracterizado porque el promotor permite la expresión en condiciones ambientales de salinidad entre 3 y 5% peso/volumen.
6. El polinucleótido según cualquiera de las reivindicaciones 3 a 5, caracterizado porque el promotor permite la expresión en condiciones ambientales de temperatura entre 32 y 40 °C.
7. El polinucleótido según la reivindicación 4, caracterizado porque el promotor permite la expresión en condiciones ambientales de temperatura entre 34 y 38 °C.
5 8. El polinucleótido según cualquiera de las reivindicaciones 3 a 7, caracterizado porque el promotor permite la expresión en condiciones ambientales de salinidad entre 2 y 6% peso/volumen y temperatura entre 32 y 40 °C.
1 O 9. El polinucleótido según la reivindicación 8, caracterizado porque el promotor permite la expresión en condiciones ambientales de salinidad entre 3 y 5% peso/volumen y temperatura entre 34 y 38 °C.
15 10. El polinucleótido según cualquiera de las reivindicaciones 3 a 9, caracterizado porque al menos uno de los promotores es la secuencia nucleotídica promotora del gen ectA.
11. El polinucleótido según la reivindicación 10, donde la secuencia nucleotídica promotora del gen ectA es SEQ ID NO: 5.
20 12. El polinucleótido según cualquiera de las reivindicaciones 1 a 11, donde la secuencia codificante del gen ectA para el enzima diaminobutirato acetiltransferasa es SEQ ID NO: 1.
25 13. El polinucleótido según cualquiera de las reivindicaciones 1 a 12, donde la secuencia codificante del gen ectB para el enzima diaminobutirato transaminasa es SEQ ID NO: 2.
30 14. El polinucleótido según cualquiera de las reivindicaciones 1 a 13, donde la secuencia codificante del gen ectC para el enzima ectoína sintasa es SEQ ID NO: 3.
15. El polinucleótido según cualquiera de las reivindicaciones 1 a 14, donde la secuencia codificante del gen ectD para el enzima ectoína hidroxilasa es SEQ ID N0:4.
16. Una construcción génica que comprende el polinucleótido según cualquiera de las reivindicaciones 1 a 15.
5 17. La construcción génica según la reivindicación 14, caracterizada porque es un vector de expresión.
1 O 18. La construcción gemca según la reivindicación 17, donde el vector de expresión comprende un origen de replicación de la familia Halomonadaceae y un origen de replicación de enterobacterias.
15 19. Una célula que comprende el polinucleótido según cualquiera de las reivindicaciones 1 a 15 o la construcción génica según cualquiera de las reivindicaciones 16 a 18.
20. La célula según la reivindicación 19 donde la especie es Chromohalobacter sa/exigens.
20 21. La célula según cualquiera de las reivindicaciones 19 o 20, donde dicha célula es incapaz de expresar los genes ectA, ectB, ectC y ectD nativos.
22. La célula según la reivindicación 21, donde dicha célula CH R137 de Chromohalobacter salexigens. es de la estirpe
25 23. Un método para la producción de ectoína e hidroxiectoína que comprende las siguientes etapas:
30 (a) cultivar una célula según cualquiera de las reivindicaciones 19 a 22, y (b) purificar la ectoína y la hidroxiectoína a partir del cultivo de (a) .
24. El método según la reivindicación 23 donde la etapa (a) se lleva a cabo en condiciones de salinidad de entre 2 y 6% peso/volumen.
25. El método según cualquiera de las reivindicaciones 23 o 24 donde la etapa
(a) se lleva a cabo en condiciones de salinidad de entre el 3 y 5% peso/volumen. 26. El método según cualquiera de las reivindicaciones 23 a 25 donde la etapa
(a) se lleva a cabo a una temperatura de entre 30 y 50° C. 27. El método según cualquiera de las reivindicaciones 23 a 26 donde la etapa
(a) se lleva a cabo a una temperatura de entre 32 y 40° C.
octAp (1.4) FIG.1 apt FIG. 2 vinc Pst I Hinc Ii hhe quot;flo Scd finc Acc Xho I Qra Apol Kpn PectA EcoR I BamH I PectB BamH I EcoR I lIntroduccion de un punto de corte BamH I por PCR +bamectC FW EcoR I pME2.1 -barnpME FW opertITI EcoR I .4— +b am ectC RV Eliminacion de un punto de corte BamH I por PCR BamH I EcoR 1 -barnpME RV pME2.2 FIG. 3 A Psi I Digestion BamH I ectZ Sal I lIntroduccion de un punto de corte Bc1 I por PCR Bat I +bclectD FW • —÷ Sal I pEctD.1 4— +bclectD RV DigestiOn BcI I V pME2.2 Ligacion + Transformacion Digestion EcoR I pHS15 gt; Ligacion + Transformacian pEctABCD Digestion EcoR I pHS15ABCD FIG. 3 B PectA EcoR I I Introduccion de un punto de corte BamH I por PCR EcoR 1 bamectCD FW BamH 1 EcoR 1 1 Eliminacion de un punto de code BamH I por PCR EcoR I Digestion BamH I bamectCD RV pEctABCD.1 -bamectD FW BamHI EcoR 1 4— -bamectD RV Ligacion + Transformacion 4.•-• Digest iOn BcI I pEctABCD.2 pEctD.1 pEctDD 1 Digestion EcoR I Digestion EcoR I Ligacion + Transformacion pHS15DD pHS15 FIG. 4 LISTADO DE SECUENCIAS lt;110gt; Universidad de Sevilla lt;120gt; METOD0 PARA LA PRODUCCION DE HIDROXIECTOINA lt;130gt; ES1650.24 lt;140gt; lt;141gt; 2012-03-08 lt;160gt; 12 lt;170gt; PatentIn version 3.5 lt;210gt; 1 lt;211gt; 585 lt;212gt; DNA lt;213gt; Chromohalobacter salexigens lt;220gt; lt;223gt; Secuencia codificante del gen ectA para la enzima diaminobutirato acetiltransferasa lt;400gt; 1 atggatatga cgcctacaac cgagaacttc acgccttccg ccgatctggc ccgcccgagc 60 gttgccgaca cggtcatcgg cagcgcgaag aaaaccctct tcatccgtaa gcccacgacc 120 gacgacgggt ggggcattta cgaactcgtc aaggcatgcc cgccactgga cgtcaactcg 180 gggtacgcct atctgctgct tgccacccag ttccgcgata cctgcgccgt ggctacggac 240 gaagaagggg agatcgtcgg tttcgtctcc ggctacgtca aacgcaacgc accggacacc 300 tactttctat ggcaagtcgc cgtcggcgag aaagcgcggg gcacgggcct ggcgcgacgc 360 ctcgtcgaag ccgtgctgat gcgccccggc atgggcgatg tccgccatct cgaaactacc 420 atcacgcccg acaatgaggc atcgtgggga ctgttcaagc gccttgccga tcgctggcaa 480 gcccccctga acagccgtga atacttctcc acggggcagt tgggtggcga gcacgatccg 540 gaaaatctcg tccgtatcgg cccgttcgag ccgcaacaaa tttga 585 lt;210gt; 2 lt;211gt; 1272 lt;212gt; DNA lt;213gt; Chromohalobacter salexigens lt;220gt; lt;223gt; secuencia codificante del gen ectB para la enzima diaminobutirato transaminasa lt;400gt; 2 atgcagaccc agattctCga acgcatggaa tccgaagtcc ggacctattc acgttctttt 60 cctaccgttt tcactgaagc caagggcgcg cgcctgcatg ccgaggacgg caaccagtac 120 atcgattttc tcgccggcgc cggcacgctc aactacggtc acaaccaccc caagctcaag 180 caggcactgg ccgattacat cgcctccgat ggcatcgtcc atggtctgga catgtggagc 240 gcggccaagc gcgactatct ggaaaccctc gaagaggtga tcctcaagcc gcgtggcctg 300 gattacaagg ttcatctgcc gggcccgacg ggcaccaatg ccgtggaagc cgccattcga 360 ctggcgcgca acgccaaggg tcgtcacaac atcgtcacct tcaccaacgg attccatggc 420 gtgaccatgg gcgcgctggc caccaccggc aatcgcaagt tccgtgaagc caccggcggt 480 atcccgaccc agggcgccag cttcatgccg ttcgatggct acatgggcga gggcgtcgac 540 accctgagct acttcgagaa actgctcggc gacaactccg gtggtctcga cgttcccgcg 600 gccgtgatca tcgagacggt gcagggcgag ggcggtatca atccggccgg catcccgtgg 660 ctgcagcgcc tggaaaagat ctgccgcgat cacgacatgc tgctgatcgt cgacgacatt 720 caggccggct gcggtcgtac gggcaagttc ttcagcttcg agcatgccgg catcacgccg 780 gacatcgtca ccaactccaa gtccctgtcg ggtttcggcc tgccgttcgc gcatgtgctg 840 atgcgcccgg aactggatat ctggaagccc ggccagtaca acggcacgtt ccgtggtttc 900 aacctggcct tcgtcacggc cgccgccgcg atgcgtcact tctggagcga cgacaccttc 960 gagcgcgacg ttcagcgcaa gggccgtgtg gtcgaggatc gcttccagaa gcttgccagc 1020 ttcatgaccg agaaagggca tccggccagc gagcgtggcc gtggcctgat gcgtggcctg 1080 gacgtcggtg acggcgacat ggccgacaag atcaccgcac aagcgttcaa gaacgggctg 1140 atcatcgaga catccggcca ttcaggccag gtgatcaagt gcctttgccc gttgaccatt 1200 accgacgaag acctcgtcgg cggcctggac atcctcgagc agagcgtcaa ggaagtcttc 1260 ggtcaagcct aa 1272 lt;210gt; lt;211gt; 3 393 lt;212gt; DNA lt;213gt; chromohalobacter salexigens lt;220gt; lt;223gt; Secuencia codificante del gen ectC para la enzima ectoina sintasa lt;400gt; 3 atgatcgttc gtaacctgga agaatgccgc aagaccgagc gcttcgtcga agccgaaaac 60 ggcaactggg acagcacccg tctggtgctg gccgacgaca acgtcggttt ctcgttcaac 120 atcacccgca ttcatccggg taccgagacg catatccatt acaagcatca cttcgaggcg 180 gttttctgct acgaaggcga aggcgaagtc gaaacgctgg ccgatggcaa gatccatccc 240 atcaaggccg gcgacatgta cttgctcgat cagcacgacg agcacctgct gcgcggcaag 300 gaaaaaggca tgaccgtggc atgcgtgttc aatccggcgc tgacgggccg cgaagtgcac 360 cgtgaagacg gttcctacgc accggtcgat tga 393 lt;210gt; 4 lt;211gt; 945 lt;212gt; DNA lt;213gt; Chromohalobacter salexigens lt;220gt; lt;223gt; secuencia codificante del gen ectD para la enzima ectoina hidroxilasa lt;400gt; 4 atgtcagtgc aaagcgcatt tcgacatgaa gacgtgacat tcggcaacgc gttcgatgcg 60 taccccaccc gcctgagcga tgccactggt tcgctttggc aagaccgcaa ggatgccgtg 120 gtcaaggggc gcgccgagga tggaccgctg agtcgcgacc agctggcgcg tttcgagcgc 180 gacggctttt tgttcgaacc ccattttctg gccgacgacg aggtcggtga attgcgccgc 240 gagcttgccg cgctgctgga gcgcgacgat ttccgcggcc gcgatttcag catcaccgag 300 cccgacagcc aggagatccg gtcgctgttt gccgtgcact ttctctccga gaggttccgg 360 cgcctggccg aggatccgcg cctgaccggg cgggcccgcc agatcgtggg cggtgacgtc 420 tacgtgcacc agtcgcggat caactacaag cccggtttcc atggcaaggg gttcaactgg 480 cactcggact tcgagacctg gcacgccgaa gacggcatgc cggagatgca tgcggtcagt 540 gcctccatcg tgttgaccga caatcatcac tacaacggcc cgctgatgct gatccccggg 600 tcgcacaagg tattcgtgca ttgcctcggc gagacgccgg aggatcatca caagcagtcg 660 ctgaagagcc aggagttcgg cgtgcccagc cacgaggcac tgcgacgctt gatcggcgcc 720 aacggcatcg aggccccgac gggggcggcc ggcgggctac tgctgttcga ctgcaatacg 780 ctgcatggct ccaatgccaa catgtcgccc gatccgcgca gcaatgcctt cttcgtctac 840 aaccggcgcg acaacgcctg tcacgcgcca tacggggcca agcggccgcg gccgtccttc 900 ctggcgcacg cgccggatga ggtctggaca ccggacacgc attga 945 lt;210gt; 5 lt;211gt; 233 lt;212gt; DNA lt;213gt; Chromohalobacter salexigens lt;220gt; lt;223gt; Secuencia nucleotidica promotora del gen ectA lt;400gt; 5 tgctcctgac gcatgcgtca cacgaacggg ccacccctgg gtggcccgtt tgcgttgcag 60 gaaccactca cgtcatgcag gcgtcacatc attgactaga ctgtaggaag tgccgtacct 120 gtccattcca gctataatat gaggttatct gatagcgaca cgtcgataac gatccttttg 180 ataaatacat aagccgctgt tattattgag aacacattcg ccattagcga aca 233 lt;210gt; 6 lt;211gt; 2654 lt;212gt; DNA lt;213gt; Secuencia Artificial lt;220gt; lt;223gt; Secuencia del plasmido pSK Bluescript que contiene el operon eCtABC completo lt;400gt; 6 gaattcgtca ccatgcggtc gctgtcacgg cactgatgct cctgacgcat gcgtcacacg 60 aacgggccac ccctgggtgg cccgtttgcg ttgcaggaac cactcacgtc atgcaggcgt 120 cacatcattg actagactgt aggaagtgcc gtacctgtcc attccagcta taatatgagg 180 ttatctgata gcgacacgtc gataacgatc cttttgataa atacataagc cgctgttatt 240 attgagaaca cattcgccat tagcgaacaa tggatatgac gcctacaacc gagaacttca 300 cgccttccgc cgatctggcc cgcccgagcg ttgccgacac ggtcatcggc agcgcgaaga 360 aaaccctctt catccgtaag cccacgaccg acgacgggtg gggcatttac gaactcgtca 420 aggcatgccc gccactggac gtcaactcgg ggtacgccta tctgctgctt gccacccagt 480 tccgcgatac ctgcgccgtg gctacggacg aagaagggga gatcgtcggt ttcgtctccg 540 gctacgtcaa acgcaacgca ccggacacct actttctatg gcaagtcgcc gtcggcgaga 600 aagcgcgggg cacgggcctg gcgcgacgcc tcgtcgaagc cgtgctgatg cgccccggca 660 tgggcgatgt ccgccatctc gaaactacca tcacgcccga caatgaggca tcgtggggac 720 tgttcaagcg ccttgccgat cgctggcaag cccccctgaa cagccgtgaa tacttctcca 780 cggggcagtt gggtggcgag cacgatccgg aaaatctcgt ccgtatcggc ccgttcgagc 840 cgcaacaaat ttgatccgtc aatcagaggc aatctatgca gacccagatt ctcgaacgca 900 tggaatccga agtccggacc tattcacgtt cttttcctac cgttttcact gaagccaagg 960 gcgcgcgcct gcatgccgag gacggcaacc agtacatcga ttttctcgcc ggcgccggca 1020 cgctcaacta cggtcacaac caccccaagc tcaagcaggc actggccgat tacatcgcct 1080 ccgatggcat cgtccatggt ctggacatgt ggagcgcggc caagcgcgac tatctggaaa 1140 ccctcgaaga ggtgatcctc aagccgcgtg gcctggatta caaggttcat ctgccgggcc 1200 cgacgggcac caatgccgtg gaagccgcca ttcgactggc gcgcaacgcc aagggtcgtc 1260 acaacatcgt caccttcacc aacggattcc atggcgtgac catgggcgcg ctggccacca 1320 ccggcaatcg caagttccgt gaagccaccg gcggtatccc gacccagggc gccagcttca 1380 tgccgttcga tggctacatg ggcgagggcg tcgacaccct gagctacttc gagaaactgc 1440 tcggcgacaa ctccggtggt ctcgacgttc ccgcggccgt gatcatcgag acggtgcagg 1500 gcgagggcgg tatcaatccg gccggcatcc cgtggctgca gcgcctggaa aagatctgcc 1560 gcgatcacga catgctgctg atcgtcgacg acattcaggc cggctgcggt cgtacgggca 1620 agttcttcag cttcgagcat gccggcatca cgccggacat cgtcaccaac tccaagtccc 1680 tgtcgggttt cggcctgccg ttcgcgcatg tgctgatgcg cccggaactg gatatctgga 1740 agcccggcca gtacaacggc acgttccgtg gtttcaacct ggccttcgtc acggccgccg 1800 ccgcgatgcg tcacttctgg agcgacgaca ccttcgagcg cgacgttcag cgcaagggcc 1860 gtgtggtcga ggatcgcttc cagaagcttg ccagcttcat gaccgagaaa gggcatccgg 1920 ccagcgagcg tggccgtggc ctgatgcgtg gcctggacgt cggtgacggc gacatggccg 1980 acaagatcac cgcacaagcg ttcaagaacg ggctgatcat cgagacatcc ggccattcag 2040 gccaggtgat caagtgcctt tgcccgttga ccattaccga cgaagacctc gtcggcggcc 2100 tggacatcct cgagcagagc gtcaaggaag tcttcggtca agcctaagtc cattgttcgt 2160 tagtccacta aattgtcatt cgcaaatgtg tttacagtgg gcaccgcctg cggccacgag 2220 gtcgcgggct tcttactcag atctgcagag gattgcgcca catgatcgtt cgtaacctgg 2280 aagaatgccg caagaccgag cgcttcgtcg aagccgaaaa cggcaactgg gacagcaccc 2340 gtctggtgct ggccgacgac aacgtcggtt tctcgttcaa catcacccgc attcatccgg 2400 gtaccgagac gcatatccat tacaagcatc acttcgaggc ggttttctgc tacgaaggcg 2460 aaggcgaagt cgaaacgctg gccgatggca agatccatcc catcaaggcc ggcgacatgt 2520 acttgctcga tcagcacgac gagcacctgc tgcgcggcaa ggaaaaaggc atgaccgtgg 2580 catgcgtgtt caatccggcg ctgacgggcc gcgaagtgca ccgtgaagac ggttcctacg 2640 caccggtcga ttga 2654 lt;210gt; 7 lt;211gt; 194 lt;212gt; PRT lt;213gt; Chromohalobacter salexigens lt;220gt; lt;223gt; Secuencia de la enzima diaminobutirato acetiltransferasa lt;400gt; 7 met Asp Met Thr Pro Thr Thr Glu Asn Phe Thr Pro Ser Ala Asp Leu 1 5 10 15 Ala Arg Pro Ser Val Ala Asp Thr Val Ile Gly Ser Ala Lys Lys Thr 20 25 30 Leu Phe Ile Arg Lys Pro Thr Thr Asp Asp Gly Trp Gly Ile Tyr Glu 35 40 45 Leu Val Lys Ala Cys Pro Pro Leu Asp Val Asn Ser Gly Tyr Ala Tyr 50 55 60 Leu Leu Leu Ala Thr Gin Phe Arg Asp Thr cys Ala Val Ala Thr Asp 65 70 75 80 Glu Glu Gly Glu Ile val Gly Phe Val Ser Gly Tyr Val Lys Arg Asn 85 90 95 Ala Pro Asp Thr Tyr Phe Leu Trp Gin Val Ala Val Gly Glu Lys Ala 100 105 110 Arg Gly Thr Gly Leu Ala Arg Arg Leu Val Glu Ala Val Leu Met Arg 115 120 125 Pro Gly Met Gly Asp val Arg His Leu Glu Thr Thr Ile Thr Pro Asp 130 135 140 Asn Glu Ala Ser Trp Gly Leu Phe Lys Arg Leu Ala Asp Arg Trp Gin 145 150 155 160 Ala Pro Leu Asn Ser Arg Glu Tyr Phe Ser Thr Gly Gln Leu Gly Gly 165 170 175 Glu His Asp Pro Glu Asn Leu Val Arg Ile Gly Pro Phe Glu Pro Gin 180 185 190 Gin Ile lt;210gt; 8 lt;211gt; 423 lt;212gt; PRT lt;213gt; Chromohalobacter salexigens lt;220gt; lt;223gt; Secuencia de la enzima diaminobutirato transaminasa lt;400gt; 8 Met Gin Thr Gin Ile Leu Glu Arg Met Glu Ser Glu Val Arg Thr Tyr 1 5 10 15 Ser Arg Ser Phe Pro Thr Val Phe Thr Glu Ala Lys Gly Ala Arg Leu 20 25 30 . His Ala Glu Asp Gly Asn Gin Tyr Ile Asp Phe Leu Ala Gly Ala Gly 35 40 45 Thr Leu Asn Tyr Gly His Asn His Pro Lys Leu Lys Gin Ala Leu Ala 50 55 60 Asp Tyr Ile Ala Ser Asp Gly Ile Val His Gly Leu Asp Met Trp Ser 65 70 75 80 Ala Ala Lys Arg Asp Tyr Leu Glu Thr Leu Glu Glu Val Ile Leu Lys 85 90 95 Pro Arg Gly Leu Asp Tyr Lys Val His Leu Pro Gly Pro Thr Gly Thr 100 105 110 Asn Ala Val Glu Ala Ala Ile Arg Leu Ala Arg Asn Ala Lys Gly Arg 115 120 125 His Asn Ile Val Thr Phe Thr Asn Gly Phe His Gly Val Thr Met Gly 130 135 140 Ala Leu Ala Thr Thr Gly Asn Arg Lys Phe Arg Glu Ala Thr Gly Gly 145 150 155 160 Ile Pro Thr Gin Gly Ala Ser Phe Met Pro Phe Asp Gly Tyr Met Gly 165 170 175 Glu Gly Val Asp Thr Leu Ser Tyr Phe Glu Lys Leu Leu Gly Asp Asn 180 185 190 Ser Gly Gly Leu Asp Val Pro Ala Ala Val Ile Ile Glu Thr Val Gin 195 200 205 Gly Glu Gly Gly Ile Asn Pro Ala Gly Ile Pro Trp Leu Gin Arg Leu 210 215 220 Glu Lys Ile cys Arg Asp His Asp met Leu Leu Ile Val Asp Asp Ile 225 230 235 240 Gin Ala Gly Cys Gly Arg Thr Gly Lys Phe Phe Ser Phe Glu His Ala 245 250 255 Gly Ile Thr Pro Asp Ile Val Thr Asn ser Lys Ser Leu Ser Gly Phe 260 265 270 Gly Leu Pro Phe Ala His Val Leu Met Arg Pro Glu Leu Asp Ile Trp 275 280 285 Lys Pro Gly Gin Tyr Asn Gly Thr Phe Arg Gly Phe Asn Leu Ala Phe 290 295 300 Val Thr Ala Ala Ala Ala Met Arg His Phe Trp Ser Asp Asp Thr Phe 305 310 315 320 Glu Arg Asp Val Gin Arg Lys Gly Arg Val Val Glu Asp Arg Phe Gin 325 330 335 Lys Leu Ala Ser Phe Met Thr Glu Lys Gly His Pro Ala Ser Glu Arg 340 345 350 Gly Arg Gly Leu Met Arg Gly Leu Asp Val Gly Asp Gly Asp Met Ala 355 360 365 Asp Lys Ile Thr Ala Gin Ala Phe Lys Asn Gly Leu Ile Ile Glu Thr 370 375 380 Ser Gly His Ser Gly Gin Val Ile Lys cys Leu cys Pro Leu Thr Ile 385 390 395 400 Thr Asp Glu Asp Leu Val Gly Gly Leu Asp Ile Leu Glu Gin Ser Val 405 410 415 Lys Glu Val Phe Gly Gin Ala 420 lt;210gt; 9 lt;211gt; 130 lt;212gt; PRT lt;213gt; Chromohalobacter salexigens lt;220gt; lt;223gt; Secuencia de la enzima ectoina sintasa lt;400gt; 9 Met Ile Val Arg Asn Leu Glu Glu Cys Arg Lys Thr Glu Arg Phe Val 1 5 10 15 Glu Ala Glu Asn Gly Asn Trp Asp Ser Thr Arg Leu Val Leu Ala Asp 20 25 30 Asp Asn Val Gly Phe Ser Phe Asn Ile Thr Arg Ile His Pro Gly Thr 35 40 45 Glu Thr His Ile His Tyr Lys His His Phe Glu Ala Val Phe Cys Tyr 50 55 60 Glu Gly Glu Gly Glu Val Glu Thr Leu Ala Asp Gly Lys Ile His Pro 65 70 75 80 Ile Lys Ala Gly Asp Met Tyr Leu Leu Asp Gin His Asp Glu His Leu 85 90 95 Leu Arg Gly Lys Glu Lys Gly Met Thr Val Ala Cys Val Phe Asn Pro 100 105 110 Ala Leu Thr Gly Arg Glu Val His Arg Glu Asp Gly Ser Tyr Ala Pro 115 120 125 Val Asp 130 lt;210gt; 10 lt;211gt; 314 lt;212gt; PRT lt;213gt; Chromohalobacter salexigens lt;220gt; lt;223gt; Secuencia de la enzima ectoina hidroxilasa lt;400gt; 10 Met Ser Val Gin Ser Ala Phe Arg His Glu Asp Val Thr Phe Gly Asn 1 5 10 15 Ala Phe Asp Ala Tyr Pro Thr Arg Leu Ser Asp Ala Thr Gly ser Leu 20 25 30 Trp Gin Asp Arg Lys Asp Ala Val Val Lys Gly Arg Ala Glu Asp Gly 35 40 45 Pro Leu Ser Arg Asp Gin Leu Ala Arg Phe Glu Arg Asp Gly Phe Leu 50 55 60 Phe Glu Pro His Phe Leu Ala Asp Asp Glu Val Gly Glu Leu Arg Arg 65 70 75 80 Glu Leu Ala Ala Leu Leu Glu Arg Asp Asp Phe Arg Gly Arg Asp Phe 85 90 95 Ser Ile Thr Glu Pro Asp Ser Gin Glu Ile Arg Ser Leu Phe Ala Val 100 105 110 His Phe Leu Ser Glu Arg Phe Arg Arg Leu Ala Glu Asp Pro Arg Leu 115 120 125 Thr Gly Arg Ala Arg Gin Ile Val Gly Gly Asp Val Tyr Val His Gin 130 135 140 Ser Arg Ile Asn Tyr Lys Pro Gly Phe His Gly Lys Gly Phe Asn Trp 145 150 155 160 His Ser Asp Phe Glu Thr Trp His Ala Glu Asp Gly Met Pro Glu met 165 170 175 His Ala Val Ser Ala Ser Ile Val Leu Thr Asp Asn His His Tyr Asn 180 185 190 Gly Pro Leu Met Leu Ile Pro Gly Ser His Lys Val Phe Val His Cys 195 200 205 Leu Gly Glu Thr Pro Glu Asp His His Lys Gin Ser Leu Lys Ser Gin 210 215 220 Glu Phe Gly Val Pro Ser His Glu Ala Leu Arg Arg Leu Ile Gly Ala 225 230 235 240 Asn Gly Ile Glu Ala Pro Thr Gly Ala Ala Gly Gly Leu Leu Leu Phe 245 250 255 Asp Cys Asn Thr Leu His Gly Ser Asn Ala Asn Met Ser Pro Asp Pro 260 265 270 Arg Ser Asn Ala Phe Phe Val Tyr Asn Arg Arg Asp Asn Ala Cys His 275 280 285 Ala Pro Tyr Gly Ala Lys Arg Pro Arg Pro Ser Phe Leu Ala His Ala 290 295 300 Pro Asp Glu Val Trp Thr Pro Asp Thr His 305 310 lt;210gt; 11 lt;211gt; 3780 lt;212gt; DNA lt;213gt; secuencia Artificial lt;220gt; lt;223gt; ectABCD lt;400gt; 11 gaattcgtca ccatgcggtc gctgtcacgg cactgatgct cctgacgcat gcgtcacacg aacgggccac ccctgggtgg cccgtttgcg ttgcaggaac cactcacgtc atgcaggcgt 120 cacatcattg actagactgt aggaagtgcc gtacctgtcc attccagcta taatatgagg 180 ttatctgata gcgacacgtc gataacgatc cttttgataa atacataagc cgctgttatt 240 attgagaaca cattcgccat tagcgaacaa tggatatgac gcctacaacc gagaacttca 300 cgccttccgc cgatctggcc cgcccgagcg ttgccgacac ggtcatcggc agcgcgaaga 360 aaaccctctt catccgtaag cccacgaccg acgacgggtg gggcatttac gaactcgtca 420 aggcatgccc gccactggac gtcaactcgg ggtacgccta tctgctgctt gccacccagt 480 tccgcgatac ctgcgccgtg gctacggacg aagaagggga gatcgtcggt ttcgtctccg 540 gctacgtcaa acgcaacgca ccggacacct actttctatg gcaagtcgcc gtcggcgaga 600 aagcgcgggg cacgggcctg gcgcgacgcc tcgtcgaagc cgtgctgatg cgccccggca 660 tgggcgatgt ccgccatctc gaaactacca tcacgcccga caatgaggca tcgtggggac 720 tgttcaagcg ccttgccgat cgctggcaag cccccctgaa cagccgtgaa tacttctcca 780 cggggcagtt gggtggcgag cacgatccgg aaaatctcgt ccgtatcggc ccgttcgagc 840 cgcaacaaat ttgatccgtc aatcagaggc aatctatgca gacccagatt ctcgaacgca 900 tggaatccga agtccggacc tattcacgtt cttttcctac cgttttcact gaagccaagg 960 gcgcgcgcct gcatgccgag gacggcaacc agtacatcga ttttctcgcc ggcgccggca 1020 cgctcaacta cggtcacaac caccccaagc tcaagcaggc actggccgat tacatcgcct 1080 ccgatggcat cgtccatggt ctggacatgt ggagcgcggc caagcgcgac tatctggaaa 1140 ccctcgaaga ggtgatcctc aagccgcgtg gcctggatta caaggttcat ctgccgggcc 1200 cgacgggcac caatgccgtg gaagccgcca ttcgactggc gcgcaacgcc aagggtcgtc 1260 acaacatcgt caccttcacc aacggattcc atggcgtgac catgggcgcg ctggccacca 1320 ccggcaatcg caagttccgt gaagccaccg gcggtatccc gacccagggc gccagcttca 1380 tgccgttcga tggctacatg ggcgagggcg tcgacaccct gagctacttc gagaaactgc 1440 tcggcgacaa ctccggtggt ctcgacgttc ccgcggccgt gatcatcgag acggtgcagg 1500 gcgagggcgg tatcaatccg gccggcatcc cgtggctgca gcgcctggaa aagatctgcc 1560 gcgatcacga catgctgctg atcgtcgacg acattcaggc cggctgcggt cgtacgggca 1620 agttcttcag cttcgagcat gccggcatca cgccggacat cgtcaccaac tccaagtccc 1680 tgtcgggttt cggcctgccg ttcgcgcatg tgctgatgcg cccggaactg gatatctgga 1740 agcccggcca gtacaacggc acgttccgtg gtttcaacct ggccttcgtc acggccgccg 1800 ccgcgatgcg tcacttctgg agcgacgaca ccttcgagcg cgacgttcag cgcaagggcc 1860 gtgtggtcga ggatcgcttc cagaagcttg ccagcttcat gaccgagaaa gggcatccgg 1920 ccagcgagcg tggccgtggc ctgatgcgtg gcctggacgt cggtgacggc gacatggccg 1980 acaagatcac cgcacaagcg ttcaagaacg ggctgatcat cgagacatcc ggccattcag 2040 gccaggtgat caagtgcctt tgcccgttga ccattaccga cgaagacctc gtcggcggcc 2100 tggacatcct cgagcagagc gtcaaggaag tcttcggtca agcctaagtc cattgttcgt 2160 tagtccacta aattgtcatt cgcaaatgtg tttacagtgg gcaccgcctg cggccacgag 2220 gtcgcgggct tcttactcag atctgcagag gattgcgcca catgatcgtt cgtaacctgg 2280 aagaatgccg caagaccgag cgcttcgtcg aagccgaaaa cggcaactgg gacagcaccc 2340 gtctggtgct ggccgacgac aacgtcggtt tctcgttcaa catcacccgc attcatccgg 2400 gtaccgagac gcatatccat tacaagcatc acttcgaggc ggttttctgc tacgaaggcg 2460 aaggcgaagt cgaaacgctg gccgatggca agatccatcc catcaaggcc ggcgacatgt 2520 acttgctcga tcagcacgac gagcacctgc tgcgcggcaa ggaaaaaggc atgaccgtgg 2580 catgcgtgtt caatccggcg ctgacgggcc gcgaagtgca ccgtgaagac ggttcctacg 2640 caccggtcga ttgaggatca caaacccagg aggtgattgg atgtcagtgc aaagcgcatt 2700 tcgacatgaa gacgtgacat tcggcaacgc gttcgatgcg taccccaccc gcctgagcga 2760 tgccactggt tcgctttggc aagaccgcaa ggatgccgtg gtcaaggggc gcgccgagga 2820 tggaccgctg agtcgcgacc agctggcgcg tttcgagcgc gacggctttt tgttcgaacc 2880 ccattttctg gccgacgacg aggtcggtga attgcgccgc gagcttgccg cgctgctgga 2940 gcgcgacgat ttccgcggcc gcgatttcag catcaccgag cccgacagcc aggagatccg 3000 gtcgctgttt gccgtgcact ttctctccga gaggttccgg cgcctggccg aggatccgcg 3060 cctgaccggg cgggcccgcc agatcgtggg cggtgacgtc tacgtgcacc agtcgcggat 3120 caactacaag cccggtttcc atggcaaggg gttcaactgg cactcggact tcgagacctg 3180 gcacgccgaa gacggcatgc cggagatgca tgcggtcagt gcctccatcg tgttgaccga 3240 caatcatcac tacaacggcc cgctgatgct gatccccggg tcgcacaagg tattcgtgca 3300 ttgcctcggc gagacgccgg aggatcatca caagcagtcg ctgaagagcc aggagttcgg 3360 cgtgcccagc cacgaggcac tgcgacgctt gatcggcgcc aacggcatcg aggccccgac 3420 gggggcggcc ggcgggctac tgctgttcga ctgcaatacg ctgcatggct ccaatgccaa 3480 catgtcgccc gatccgcgca gcaatgcctt cttcgtctac aaccggcgcg acaacgcctg 3540 tcacgcgcca tacggggcca agcggccgcg gccgtccttc ctggcgcacg cgccggatga 3600 ggtctggaca ccggacacgc attgacgcca tgccatgatc cagttgaact gtacccgaga 3660 aagcgctgcc gaatggcagc gctttctttt gaggtttcgt gcgcgtcggg atcgtcagac 3720 atgtcccgca cgagtgtggg tagctagcaa taaattgtgg gccgaaatgt gggcgaattc 3780 lt;210gt; 12 lt;211gt; 4761 lt;212gt; DNA lt;213gt; Secuencia Artificial lt;220gt; lt;223gt; ectABCDD lt;400gt; 12 gaattcgtca ccatgcggtc gctgtcacgg cactgatgct cctgacgcat gcgtcacacg 60 aacgggccac ccctgggtgg cccgtttgcg ttgcaggaac cactcacgtc atgcaggcgt 120 cacatcattg actagactgt aggaagtgcc gtacctgtcc attccagcta taatatgagg 180 ttatctgata gcgacacgtc gataacgatc cttttgataa atacataagc cgctgttatt 240 attgagaaca cattcgccat tagcgaacaa tggatatgac gcctacaacc gagaacttca 300 cgccttccgc cgatctggcc cgcccgagcg ttgccgacac ggtcatcggc agcgcgaaga 360 aaaccctctt catccgtaag cccacgaccg acgacgggtg gggcatttac gaactcgtca 420 aggcatgccc gccactggac gtcaactcgg ggtacgccta tctgctgctt gccacccagt 480 tccgcgatac ctgcgccgtg gctacggacg aagaagggga gatcgtcggt ttcgtctccg 540 gctacgtcaa acgcaacgca ccggacacct actttctatg gcaagtcgcc gtcggcgaga 600 aagcgcgggg cacgggcctg gcgcgacgcc tcgtcgaagc cgtgctgatg cgccccggca 660 tgggcgatgt ccgccatctc gaaactacca tcacgcccga caatgaggca tcgtggggac 720 tgttcaagcg ccttgccgat cgctggcaag cccccctgaa cagccgtgaa tacttctcca 780 cggggcagtt gggtggcgag cacgatccgg aaaatctcgt ccgtatcggc ccgttcgagc 840 cgcaacaaat ttgatccgtc aatcagaggc aatctatgca gacccagatt ctcgaacgca 900 tggaatccga agtccggacc tattcacgtt cttttcctac cgttttcact gaagccaagg 960 gcgcgcgcct gcatgccgag gacggcaacc agtacatcga ttttctcgcc ggcgccggca 1020 cgctcaacta cggtcacaac caccccaagc tcaagcaggc actggccgat tacatcgcct 1080 ccgatggcat cgtccatggt ctggacatgt ggagcgcggc caagcgcgac tatctggaaa 1140 ccctcgaaga ggtgatcctc aagccgcgtg gcctggatta caaggttcat ctgccgggcc 1200 cgacgggcac caatgccgtg gaagccgcca ttcgactggc gcgcaacgcc aagggtcgtc 1260 acaacatcgt caccttcacc aacggattcc atggcgtgac catgggcgcg ctggccacca 1320 ccggcaatcg caagttccgt gaagccaccg gcggtatccc gacccagggc gccagcttca 1380 tgccgttcga tggctacatg ggcgagggcg tcgacaccct gagctacttc gagaaactgc 1440 tcggcgacaa ctccggtggt ctcgacgttc ccgcggccgt gatcatcgag acggtgcagg 1500 gcgagggcgg tatcaatccg gccggcatcc cgtggctgca gcgcctggaa aagatctgcc 1560 gcgatcacga catgctgctg atcgtcgacg acattcaggc cggctgcggt cgtacgggca 1620 agttcttcag cttcgagcat gccggcatca cgccggacat cgtcaccaac tccaagtccc 1680 tgtcgggttt cggcctgccg ttcgcgcatg tgctgatgcg cccggaactg gatatctgga 1740 agcccggcca gtacaacggc acgttccgtg gtttcaacct ggccttcgtc acggccgccg 1800 ccgcgatgcg tcacttctgg agcgacgaca ccttcgagcg cgacgttcag cgcaagggcc 1860 gtgtggtcga ggatcgcttc cagaagcttg ccagcttcat gaccgagaaa gggcatccgg 1920 ccagcgagcg tggccgtggc ctgatgcgtg gcctggacgt cggtgacggc gacatggccg 1980 acaagatcac cgcacaagcg ttcaagaacg ggctgatcat cgagacatcc ggccattcag 2040 gccaggtgat caagtgcctt tgcccgttga ccattaccga cgaagacctc gtcggcggcc 2100 tggacatcct cgagcagagc gtcaaggaag tcttcggtca agcctaagtc cattgttcgt 2160 tagtccacta aattgtcatt cgcaaatgtg tttacagtgg gcaccgcctg cggccacgag 2220 gtcgcgggct tcttactcag atctgcagag gattgcgcCa catgatcgtt cgtaacctgg 2280 aagaatgccg caagaccgag cgcttcgtcg aagccgaaaa cggcaactgg gacagcaccc 2340 gtctggtgct ggccgacgac aacgtcggtt tctcgttcaa catcacccgc attcatccgg 2400 gtaccgagac gcatatccat tacaagcatc acttcgaggc ggttttctgc tacgaaggcg 2460 aaggcgaagt cgaaacgctg gccgatggca agatccatcc catcaaggcc ggcgacatgt 2520 acttgctcga tcagcacgac gagcacctgc tgcgcggcaa ggaaaaaggc atgaccgtgg 2580 catgcgtgtt caatccggcg ctgacgggcc gcgaagtgca ccgtgaagac ggttcctacg 2640 caccggtcga ttgaggatca caaacccagg aggtgattgg atgtcagtgc aaagcgcatt 2700 tcgacatgaa gacgtgacat tcggcaacgc gttcgatgcg taccccaccc gcctgagcga 2760 tgccactggt tcgctttggc aagaccgcaa ggatgccgtg gtcaaggggc gcgccgagga 2820 tggaccgctg agtcgcgacc agctggcgcg tttcgagcgc gacggctttt tgttcgaacc 2880 ccattttctg gccgacgacg aggtcggtga attgcgccgc gagcttgccg cgctgctgga 2940 gcgcgacgat ttccgcggcc gcgatttcag catcaccgag cccgacagcc aggagatccg 3000 gtcgctgttt gccgtgcact ttctctccga gaggttccgg cgcctggccg aggatccgcg 3060 cctgaccggg cgggcccgcc agatcgtggg cggtgacgtc tacgtgcacc agtcgcggat 3120 caactacaag cccggtttcc atggcaaggg gttcaactgg cactcggact tcgagacctg 3180 gcacgccgaa gacggcatgc cggagatgca tgcggtcagt gcctccatcg tgttgaccga 3240 caatcatcac tacaacggcc cgctgatgCt gatccccggg tcgcacaagg tattcgtgca 3300 ttgcctcggc gagacgccgg aggatcatca caagcagtcg ctgaagagcc aggagttcgg 3360 cgtgcccagc cacgaggcac tgcgacgctt gatcggcgcc aacggcatcg aggccccgac 3420 gggggcggcc ggcgggctac tgctgttcga ctgcaatacg ctgcatggct ccaatgccaa 3480 catgtcgccc gatccgcgca gcaatgcctt cttcgtctac aaccggcgcg acaacgcctg 3540 tcacgcgcca tacggggcca agcggccgcg gccgtccttc ctggcgcacg cgccggatga 3600 ggtctggaca ccggacacgc attgacgcca tgccatgatc ccaaacccag gaggtgattg 3660 gatgtcagtg caaagcgcat ttcgacatga agacgtgaca ttcggcaacg cgttcgatgc 3720 gtaccccacc cgcctgagcg atgccactgg ttcgctttgg caagaccgca aggatgccgt 3780 ggtcaagggg cgcgccgagg atggaccgct gagtcgcgac cagctggcgc gtttcgagcg 3840 cgacggcttt ttgttcgaac cccattttct ggccgacgac gaggtcggtg aattgcgccg 3900 cgagcttgcc gcgctgctgg agcgcgacga tttccgcggc cgcgatttca gcatcaccga 3960 gcccgacagc caggagatcc ggtcgctgtt tgccgtgcac tttctctccg agaggttccg 4020 gcgcctggcc gaagatccgc gcctgaccgg gcgggcccgc cagatcgtgg gcggtgacgt 4080 ctacgtgcac cagtcgcgga tcaactacaa gcccggtttc catggcaagg ggttcaactg 4140 gcactcggac ttcgagacct ggcacgccga agacggcatg ccggagatgc atgcggtcag 4200 tgcctccatc gtgttgaccg acaatcatca ctacaacggc ccgctgatgc tgatccccgg 4260 gtcgcacaag gtattcgtgc attgcctcgg cgagacgccg gaggatcatc acaagcagtc 4320 gctgaagagc caggagttcg gcgtgcccag ccacgaggca ctgcgacgct tgatcggcgc 4380 caacggcatc gaggccccga cgggggcggc cggcgggcta ctgctgttcg actgcaatac 4440 gctgcatggc tccaatgcca acatgtcgcc cgatccgcgc agcaatgcct tcttcgtcta 4500 caaccggcgc gacaacgcct gtcacgcgcc atacggggcc aagcggccgc ggccgtcctt 4560 cctggcgcac gcgccggatg aggtctggac accggacacg cattgacgcc atgccatgat 4620 ccagttgaac tgtacccgag aaagcgctgc cgaatggcag cgctttcttt tgaggtttcg 4680 tgcgcgtcgg gatcgtcaga catgtcccgc acgagtgtgg gtagctagca ataaattgtg 4740 ggccgaaatg tgggcgaatt c 4761
Patentes similares o relacionadas:
Cadena ligera de enteroquinasa modificada, del 22 de Julio de 2020, de NOVO NORDISK A/S: Un análogo de la cadena ligera de la enteroquinasa bovina que comprende una secuencia de aminoácidos establecida en la SEQ ID NO: 1, en donde dicho análogo comprende […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos y composiciones para ingeniería genómica, del 3 de Junio de 2020, de Sangamo Therapeutics, Inc: Una pareja de nucleasas de dedo de zinc (ZFN) que comprende una ZFN izquierda y una ZFN derecha, comprendiendo cada ZFN un dominio de escisión […]
Métodos y composiciones para escisión dirigida y recombinación, del 20 de Mayo de 2020, de Sangamo Therapeutics, Inc: Un método in vitro para la escisión selectiva de un gen HLA clase I, un gen HLA que codifica una proteína de clase 1 del Complejo de Histocompatibilidad Mayor (MHC) […]
Antígenos de coagulasa estafilocócica y métodos para su uso, del 13 de Mayo de 2020, de UNIVERSITY OF CHICAGO: Una composición inmunógena que comprende al menos dos dominios 1-2 de coagulasa estafilocócica diferentes, en donde cada uno de los al menos dos dominios […]
Reconocimiento de unión a diana celular mediante un agente bioactivo usando transferencia de energía de resonancia de bioluminiscencia intracelular, del 6 de Mayo de 2020, de PROMEGA CORPORATION: Un sistema de ensayo que comprende: (a) una biblioteca de agentes bioactivos, cada uno de los cuales está fijado a un fluoróforo; (b) una diana celular fusionada a […]
Etiqueta de epítopo y método de detección, captura y/o purificación de polipéptidos etiquetados, del 15 de Abril de 2020, de ChromoTek GmbH: Péptido epítopo aislado que tiene de 12 a 25 aminoácidos, en donde la secuencia de aminoácidos comprende una secuencia según se define en SEQ ID NO: 32 (X1X2RX4X5AX7SX9WX11X12), […]
Utilización diagnóstica de un polipéptido de fusión que comprende una proteína vírica y un enzima MGMT, del 15 de Abril de 2020, de INSTITUT PASTEUR: Utilización in vitro de un polipéptido de fusión que comprende una proteína vírica y i) el enzima 6-metilguanina-ADN-metiltransferasa (MGMT, EC 2.1.1.63) o un homólogo […]