METODO PARA LA MEDICION CUANTITATIVA Y/O COMPARATIVA DE NIVELES DE EXPRESION DE MRNA EN MUESTRAS BIOLOGICAS PEQUEÑAS.
Un método para la valoración cuantitativa y/o comparativa de las cantidades relativas de transcritos de mRNA presentes en una muestra celular o tisular,
caracterizado por las operaciones de:
(a) llevar a cabo la transcripción inversa del mRNA contenido en la muestra utilizando cebadores modificadores de secuencia para diversos genes en la misma reacción, para obtener un conjunto de moléculas de cDNA secuencialmente modificadas, en que los cebadores modificadores de secuencia son oligonucleótidos que comprenden tres segmentos funcionales:
(i) un segmento 5''-terminal que comprende una secuencia nucleotídica, que es complementaria de la secuencia de mRNA así como de la secuencia de DNA de cadena sentido de un gen específico y contiene la secuencia nucleotídica complementaria de los sitios ligantes para los cebadores cadena abajo utilizados en subsiguientes reacciones de multiplicación,
(ii) un segmento central que consiste en una secuencia nucleotídica que comprende uno o múltiples nucleótidos, que no son complementarios de la secuencia de mRNA ni de la secuencia de DNA de cadena sentido de dicho gen, y
(iii) un segmento 3''-terminal que comprende una secuencia nucleotídica, que es complementaria de la secuencia de mRNA así como de la secuencia de DNA de cadena sentido de dicho gen,
(b) eliminar o inactivar los cebadores modificadores de secuencia superfluos después de la compleción de la transcripción inversa,
(c) llevar a cabo reacciones de multiplicación individuales para cada uno de los genes analizados, de modo que, en cada una de las reacciones, moldes de cDNA secuencialmente modificados se multipliquen conjuntamente con un molde de DNA de referencia, de manera competitiva utilizando los mismos cebadores génicamente específicos para ambos moldes para generar cantidades mensurables de productos de multiplicación,
(d) medir cuantitativamente las cantidades y determinar los niveles relativos de los productos de multiplicación derivados de los moldes de cDNA secuencialmente modificado y de DNA de referencia para obtener una relación de cDNA a DNA génicamente específica en cada una de las reacciones de multiplicación individuales, utilizando las modificaciones secuenciales en las moléculas de cDNA generadas durante la transcripción inversa para distinguir los productos de multiplicación derivados de cDNA de los productos de multiplicación derivados del molde de cDNA de referencia presentes en la misma reacción de multiplicación, y
(e) combinar las relaciones de cDNA a DNA génicamente específicas determinadas en las reacciones de multiplicación individuales para generar un perfil de dichas relaciones, específico de la muestra, que refleje las cantidades relativas de transcritos de mRNA originalmente presentes en la muestra
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/FI2005/050176.
Solicitante: EXPRESSION ANALYTICS OY.
Nacionalidad solicitante: Finlandia.
Dirección: KULMATIE 11,02730 ESPOO.
Inventor/es: EXPRESSION ANALYTICS OY.
Fecha de Publicación: .
Fecha Concesión Europea: 10 de Febrero de 2010.
Clasificación Internacional de Patentes:
- C12Q1/68D2C
Clasificación PCT:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Clasificación antigua:
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
Fragmento de la descripción:
Método para la medición cuantitativa y/o comparativa de niveles de expresión de mRNA en nuestras biológicas pequeñas.
Campo del invento
El presente invento se refiere al diagnóstico de estados patológicos y fisiológicos en seres humanos y al control del tratamiento así como al seguimiento de dichos estados. El invento se refiere también a la caracterización de cambios celular o tisularmente específicos en los patrones y vías de expresión génica de diferentes estados. La técnica descrita permite la medición comparativa y/o cuantitativa de transcritos de mRNA contenidos en pequeñas muestras celulares o tisulares.
Fundamento del invento
El desarrollo de tecnologías relacionadas con ciencias biológicas ha conducido a un rápido aumento de la información genética disponible. La compleción de la secuenciación del genoma humano y la política del Proyecto del Genoma Humano sobre accesibilidad inmediata a los datos han permitido crear el fundamento para estudiar la expresión génica en estados fisiológicos y patológicos. Técnicas tales como las micromatrices para expresión génica, la reacción en cadena de la polimerasa con presentación diferencial (DD-PCR; del inglés, differential display polymerase chain reaction) y la reacción en cadena de la polimerasa con transcriptasa inversa en modo cuantitativo (qRT-PCR; del inglés, quantitative reverse transcriptase polymerase chain reaction), junto con algoritmos estadísticos de agrupamiento, han proporcionado herramientas para la caracterización de patrones y vías de expresión génica. Esto ha conducido a la definición de nuevas subcategorías de enfermedades conocidas, basadas en perfiles de expresión génica, con pronósticos diferentes y respuestas potencialmente diferentes a fármacos específicos y otras modalidades de tratamiento.
La tecnología de micromatrices de DNA ha sido el principal contribuyente a la rápida acumulación de datos de expresión génica. Utilizando esta tecnología, se han desarrollado clasificaciones basadas en expresión génica para diversas enfermedades malignas. Sin embargo, en la actualidad, la tecnología de micromatrices para expresión génica presenta importantes limitaciones técnicas como método diagnóstico en un laboratorio clínico ya que es bastante insensible y su precisión cuantitativa sólo es moderada.
El análisis de la expresión génica en un escenario clínico está sometido a desafíos técnicos específicos a causa de las características de los diferentes tipos de muestras clínicas. Las muestras tisulares sólidas, tal como una muestra histológica de tejido embebido en parafina o congelado, contienen normalmente una mezcla heterogénea de diferentes tipos celulares. La medición de la expresión génica en un tipo celular o tisular específico de una muestra requiere el aislamiento de células o islotes de células a partir de secciones microscópicas, por ejemplo, por microdisección, o la graduación de las cantidades relativas de los diferentes tipos celulares presentes en secciones cortadas de la muestra. En biopsias con aguja y muestras citológicas, la cantidad de células muestreadas es normalmente pequeña y se desconoce la cantidad exacta de células. En muestras de sangre, la proporción de células diana es pequeña en muchos casos y se requiere el enriquecimiento de las muestras en cuanto a tipos celulares específicos. La recuperación de RNA mensajero (mRNA) de estos tipos de muestras es a menudo limitada, lo que aumenta los requisitos de sensibilidad para las técnicas utilizadas para el análisis de la expresión génica. Además, durante la preparación y el almacenamiento de las muestras, así como durante la purificación del ácido nucleico, se producen variaciones en la degradación y recuperación del mRNA de una muestra a otra. Con objeto de obtener resultados comparables de las muestras tisulares obtenidas in vivo, se requiere la normalización de las variaciones en los niveles y la integridad del mRNA de una muestra a otra.
La qRT-PCR ha sido muy usada para validar los resultados sobre niveles de expresión génica que se han obtenido al utilizar micromatrices para expresión génica. La sensibilidad de la qRT-PCR es suficiente para la medición cuantitativa de la expresión génica en muestras que contienen cantidades mínimas de células o incluso células sueltas. A causa de la naturaleza logarítmica de la multiplicación de los ácidos nucleicos durante la PCR, esta técnica es sensible a variaciones de un tubo a otro debidas a pequeñas diferencias en las eficacias de reacción. Para solventar este problema, se pueden añadir moldes testigo internos de RNA o DNA a las muestras para determinar las eficacias de reacción en las reacciones individuales. Las variaciones en los niveles y la integridad del mRNA de una muestra a otra son típicamente controladas normalizando los niveles de expresión de mRNA de los genes específicos de interés con respecto a los niveles de expresión de genes domésticos o a la cantidad de RNA total o RNA ribosómico (rRNA) en la muestra, o mediante la cuantificación comparativa del mRNA de múltiples genes en la misma muestra.
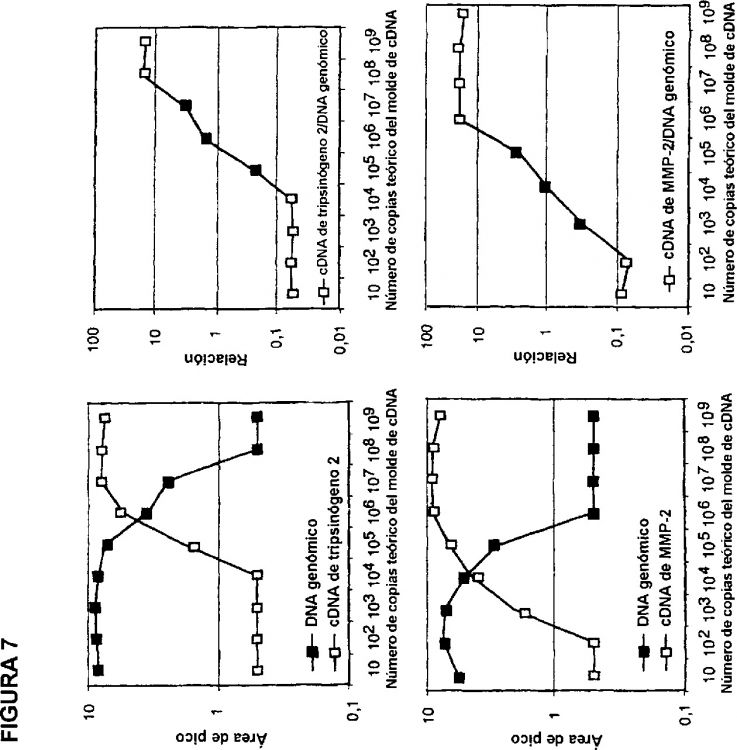
Joo et al. [en el Journal of Virological Methods 100 (2002), páginas 71-81, y en la solicitud KR2002089746 A de patente] han descrito un método para modificar el tamaño del molde de cDNA durante la transcripción inversa con objeto de discriminarlo del DNA genómico. En esta técnica, se utiliza un cebador ancla modificador del tamaño en la reacción de transcripción inversa para insertar, en el cDNA generado, un sitio ligante del cebador modificado. Esta técnica se basa en la suposición de que el DNA genómico permanece como doble cadena cuando se lleva a cabo la transcripción inversa bajo condiciones no desnaturalizantes y, por lo tanto, no puede actuar como molde para la transcripción inversa. En la operación de multiplicación, se multiplican los moldes de cDNA y de DNA genómico del mismo gen utilizando un cebador común cadena arriba y cebadores cadena abajo distintos. Los amplicones derivados de cDNA y DNA genómico generados difieren en cuanto a longitud para permitir la detección y la cuantificación separadas. Generalmente, las técnicas de RT-PCR requieren la multiplicación de una secuencia que atraviesa al menos un límite de exón-exón, con objeto de permitir la separación de los productos de multiplicación derivados de cDNA y de DNA genómico. La técnica descrita por Joo et al. permite la diferenciación del cDNA de su correspondiente DNA genómico dentro de los límites de un solo exón después de la multiplicación por RT-PCR. También permite la comparación de los niveles relativos de mRNA y DNA génicamente específicos.
D. Sun et al. ("Quantifying porphobilinogen deaminase mRNA in microdissected nephron segments by a modified RT-PCR", Kidney International, volumen 61, 2002, páginas 336-341) describen un método para la valoración cuantitativa de la cantidad de transcritos de mRNA presentes en una muestra celular o tisular mediante RT-PCR mutagénica ("MRT-PCR", páginas 337-338).
El presente invento proporciona un método para la valoración cuantitativa y/o comparativa de las cantidades relativas de transcritos de mRNA presentes en una muestra celular o tisular. En este método, moldes de cDNA secuencialmente modificados son conjunta y competitivamente multiplicados con un molde de DNA de referencia utilizando los mismísimos cebadores. Esto permite utilizar el DNA genómico contenido en, o añadido a, la muestra como un molde de referencia universal para normalizar las variaciones en las eficacias de multiplicación de un tubo a otro entre distintas reacciones de multiplicación génicamente específicas. Al medir cuantitativamente las cantidades y determinar los niveles relativos de los productos de multiplicación derivados de moldes de cDNA secuencialmente modificado y de DNA de referencia, se obtiene una relación génicamente específica de cDNA a DNA en cada reacción de multiplicación individual. Al combinar las relaciones de cDNA a DNA para cada uno de los genes analizados, se genera un perfil de expresión de genes específicos de muestras que refleja las cantidades relativas de transcritos de mRNA originalmente presentes en la muestra.
En el presente invento, se multiplican conjuntamente moldes de cDNA secuencialmente modificado y de DNA de referencia con los mismísimos cebadores en los mismos recipientes de reacción. De esta manera, los niveles relativos de los productos de multiplicación derivados de los moldes de cDNA y de referencia permanecen constantes incluso cuando...
Reivindicaciones:
1. Un método para la valoración cuantitativa y/o comparativa de las cantidades relativas de transcritos de mRNA presentes en una muestra celular o tisular, caracterizado por las operaciones de:
2. Un método de acuerdo con la Reivindicación 1, caracterizado por que la muestra comprende un producto celular o tisular de lisis u homogeneización.
3. Un método de acuerdo con la Reivindicación 1, caracterizado por que, ante de la transcripción inversa, se aísla RNA o RNA y DNA de la muestra.
4. Un método de acuerdo con la Reivindicación 1, caracterizado por que la reacción de transcripción inversa se lleva a cabo en unas condiciones bajo las cuales el DNA contenido en la muestra permanece en forma de doble cadena.
5. Un método de acuerdo con la Reivindicación 1, caracterizado por que los cebadores modificadores de secuencia superfluos son eliminados o inactivados por degradación enzimática después de la compleción de la transcripción inversa.
6. Un método de acuerdo con la Reivindicación 5, caracterizado por que dicha degradación enzimática se lleva a cabo utilizando una exonucleasa específica de DNA de cadena sencilla.
7. Un método de acuerdo con la Reivindicación 1, caracterizado por que las reacciones de multiplicación individuales se llevan a cabo en recipientes de reacción físicamente separados.
8. Un método de acuerdo con la Reivindicación 1, caracterizado por que el molde de DNA de referencia comprende DNA genómico contenido en la muestra o aislado de una fuente distinta.
9. Un método de acuerdo con la Reivindicación 1, caracterizado por que el molde de DNA de referencia comprende oligonucleótidos o polinucleótidos de DNA clonados o sintetizados.
10. Un método de acuerdo con la Reivindicación 1, caracterizado por que la medición cuantitativa de los productos de multiplicación se lleva a cabo durante la multiplicación.
11. Un método de acuerdo con la Reivindicación 1, caracterizado por que la medición cuantitativa de los productos de multiplicación se lleva a cabo después de la compleción de la reacción de multiplicación.
12. Un método de acuerdo con la Reivindicación 1, caracterizado por que los niveles relativos de los productos de multiplicación que derivan de los moldes de cDNA secuencialmente modificado y de DNA de referencia en una reacción de multiplicación individual génicamente específica reflejan las cantidades relativas de dichos moldes originalmente presentes en la reacción de multiplicación antes de la multiplicación.
13. Un método de acuerdo con la Reivindicación 1, caracterizado por que, para la multiplicación de los moldes de cDNA y de DNA de referencia, se utiliza la reacción en cadena de la polimerasa, la reacción en cadena de la ligasa o la multiplicación mediada por transcripción.
14. Un método de acuerdo con la Reivindicación 1, caracterizado por que, para la medición cuantitativa de los productos de multiplicación, se utilizan el análisis de curvas de fusión, la extensión de cebadores en el sitio de diferenciación, sondas de hibridación secuencialmente específicas, la espectrometría de masas, o ensayos basados en la transferencia directa o indirecta de energía por resonancia de fluorescencia.
15. Un método de acuerdo con la Reivindicación 14, caracterizado por que la extensión de cebadores en el sitio de diferenciación es seleccionada del grupo que consiste en minisecuenciación, minisecuenciación cíclica, Pyro-sequencing® y extensión de cebadores específicos de alelo.
16. Un método de acuerdo con la Reivindicación 14, caracterizado por que las sondas de hibridación secuencialmente específicas son seleccionadas del grupo que consiste en sondas de hidrólisis doblemente marcadas y sondas Molecular Beacon®.
17. Un método de acuerdo con la Reivindicación 1, caracterizado por que en el método se aplica un kit de ensayo que comprende cebadores modificadores de secuencia para varios genes, un medio para eliminar o inactivar cebadores modificadores de secuencia superfluos después de la compleción de la transcripción inversa, y cebadores génicamente específicos adecuados para la multiplicación conjunta de moldes de cDNA secuencialmente modificado y un molde de DNA de referencia de manera competitiva en reacciones de multiplicación individuales para cada uno de los genes analizados.
Patentes similares o relacionadas:
MÉTODO DE DIAGNÓSTICO Y DE SEGUIMIENTO DE UNA VAGINOSIS BACTERIANA MEDIANTE CUANTIFICACIÓN MOLECULAR, del 28 de Diciembre de 2011, de UNIVERSITE DE LA MEDITERRANEE, AIX-MARSEILLE II: Método de diagnóstico y de seguimiento in vitro del estado de la flora bacteriana vaginal frente a la presencia de una vaginosis bacteriana […]
SISTEMAS Y MÉTODOS PARA DETERMINAR LOS COEFICIENTES DE INTERFERENCIA EN LA PCR Y OTROS CONJUNTOS DE DATOS, del 21 de Diciembre de 2011, de F. HOFFMANN-LA ROCHE AG: Un método para determinar los coeficientes de interferencia de un sistema de detección óptica de la reacción en cadena de la polimerasa (PCR) con al menos dos […]
METODO PARA LA SUBCLASIFICACION DE TUMORES, del 20 de Junio de 2011, de FUNDACION PARA LA INVESTIGACION BIOMEDICA DEL HOSPITAL UNIVERSITARIO LA PAZ: Método para la subclasificación de tumores.La presente invención se encuadra dentro del campo de la biología molecular y la medicina. Específicamente, la presente invención […]
 MÉTODO Y SISTEMA PARA ANALIZAR REACCIONES USANDO UN SISTEMA DE INFORMACIÓN, del 4 de Mayo de 2011, de ABBOTT LABORATORIES: Un método de determinación de si una muestra de test contiene un ácido nucleico objetivo, comprendiendo el método: (a) poner en contacto […]
MÉTODO Y SISTEMA PARA ANALIZAR REACCIONES USANDO UN SISTEMA DE INFORMACIÓN, del 4 de Mayo de 2011, de ABBOTT LABORATORIES: Un método de determinación de si una muestra de test contiene un ácido nucleico objetivo, comprendiendo el método: (a) poner en contacto […]
PCR EN TIEMPO REAL CON ADICION DE PIROFOSFATASA, del 11 de Mayo de 2010, de ROCHE DIAGNOSTICS GMBH F.HOFFMANN-LA ROCHE AG: Composición para amplificar y detectar un ácido nucleico diana, que consta de - una ADN polimerasa termoestable - una mezcla de desoxinucleósido-trifosfatos - […]
 PROCEDIMIENTO PARA DETERMINAR EL NUMERO DE COPIAS, del 21 de Abril de 2010, de MEDICAL RESEARCH COUNCIL: Procedimiento de medición de la frecuencia del número de copias de una o más secuencias de ácido nucleico en una muestra, que comprende las etapas siguientes:
(a) […]
PROCEDIMIENTO PARA DETERMINAR EL NUMERO DE COPIAS, del 21 de Abril de 2010, de MEDICAL RESEARCH COUNCIL: Procedimiento de medición de la frecuencia del número de copias de una o más secuencias de ácido nucleico en una muestra, que comprende las etapas siguientes:
(a) […]
DETECCION DE BACTERIAS DEL ACIDO LACTICO PRODUCTORAS DE HISTAMINA MEDIANTE REACCION EN CADENA DE LA POLIMERASA A TIEMPO REAL (QRT-PCR) Y SU USO, del 16 de Noviembre de 2009, de CONSEJO SUPERIOR INVESTIG. CIENTIFICAS: Detección de bacterias del ácido láctico productoras de histamina mediante reacción en cadena de la polimerasa a tiempo real (QRT-PCR) y su uso. La presente invención […]
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]