MÉTODO DE EVALUACIÓN PARA EVALUAR UNA POSIBILIDAD DE CÁNCER DE MAMA.
Método de evaluación para evaluar una posibilidad de cáncer de mama.
La presente invención se refiere a un método para evaluar una posibilidad de cáncer de mama en un sujeto, donde el método incluye detectar y cuantificar en suero o plasma micro-RNA específicos cuya expresión está relacionada con la presencia de tumor de mama. Igualmente, la invención se refiere al uso de micro-RNA específicos cuya expresión está relacionada con la presencia de tumor de mama.
Tipo: Patente de Invención. Resumen de patente/invención. Número de Solicitud: P201430901.
Solicitante: Sistemas Genómicos, S.L.
Nacionalidad solicitante: España.
Inventor/es: SAUS CANO,Lorena, BALLESTER DE MATIAS,Alida, TRIVIÑO PARDO,Juan Carlos, CABO DÍEZ,Miguel, RODRIGUEZ CRUZ,Oscar.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
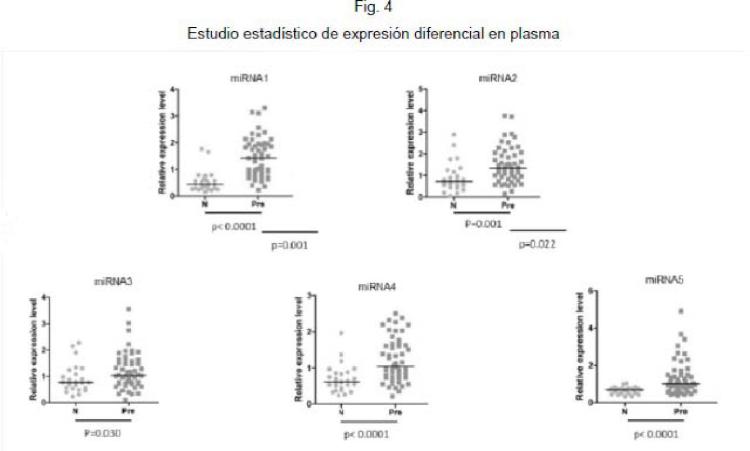
Fragmento de la descripción:
Método de evaluación para evaluar una posibilidad de cáncer de mama.
La presente invención se refiere a un método para evaluar una posibilidad de cáncer de mama en un sujeto, donde el método incluye detectar y cuantificar en suero o plasma micro-RNA específicos cuya expresión está relacionada con la presencia de tumor de mama. Igualmente, la invención se refiere al uso de micro-RNA específicos cuya expresión está relacionada con la presencia de tumor de mama.
La presente invención se enmarca en el campo del desarrollo de métodos no invasivos de diagnóstico precoz de transformaciones neoplásicas mamarias, por ejemplo como complemento a los habituales diagnósticos por imagen.
El cáncer de mama es uno de los cinco tipos de cáncer más comunes, siendo su elevada incidencia la causa principal de intensas investigaciones enfocadas principalmente a una detección precoz y un rápido diagnóstico. En este contexto, las ciencias ómicas han permitido grandes avances en la búsqueda de marcadores moleculares específicos para la detección precoz y el diagnóstico de este de tipo de cáncer (Kim BMC Bioinformatics 2010). De entre los marcadores moleculares estudiados, ciertos micro-RNA se están valorando en cuanto a su uso potencial como marcadores de diagnóstico y pronóstico (Shi, Cáncer TRat Rev 2009).
Los micro-RNA (miRNA) son pequeñas moléculas de RNA no codificante de cadena simple que modulan la expresión génica y cuyo tamaño es de dieciocho a veinticinco nucleótidos (Aoife L. Clin Can Res, 2008; Bartels, Clin Chem 2009; Zoon, Exp. REv. Mol. Diagn. 2009). Los mi-RNA actúan como reguladores post-transcripcionales y controlan procesos celulares muy diversos, como desarrollo y diferenciación, ciclo celular y apoptosis. Así, se expresan en el núcleo celular de eucariotas, tanto en regiones intragénicas como intergénicas, y pueden regular la expresión de genes a nivel post-transcripcional bloqueando la traducción y/o induciendo la degradación del RNA mensajero (mRNA) uniéndose a su región 3 no traducida (UTR) complementaria. Diversos estudios recientes indican que los microRNA están implicados en la patogénesis de todos los tipos de cáncer en humanos (MICRO-RNAS: NUEVOS MARCADORES DE INTERÉS EN ONCOLOGÍA, Ginés Luengo Gil, Servicio de Hematología y Oncología Médica, Hospital Universitario Morales Meseguer, Centro Regional de Hemodonación y Departamento de Medicina Interna, Universidad de Murcia, REVISTA EUBACTERIA (ABRIL 2012) N°28 / ISSN-1697-0071).
Hasta el momento se han identificado aproximadamente unos 1.000 miRNA en humanos, de los cuales aproximadamente 200 están relacionados con procesos tumorales, aunque se sugiere que existen decenas de miles. La aparición y el continuo desarrollo de técnicas de secuenciación masiva ha sido clave para el descubrimiento y la identificación de este tipo de moléculas, ya que su pequeño tamaño y la baja concentración en la que están presentes en las células eucarióticas dificulta su detección mediante técnicas moleculares tradicionales (Nygaard, BMC Med Gen, 2009). Además, un aspecto clave en la biogénesis de estas pequeñas moléculas de RNA que las hace atractivas como biomarcadores moleculares es el hecho de que se mantienen estables en suero y plasma, permitiendo su detección tanto en tejidos específicos como en este tipo de fluidos (Lodes, PLoS one, 2009).
En el sistema de nomenclatura estándar, los microRNA (miRBase) se nombran con un nombre compuesto de 4 partes, del tipo mmu-miR-375-5p. Las tres primeras letras indican la especie, por ejemplo hsa (homo sapiens) para humano. Un nombre de microRNA en minúscula (por ejemplo hsa-mir-22) hace referencia al gen de microRNA o al pre-microRNA, mientras que al microRNA maduro se le asocia el término miR (como en hsa-miR-22-5p). La referencia numérica asociada al nombre se asigna de forma secuencial, mientras que la última referencia alfanumérica se refiere al brazo (5p o 3p, respectivamente 5 o 3).
Se han caracterizado aproximadamente unos 20-30 miRNA específicos diferencialmente expresados en cáncer de mama. Algunos de los ejemplos mejor caracterizados son miR-21, que tiene un carácter oncogénico; miR-29A, miR-17-5p, miR-26A, miR-181A, miR-22, miR196A-2, etc. (Maillot, Cáncer Res 2009; Bourguignon JBC 2009; Wu, Journal of Biomedicine and Biotechnology, 2011). Actualmente, los patrones de expresión de estos miRNA específicos se están utilizando para diferenciar el tejido sano mamario adyacente al tejido tumoral con el fin de clasificar los distintos tipos de tumores y avanzar en el pronóstico (Lowery, Clin Can Res 2008; Enerly, PLoS One 2011, Rothe, PLoS One 2011).
Los cánceres de mama se pueden clasificar en subtipos moleculares en función de diferentes perfiles de expresión genética. Perou y col. (Perou, C.M., Sorlie, T., Eisen, M.B., van de Rijn, M., Jeffrey, S.S., Rees, C.A., Pollack, J. R., Ross, D.T., Johnsen, H., Akslen, LA., y col. (2000), Molecular portraits of human breast tumours, Nature 406, 747-752) revelaron la estratificación molecular de los tumores mamarios en subtipos moleculares reproducibles de cáncer de mama con diferentes características biológicas, resultados clínicos y respuestas a la terapia. La clasificación incluye dos grupos basados en la expresión génica del receptor estrógeno (ER): ER+ (luminal A, luminal B, luminal C) y ER- (Her-2, tipo basal o triple negativo, tipo normal). Estos subgrupos se han relacionado con diferentes evoluciones de la enfermedad, lo que indica la existencia de una base biológica como causa de la heterogeneidad clínica del cáncer de mama.
Así, la identificación de microRNA específicos para los distintos tumores de mama podría contribuir de manera efectiva y probablemente más precisa al diagnóstico y pronóstico de la enfermedad.
Por tanto, dado el potencial de este tipo de marcadores, se ha investigado activamente cómo se produce se liberación de los miRNA específicos expresados en las células malignas tumorales al torrente sanguíneo. Para algunos MiRNA como miR-451 y miR-1246 se ha demostrado que son expresados en células epiteliales mamarias malignas y liberados selectivamente a la circulación, mientras que son retenidos intracelularmente en células epiteliales sanas (Pigati, PLoS One, 2010). Estudios recientes demuestran que estos miRNA específicos pueden ser detectadles mediante reacciones de amplificación a partir de sangre periférica, ya que estas moléculas son de pequeño tamaño y muy estables (Zhao, Cáncer PLoS One 2010).
Por ejemplo, del documento WO2014023797 se conoce un método ex vivo para determinar si las células tumorales de un paciente son o podrían ser resistentes a un medicamento que modula la unión de una hormona esteroidea a su receptor. El método descrito en este documento permite seleccionar qué tipo de tratamiento hormonal sería más adecuado en cada caso para ciertos tipos de cáncer de mama.
La US 20120214864 se refiere a un método para identificar un tumor triple negativo en una muestra procedente de un sujeto, en particular tejido tumoral, donde un nivel disminuido de determinados miRNA comparados con los correspondientes a un tumor luminal indican que el sujeto padece un cáncer triple negativo.
La presente invención proporciona un método para evaluar una posibilidad de cáncer de mama en un sujeto, donde el método incluye detectar y cuantificar en una muestra de suero o plasma del sujeto en cuestión la expresión de al menos un micro-RNA específico seleccionado de entre hsa-m¡R-142-3p (UGUAGUGUUUCCUACUUUAUGGA, SEQ ID NO: 1, N° de acceso MIMAT0000434), hsa-miR-505-5p (GGGAGCCAGGAAGUAUUGAUGU, SEQ ID NO: 2, N° de acceso MIMAT0004776), hsa-miR-21-5p (UAGCUUAUCAGACUGAUGUUGA, SEQ ID NO: 3, N° de acceso MIMAT0000076), hsa-m¡R-125b-5p (UCCCUGAGACCCUAACUUGUGA, SEQ ID NO: 4, N° de acceso MIMAT0000423) y hsa-miR-3656 (GGCGGGUGCGGGGGUGG, SEQ ID NO: 5, N° de acceso MIMAT0018076) y/o combinaciones de los mismos, específicos de la
población española, cuya expresión está relacionada con la presencia de tumor de mama; comparar la expresión mediante análisis estadístico con un valor de referencia obtenido de un sujeto sano para determinar si existe sobreexpresión; y evaluar la posibilidad de cáncer de mama en base a una posible presencia tumoral cuando el al menos un miRNa está sobreexpresado en la muestra en un valor superior a un valor de corte óptimo.
Preferentemente, en el método anteriormente descrito se utiliza una combinación de hsa-miR- 125b-5p (UCCCUGAGACCCUAACUUGUGA, SEQ ID NO: 4, N° de acceso MIMAT0000423) y hsa-m¡R-3656 (GGCGGGUGCGGGGGUGG, SEQ ID NO: 5, N° de acceso MIMAT0018076).
Igualmente, la invención se refiere al uso de los micro-RNA específicos citados o de combinaciones de los...
Reivindicaciones:
Método para evaluar una posibilidad de cáncer de mama en un sujeto, incluyendo el método
i. detectar y cuantificar en una muestra de suero o plasma del sujeto la expresión de al menos un micro-RNA específico seleccionado de entre hsa-miR-142-3p (UGUAGUGUUUCCUACUUUAUGGA, SEQ ID NO: 1), hsa-miR-505-5p (GGGAGCCAGGAAGUAUUGAUGU, SEQ ID NO: 2), hsa-miR-21-5p (UAGCUUAUCAGACUGAUGUUGA, SEQ ID NO: 3), hsa-miR-125b-5p (UCCCUGAGACCCUAACUUGUGA, SEQ ID NO: 4) y hsa-miR-3656 (GGCGGGUGCGGGGGUGG, SEQ ID NO: 5)
y/o de cualesquiera combinaciones de los mismos, específicos de la población española;
ii. comparar la expresión mediante análisis estadístico con un valor de referencia obtenido de un sujeto sano para determinar si existe sobreexpresión; y
iii. evaluar la posibilidad de cáncer de mama en base a una posible presencia tumoral cuando el al menos un miRNa o una combinación de cualesquiera de ellos está sobreexpresado en la muestra en un valor superior a un valor de corte óptimo.
Método para evaluar una posibilidad de cáncer de mama en un sujeto según la reivindicación 1, caracterizado porque en el paso i. se selecciona la combinación de hsa-miR-125b-5p, SEQ ID NO: 4, y hsa-miR-3656, SEQ ID NO: 5.
Utilización de al menos un micro-RNA específico seleccionado de entre hsa-miR-142-3p (UGUAGUGUUUCCUACUUUAUGGA, SEQ ID NO: 1), hsa-miR-505-5p (GGGAGCCAGGAAGUAUUGAUGU, SEQ ID NO: 2), hsa-miR-21-5p (UAGCUUAUCAGACUGAUGUUGA, SEQ ID NO: 3), hsa-miR-125b-5p (UCCCUGAGACCCUAACUUGUGA, SEQ ID NO: 4) y hsa-miR-3656 (GGCGGGUGCGGGGGUGG, SEQ ID NO: 5) y/o de combinaciones de cualesquiera de los mismos,
específicos de la población española, en un método según la reivindicación 1.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]