LIGANDOS DE ÁCIDO NUCLEICO QUE SE UNEN A ADN POLIMERASAS Y LAS INHIBEN.
Ligando de ácido nucleico para una ADN polimerasa termoestable,
en el que el ligando de ácido nucleico se une a dicha polimerasa e inhibe a la misma a temperaturas ambientes, aunque permite que la polimerasa se active a temperaturas elevadas
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E09000032.
Solicitante: GILEAD SCIENCES, INC..
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 333 LAKESIDE DRIVE FOSTER CITY, CA 94404 ESTADOS UNIDOS DE AMERICA.
Inventor/es: GOLD, LARRY, JAYASENA,SUMEDHA.
Fecha de Publicación: .
Fecha Solicitud PCT: 5 de Junio de 1996.
Clasificación PCT:
- C07H21/04 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07H AZUCARES; SUS DERIVADOS; NUCLEOSIDOS; NUCLEOTIDOS; ACIDOS NUCLEICOS (derivados de ácidos aldónicos o sacáricos C07C, C07D; ácidos aldónicos, ácidos sacáricos C07C 59/105, C07C 59/285; cianohidrinas C07C 255/16; glicales C07D; compuestos de constitución indeterminada C07G; polisacáridos, sus derivados C08B; ADN o ARN concerniente a la ingeniería genética, vectores, p. ej. plásmidos o su aislamiento, preparación o purificación C12N 15/00; industria del azúcar C13). › C07H 21/00 Compuestos que contienen al menos dos unidades mononucleótido que tienen cada una grupos fosfato o polifosfato distintos unidos a los radicales sacárido de los grupos nucleósido, p. ej. ácidos nucleicos. › con desoxirribosilo como radical sacárido.
- C12P19/34 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 19/00 Preparación de compuestos que contienen radicales sacárido (ácido cetoaldónico C12P 7/58). › Polinucleótidos, p. ej. ácidos nucleicos, oligorribonucleótidos.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
Países PCT: Austria, Bélgica, Suiza, Alemania, Dinamarca, España, Francia, Reino Unido, Grecia, Italia, Liechtensein, Luxemburgo, Países Bajos, Suecia, Mónaco, Portugal, Irlanda, Finlandia.
PDF original: ES-2358235_T3.pdf

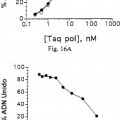

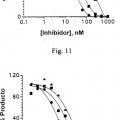

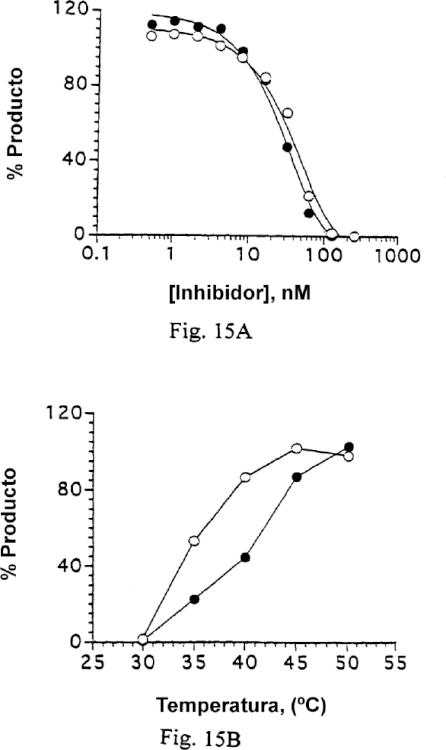
Fragmento de la descripción:
Campo de la invención 5
En la presente invención se describen procedimientos para identificar y preparar ligandos de ácido nucleico con alta afinidad por las ADN polimerasas, específicamente para las ADN polimerasas termoestables. En una realización preferida, la ADN polimerasa se selecciona de entre la polimerasa Taq, una polimerasa termoestable aislada de Thermus aquaticus, o la polimerasa Tth, una ADN polimerasa y transcriptasa inversa termoestable aislada 10 de Thermus thermophilus. El procedimiento utilizado en la presente invención para identificar tales ligandos de ácido nucleico se denomina SELEX, un acrónimo (en inglés) de Evolución Sistemática Ligandos mediante Enriquecimiento Exponencial. También se describen en la presente invención un procedimiento mejorado para realizar la Reacción en Cadena de la Polimerasa usando los ligandos de ácido nucleico de esta invención. En la presente invención específicamente se describen ligandos de ácido nucleico de elevada afinidad por la polimerasa Taq y polimerasa 15 Tth. La invención incluye ligandos de ADN de alta afinidad, los cuales se unen a la polimerasa Taq y polimerasa Tth, inhibiendo de ese modo su capacidad para polimerizar la síntesis del ADN a temperaturas ambiente. En esta invención se incluyen además interruptores de ácido nucleico. Los ligandos de ácido nucleico de esta invención, con unión dependiente de la temperatura a las ADN polimerasas, son ejemplo de ligandos cuyas propiedades deseables puede activarse y desactivarse en base a cualquier número de condiciones de reacción. 20
Antecedentes de la invención
La Reacción en Cadena de la Polimerasa (PCR) es una técnica recientemente desarrollada que ha tenido un impacto significativo en muchas áreas de la ciencia. La PCR es un procedimiento rápido y sencillo para 25 amplificar específicamente una secuencia diana de ADN de forma exponencial. (Saiki et al. (1985) Science 230:1350; Mullis y Faloona (1987) Methods Enzymol. 155:335). De forma resumida, el procedimiento consiste en sintetizar un grupo de cebadores que tienen secuencias nucleotídicas complementarias del ADN que flanquea la secuencia diana. Los cebadores se mezclan a continuación con una solución del ADN diana, una polimerasa de ADN termoestable, y los cuatro desoxinucleótidos (A, T, C y G). La solución se calienta entonces hasta una 30 temperatura suficiente para separar las hebras de ADN complementarias (aproximadamente 95˚C), y se enfría a continuación hasta una temperatura suficiente para permitir que los cebadores se unan a las secuencias flanqueantes. La mezcla de reacción se caliente entonces de nuevo (hasta aproximadamente 72˚C) para permitir el progreso de la síntesis del ADN. Después de un breve período de tiempo, la temperatura de la reacción se incrementa de nuevo hasta una temperatura suficiente para separar el recién formado ADN de doble hebra, 35 completándose de este modo el primer ciclo de la PCR. La mezcla de reacción se enfría entonces y se repite el ciclo. Por tanto, la PCR consiste en ciclos repetitivos de fusión, hibridación y síntesis del ADN. Veinte ciclos de replicación pueden rendir una amplificación de hasta un millón de veces de la secuencia diana de ADN. La capacidad para amplificar una única molécula de ADN mediante la PCR tiene aplicaciones en la microbiología ambiental y alimentaria (Wernars et al. (1991) Appl. Env. Microbiol. 57:1914-1919; Hill y Keasler (1991) Int. J. Food Microbiol. 40 12:67-75), en la microbiología clínica (Wages et al. (1991) J. Med. Virol. 33:58-63; Sacramento et al. (1991) Mol. Cell Probes 5:229-240; Laure et al. (1988) Lancet 2:538), en la oncología (Kumar y Barbacid (1988) Oncogene 3:647-651; McCormick (1989) Cancer Cells 1:56-61; Crescenzi et al. (1988) Proc. Natl. Acad. Sci. USA 85:4869), en la prognosis de enfermedades genéticas (Handyside et al. (1990) Nature 344:768-770), en los bancos de sangre (Jackson (1990) Transfusion 30:51-57) y en las ciencias forenses (Higuchi et al. (1988) Nature (London) 332:543). 45
La disponibilidad de ADN polimerasas termoestables, tales como la polimerasa de ADN Taq, ha simplificado y mejorado la PCR. Originalmente, sólo estaban disponibles polimerasas sensibles al calor, tales como la ADN polimerasa de E. coli, para su uso en la PCR. Sin embargo, las polimerasas sensibles al calor se destruyen a las temperaturas necesarias para fundir el ADN de doble hebra, y debe añadirse polimerasa adicional después de 50 cada ciclo de la PCR. La polimerasa de ADN Taq, aislada de la bacteria termofílica Thermus aquaticus, es estable hasta los 95˚C, y su uso en la PCR ha eliminado la necesidad de añadir de forma repetitiva de polimerasas sensibles a la temperatura después de cada ciclo térmico. Adicionalmente, puesto que la polimerasa Taq puede usarse a temperaturas más elevadas, ha mejorado la especificidad y sensibilidad de la PCR. La razón de la especificidad mejorada y que a temperaturas más elevadas, la unión de los promotores a sitios distintos de los deseados 55 (fenómeno denominado "mispriming") está significativamente reducida.
Desde su descubrimiento, la Reacción en Cadena de la Polimerasa ha sido modificada para varias aplicaciones, tales como la PCR in situ, en la cual el límite de detección de la hibridación in situ tradicional se ha llevado hasta el nivel de la copia única (Haase et al. (1990) Proc. Natl. Acad. Sci. USA 87:4971-4975), y la PCR con 60 transcriptasa inversa (RT-PCR), en donde una secuencia de ARN se convierte en su copia de ADN (ADNc) mediante la transcriptasa inversa (RT) antes de ser amplificada por la PCR, convirtiendo el ARN en un sustrato para la PCR
(Kawasaki (1991) "Amplification of RNA in PCR Protocols. A Guide to Methods and Applications," editores Innis et al., Academic Press Inc., San Diego, CA, 21-27). Sin embargo, las transcriptasas inversas víricas mesofílicas a menudo son incapaces de sintetizar moléculas de ADNc de longitud completa, porque no pueden "leer a lo largo" de las estructuras secundarias estables de las moléculas de ARN. Esta limitación se ha superado recientemente mediante el uso de una polimerasa aislada de Thermus thermophilus (polimerasa Tth). La polimerasa Tth es una 5 polimerasa termoestable que puede funcionar como transcriptasa inversa y como ADN polimerasa (Myers y Gelfand (1991) Biochemistry 30:7662-7666). La transcripción inversa realizada a una temperatura elevada usando la polimerasa Tth elimina las estructuras secundarias del ARN plantilla, haciendo posible la síntesis de ADNc de longitud completa.
10
Aunque se ha hecho un progreso significativo en la tecnología de la PCR, la amplificación de oligonucleótidos que no son diana debida a reacciones secundarias, tales como el cebado incorrecto del ADN de fondo y/o la oligomerización del cebador, todavía presenta un problema significativo. Esto es especialmente cierto en las aplicaciones de diagnóstico en las cuales la PCR se lleva a cabo en un medio que contiene ADN de fondo, mientras que el ADN puede estar presente en una única copia (Chou et al. (1992) Nucleic Acid Res. 20:717-1723). 15 Se ha determinado que estas reacciones secundarias a menudo ocurren cuando todos los reactivos se han mezclado a temperatura ambiente antes de iniciarse el ciclado térmico.
Se han descrito dos procedimientos que minimizan estas reacciones secundarias. En el primer procedimiento, denominado PCR "de arranque en caliente (hot start)" (Chou et al. (1992) Nucleic Acid Res. 20:1717-20 1723; D'Aquila et al. (1991) Nucleic Acid Res. 19:3749), todos los reactivos se calientan a 72˚C antes de añadir el reactivo final, usualmente la polimerasa. En la PCR "de arranque en caliente" mediada por cera, un componente o componentes cruciales para la actividad de la polimerasa se separa físicamente, a baja temperatura, del resto de la mezcla de reacción mediante una capa de cera que se funde a partir del calentamiento en el primer ciclo (Chou et al. (1992) Nucleic Acids Res. 20:1717; Horton et al. (1994) BioTechniques 16:42). La PCR "de arranque en caliente" 25 tiene ciertas desventajas; la obligatoriedad de reabrir los tubos antes de iniciar el termociclado incrementa la contaminación entrecruzada y el pipeteado repetido la hace tediosa a la hora de manipular un número elevado de muestras. Un reactivo que pueda colocarse directamente en la mezcla de reacción, junto con el resto de los componentes de reacción, e inhiba la polimerasa a temperatura ambiente será útil para superar las limitaciones... [Seguir leyendo]
Reivindicaciones:
1. Ligando de ácido nucleico para una ADN polimerasa termoestable, en el que el ligando de ácido nucleico se une a dicha polimerasa e inhibe a la misma a temperaturas ambientes, aunque permite que la polimerasa se active a temperaturas elevadas. 5
2. Ligando de ácido nucleico según la reivindicación 1, en el que la temperatura ambiente es la temperatura de la habitación o inferior a 40ºC.
3. Ligando de ácido nucleico según la reivindicación 1 ó la reivindicación 2, en el que la temperatura 10 elevada es superior a 40ºC.
4. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 3, en el que el ligando de ácido nucleico es ADN.
15
5. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 4, en el que dicho ligando de ácido nucleico es de cadena sencilla.
6. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 5, en el que dicha polimerasa deriva de la especie Thermus. 20
7. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 6, en el que dicha ADN polimerasa termoestable es polimerasa de Thermus aquaticus (Taq), polimerasa de Thermus thermophilus (Tth) o una transcriptasa inversa.
25
8. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 7, en el que el ligando de ácido nucleico comprende la secuencia de consenso:
5'-A/GA/GTGTG/ACAGTAT/GC-3'.
9. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 7, en el que el ligando de ácido 30 nucleico comprende la secuencia de una cualquiera de las SEC ID Nos. 50, 67, 68, 69 ó 74.
10. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 7, en el que el ligando de ácido nucleico es uno cualquiera de las SEC ID Nos. 50, 67, 68, 69 ó 74 o un ligando de ácido nucleico que tiene sustancialmente la misma capacidad de unirse e inhibir la actividad de Taq o Tth polimerasa que una de las 35 SEC ID Nos: 50, 67, 68, 69 ó 74.
11. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 7, en el que el ligando de ácido nucleico comprende la secuencia consenso:
5'-A/GCGTTTTG-3'. 40
12. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 7, en el que el ligando de ácido nucleico comprende la secuencia de cualquiera de las SEC ID Nos. 59 ó 70.
13. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 7, en el que el ligando de ácido 45 nucleico es la SEC ID Nos. 59 ó 70 o un ligando de ácido nucleico que tiene sustancialmente la misma capacidad de unirse e inhibir la actividad de Taq o Tth polimerasa que la SEC ID No: 59 ó 70.
14. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 7, en el que dicho ligando de ácido nucleico se selecciona del grupo que consiste en las secuencias establecidas en la Tabla 3 (SEC ID 50 Nos: 36-66, 76, 77), Tabla 2 (SEC ID Nos. 7-35), Tabla 4 (SEC ID Nos. 67-73) o la Tabla 5 (SEC ID No: 74), o las correspondientes secuencias complementarias de las mismas.
15. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 7, en el que dicho ligando de ácido nucleico tiene un grado de homología de secuencia primaria superior al 70% y tiene sustancialmente 55 la misma capacidad de unirse a Taq o Tth polimerasa que un ligando seleccionado del grupo que consiste en las secuencias establecidas en la Tabla 3 (SEC ID Nos: 36-66, 76, 77), Tabla 2 (SEC ID Nos. 7-35), Tabla 4 (SEC ID Nos. 67-73) o la Tabla 5 (SEC ID No: 74), o las correspondientes secuencias complementarias de las mismas.
60
16. Ligando de ácido nucleico según cualquiera de las reivindicaciones 1 a 7, en el que el ligando de ácido nucleico tiene una caperuza en 3'.
17. Reactivo de PCR que comprende un ligando de ácido nucleico según cualquiera de las reivindicaciones anteriores.
18. Método de producción de un ligando de ácido nucleico para un compuesto diana, en el que el ligando de ácido nucleico es un interruptor de ácido nucleico, comprendiendo dicho método la etapa de producir un 5 ligando de ácido nucleico basado en una secuencia de ácido nucleico identificada por un proceso que comprende las etapas de:
a) preparar una mezcla candidata de ácidos nucleicos;
b) poner en contacto la mezcla candidata de ácidos nucleicos con un compuesto diana, donde los ácidos nucleicos que tienen una mayor afinidad por la diana en relación con la mezcla candidata 10 se pueden separar del resto de la mezcla candidata;
c) separar los ácidos nucleicos de mayor afinidad del resto de la mezcla candidata, mediante lo cual los ácidos nucleicos con una mayor afinidad se separan posteriormente en base a su falta de afinidad con la diana después de la variación de un parámetro del medio; y
d) amplificar los ácidos nucleicos de mayor afinidad separados posteriormente en la 15 etapa c) para producir una mezcla de ácidos nucleicos enriquecida en secuencias de ácidos nucleicos con una afinidad y especificidad relativamente superior para la unión a la diana y la falta de afinidad con la diana después de la variación de un parámetro del medio,
mediante lo cual se pueden identificar los ligandos de ácido nucleico de la diana.
20
19. Método según la reivindicación 18, en el que el parámetro del medio es la temperatura.
20. Método según la reivindicación 18 ó 19, en el que la diana es una ADN polimerasa termoestable.
25
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]