Incorporación in vivo de alquinil aminoácidos a proteínas en eubacterias.
Una célula eubacteriana que comprende una primera aminoacil-ARNt sintetasa ortogonal (O-RS) que actúa en la célula,
en donde la O-RS preferentemente aminoacila un primer ARNt ortogonal (O-ARNt) con un primer aminoácido no natural que es un alquinil aminoácido;
en donde el alquinil aminoácido es una tirosina para-sustituida o una fenilalanina para-sustituida, en donde la tirosina o la fenilalanina están sustituidas en la posición para con un grupo etinilo o propinilo; y
en donde la O-RS aminoacila el O-ARNt con el primer aminoácido no natural con una eficiencia de supresión de al menos el 50 % de la eficiencia de supresión de un par sintetasa ortogonal, par que es el O-ARNt de la SEQ ID NO: 1 y una polipéptido sintetasa que comprende una secuencia de aminoácidos seleccionada entre una cualquiera de las SEQ ID NO: 4, 6, 8, 10, 12, 14, 16, 18, 21, 22, 23, 24 y 25.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2005/033784.
Solicitante: THE SCRIPPS RESEARCH INSTITUTE.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 10550 NORTH TORREY PINES ROAD LA JOLLA, CA 92037 ESTADOS UNIDOS DE AMERICA.
Inventor/es: SCHULTZ, PETER, DEITERS,ALEXANDER.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C07H21/04 QUIMICA; METALURGIA. › C07 QUIMICA ORGANICA. › C07H AZUCARES; SUS DERIVADOS; NUCLEOSIDOS; NUCLEOTIDOS; ACIDOS NUCLEICOS (derivados de ácidos aldónicos o sacáricos C07C, C07D; ácidos aldónicos, ácidos sacáricos C07C 59/105, C07C 59/285; cianohidrinas C07C 255/16; glicales C07D; compuestos de constitución indeterminada C07G; polisacáridos, sus derivados C08B; ADN o ARN concerniente a la ingeniería genética, vectores, p. ej. plásmidos o su aislamiento, preparación o purificación C12N 15/00; industria del azúcar C13). › C07H 21/00 Compuestos que contienen al menos dos unidades mononucleótido que tienen cada una grupos fosfato o polifosfato distintos unidos a los radicales sacárido de los grupos nucleósido, p. ej. ácidos nucleicos. › con desoxirribosilo como radical sacárido.
- C12N1/21 C […] › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 1/00 Microorganismos, p.ej. protozoos; Composiciones que los contienen (preparaciones de uso médico que contienen material de protozoos, bacterias o virus A61K 35/66, de algas A61K 36/02, de hongos A61K 36/06; preparación de composiciones de uso médico que contienen antígenos o anticuerpos bacterianos, p. ej. vacunas bacterianas, A61K 39/00 ); Procesos de cultivo o conservación de microorganismos, o de composiciones que los contienen; Procesos de preparación o aislamiento de una composición que contiene un microorganismo; Sus medios de cultivo. › modificados por la introducción de material genético extraño.
- C12N15/74 C12N […] › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Vectores o sistemas de expresión especialmente adaptados a huéspedes procariotas distintos a E. coli, p. ej. Lactobacillus, Micromonospora.
- C12N9/22 C12N […] › C12N 9/00 Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas. › Ribonucleasas.
- C12P21/06 C12 […] › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 21/00 Preparación de péptidos o de proteínas (proteína monocelular C12N 1/00). › preparados por hidrólisis de un enlace peptídico, p. ej. hidrolizados.
- C12Q1/68 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2519444_T3.pdf



Fragmento de la descripción:
Incorporación in vivo de alquinil aminoácidos a proteínas en eubacterias Campo de la invención
La invención pertenece al campo de la bioquímica de la traducción. La invención se refiere a composiciones y a métodos para la preparación y uso de ARNt ortogonales, aminoacil-ARNt sintetasas ortogonales, y sus pares, que incorporan alquinil aminoácidos a proteínas. La invención también se refiere a métodos de producción de proteínas en células usando dichos pares y composiciones relacionadas.
Antecedentes de la invención
La capacidad para modificar químicamente proteínas en lugares específicos con moléculas no peptídicas tales como sondas espectroscópicas, adyuvantes catalíticos, o polímeros, o para entrecruzar covalentemente una proteína a otra proteína o cualquier otro resto, proporciona un poderoso medio tanto para investigar como para manipular las propiedades químicas y biológicas de las proteínas. Un enfoque habitual supone la bioconjugación de restos de superficie nucleófila sobre la proteína, por ejemplo, las cadenas laterales de lisina, histidina, o cisterna, con grupos electrófilos sobre una molécula exógena, tales como aldehidos, a-halo carboxamidas, y N-hidroxi succinimidas (Lemineux, G. A.; Bertozzi, C. R. TIBTECH 1996, 16, 56).
Desgraciadamente, un desafío al usar las dianas nucleófilas de origen natural en una proteína para modificaciones dirigidas es la poca selectividad de estas reacciones y la aparición de múltiples aminoácidos nucleófilos en proteínas, lo que da lugar a la formación de mezclas heterogéneas de proteínas marcadas. Además, las reacciones de modificación selectivas para nucleófilos con frecuencia requieren condiciones no fisiológicas, que pueden limitar las estrategias de modificación in vivo y/o producir la pérdida de actividad biológica de la proteína.
Existe una necesidad en la técnica de crear nuevas dianas y nuevas estrategias para las modificaciones específicas y dirigidas de proteínas. Desgraciadamente, cada organismo conocido, desde bacterias a seres humanos, codifica los mismos 2 aminoácidos habituales (con las excepciones raras de la selenocisteína (véase, por ejemplo, A. Bock y col., (1991), Molecular Microbiology 5:515-2) y la pirrolisina (véase, por ejemplo, G. Srinivasan, y col., (22), Science 296:1459-62). Esta característica limita el uso de aminoácidos naturales en el desarrollo de nuevas químicas para la modificación dirigida de proteínas.
Una estrategia para superar esta limitación es expandir el código genético y añadir aminoácidos que tengan propiedades químicas distintas a las del repertorio biológico. Este enfoque se ha demostrado factible con el uso de ARNt "ortogonales" y las nuevas aminoacil-ARNt sintetasas "ortogonales" correspondientes para añadir aminoácidos no naturales a proteínas que usan la maquinaria biosintética de proteínas in vivo de la eubacteria Escherichia coli (E. coii) y otros organismos (por ejemplo, Wang y col., (21), Science 292:498-5; Chin y col., (22) Journal of the American Chemical Society 124:926-927; Chin y Schultz, (22), ChemBioChem 11:1135-1137; Chin, y col., (22), PNAS United States of America 99:112-1124; y Wang y Schultz, (22), Chem. Comm., 1-1). Véanse también, las Publicaciones internacionales WO 22/8675, titulada "METHODS AND COMPOSITIONS FOR THE PRODUCTION OF ORTHOGONAL tRNA AMINOACYL-tRNA SYNTHETASE PAIRS"; WO 22/85923, titulada "IN VIVO INCORPORATION OF UNNATURAL AMINO ACIDS"; WO 24/94593, titulada "EXPANDING THE EUKARYOTIC GENETIC CODE"; WO 25/19415, presentada el 7 de julio de 24; WO 25/787, presentada el 7 de julio de 24; y WO 25/7624, presentada el 7 de julio de 24.
Existe una necesidad en la técnica de nuevos métodos para conseguir modificaciones de proteínas altamente específicas y dirigidas. Existe una necesidad en la técnica del desarrollo de componentes de traducción ortogonales que incorporen aminoácidos no naturales in vivo a proteínas en E. coli, en donde los aminoácidos no naturales se pueden incorporar en una posición definida, y en donde los aminoácidos no naturales tienen propiedades químicas distintas que les permiten servir como dianas para la modificación específica por la exclusión de reacciones cruzadas o reacciones secundarias con otras partes de las proteínas. Esta necesidad en la técnica es especialmente aplicable a E. coli, puesto que los sistemas de expresión de proteínas eubacterianas pueden producir grandes cantidades de material proteico recombinante para su estudio científico o aplicaciones terapéuticas. Esta invención satisface estas y otras necesidades, como será evidente tras la revisión de la siguiente divulgación.
El documento de Estados Unidos 23/18885 A1 describe composiciones y métodos para generar componentes de la maquinaria biosintética de proteínas que incluyen ARNt ortogonales, aminoacil-ARNt sintetasas ortogonales, y pares ortogonales de ARNt/sintetasas.
Wang y col., 21, Science, Vol. 292, pp. 498-5, describe un método de expansión del código genético de E. coli.
Kobayashi y col., 23, Nature Structural Biology, Vol. 1, No. 6, pp. 425-432, describe una base estructural para las especificidades del ARNt ortogonal de tirosil-ARNt sintetasas para la expansión del código genético.
El documento WO 2/85923 describe métodos y composiciones para la incorporación in vivo de aminoácidos no naturales.
Sumario de la invención
En un aspecto, la presente invención proporciona una célula eubacteriana que comprende una primera aminoacil- ARNt sintetasa ortogonal (O-RS) que actúa en la célula, en la que la O-RS preferentemente aminoacila un primer ARNt ortogonal (O-ARNt) con un primer aminoácido no natural que es un alquinil aminoácido; en el que el alquinil aminoácido es una tirosina para-sustituida o una fenilalanina para-sustituida, en la que la tirosina o la fenilalanina están sustituidas en la posición para con un grupo etinilo o propinilo; y
en el que la O-RS aminoacila el O-ARNt con el primer aminoácido no natural con una eficiencia de supresión de al menos el 5 % de la eficiencia de supresión de un par sintetasa ortogonal, par que es el O-ARNt de la SEQ ID NO: 1 y una polipéptido sintetasa que comprende una secuencia de aminoácidos seleccionada entre una cualquiera de las SEQ ID NO: 4, 6, 8, 1, 12, 14, 16, 18, 21,22, 23, 24 y 25.
En un aspecto adicional, la presente invención proporciona un método de producción de una proteína que comprende un alquinil aminoácido no natural en una célula eubacteriana, en el que el alquinil aminoácido está en una posición específica, el método que comprende:
(a) el suministro de una célula eubacteriana que comprende:
(i) , una aminoacil-ARNt sintetasa ortogonal (O-RS);
(ii). un ARNt ortogonal (O-ARNt),
(iii). en el que dicha O-RS preferentemente aminoacila dicho O-ARNt con dicho alquinil aminoácido con una eficiencia de supresión de al menos el 5 % de la eficiencia de supresión de un par sintetasa ortogonal, par que es el O-ARNt de la SEQ ID NO: 1 y una polipéptido sintetasa que comprende una secuencia de aminoácidos seleccionada entre una cualquiera de las SEQ ID NO: 4, 6, 8, 1, 12, 14, 16, 18, 21,22, 23, 24 y 25;
(iv). un ácido nucleico que codifica dicha proteína, en el que el ácido nucleico comprende al menos un codón selector que es reconocido por el O-ARNt; y
(v). dicho alquinil aminoácido, alquinil aminoácido que es una tirosina para-sustituida o una fenilalanina para-sustituida, en la que la tirosina o la fenilalanina están sustituidas en la posición para con un grupo etinilo o propinilo; y
(b) el crecimiento de dicha célula;
(c) la incorporación de dicho alquinil aminoácido a dicha posición específica en la proteína codificada por el ácido nucleico durante la traducción de la proteína, en el que la posición específica en la proteína corresponde a la posición del codón selector en dicho ácido nucleico, produciendo así dicha proteína que comprende dicho alquinil aminoácido en dicha posición específica.
De acuerdo con la presente invención, la célula eubacteriana puede ser una célula de E. coli.
De acuerdo con la presente invención:
(i) , la O-RS puede ser idéntica en al menos un 95 % a una aminoacil-ARNt sintetasa de Methanococcus jannaschii; o
(ii). la O-RS puede ser idéntica en al menos un 95 % a una tirosll-ARNt sintetasa de Methanococcus jannaschii; o
(iii). la O-RS puede ser idéntica en al menos un 95 % a la tirosil-ARNt sintetasa de tipo silvestre de Methanococcus jannaschii que tiene la secuencia de aminoácidos de la SEQ ID NO: 2; o
(iv). la O-RS puede ser idéntica en al menos un 95 % a la tirosil-ARNt sintetasa de tipo silvestre de Methanococcus... [Seguir leyendo]
Reivindicaciones:
1. Una célula eubacteriana que comprende una primera aminoacil-ARNt sintetasa ortogonal (O-RS) que actúa en la célula, en donde la O-RS preferentemente aminoacila un primer ARNt ortogonal (O-ARNt) con un primer aminoácido no natural que es un alquinil aminoácido;
en donde el alquinil aminoácido es una tirosina para-sustituida o una fenilalanina para-sustituida, en donde la tirosina o la fenilalanina están sustituidas en la posición para con un grupo etinilo o propinilo; y
en donde la O-RS aminoacila el O-ARNt con el primer aminoácido no natural con una eficiencia de supresión de al menos el 5 % de la eficiencia de supresión de un par sintetasa ortogonal, par que es el O-ARNt de la SEO ID NO: 1 y una polipéptido sintetasa que comprende una secuencia de aminoácidos seleccionada entre una cualquiera de las SEQ ID NO: 4, 6, 8, 1, 12, 14, 16, 18, 21,22, 23, 24 y 25.
2. Un método de producción de una proteína que comprende un alquinil aminoácido no natural en una célula eubacteriana, en donde el alquinil aminoácido está en una posición específica, comprendiendo el método:
(a) el suministro de una célula eubacteriana que comprende:
(i). una aminoacil-ARNt sintetasa ortogonal (O-RS);
(ii). un ARNt ortogonal (O-ARNt),
en donde dicha O-RS preferentemente aminoacila dicho O-ARNt con dicho alquinil aminoácido con una eficiencia de supresión de al menos el 5 % de la eficiencia de supresión de un par sintetasa ortogonal, par que es el O-ARNt de la SEQ ID NO: 1 y una polipéptido sintetasa que comprende una secuencia de aminoácidos seleccionada entre una cualquiera de las SEQ ID NO: 4, 6, 8, 1, 12, 14, 16, 18, 21,22, 23, 24 y 25;
(iii). un ácido nucleico que codifica dicha proteína, en donde el ácido nucleico comprende al menos un codón selector que es reconocido por el O-ARNt; y
(iv). dicho alquinil aminoácido, alquinil aminoácido que es una tirosina para-sustituida o una fenilalanina para-sustituida, en donde la tirosina o la fenilalanina están sustituidas en la posición para con un grupo etinilo o propinilo; y
(b) el cultivo de dicha célula;
(c) la incorporación de dicho alquinil aminoácido a dicha posición específica en la proteína codificada por el ácido nucleico durante la traducción de la proteína, en donde la posición específica en la proteína corresponde a la posición del codón selector en dicho ácido nucleico, produciendo así dicha proteína que comprende dicho alquinil aminoácido en dicha posición específica.
3. La célula de la reivindicación 1 o el método de la reivindicación 2, en donde célula eubacteriana es una célula de E. coli.
4. La célula de la reivindicación 1 o el método de la reivindicación 2, en donde:
(i) , la O-RS es idéntica en al menos un 95 % a una aminoacil-ARNt sintetasa de Methanococcus jannaschii', o
(ii). la O-RS es idéntica en al menos un 95 % a una tirosil-ARNt sintetasa de Methanococcus jannaschii', o
(iii). la O-RS es idéntica en al menos un 95 % a la tirosil-ARNt sintetasa de tipo silvestre de Methanococcus jannaschiique tiene la secuencia de aminoácidos de la SEQ ID NO: 2; o
(iv). la O-RS es idéntica en al menos un 95 % a la tirosil-ARNt sintetasa de tipo silvestre de Methanococcus jannaschii que tiene la secuencia de aminoácidos de la SEQ ID NO: 2, en donde la O-RS tiene una secuencia de aminoácidos que comprende:
(a) alanina, histidina, serina o treonina en una posición que corresponde a la posición del aminoácido 32 de la SEQ ID NO: 21;
(b) prolina, glutamina, lisina, arginina, serina o alanina en una posición que corresponde a la posición del aminoácido 17 de la SEQ ID NO: 21;
(c) alanina, leucina, prolina o serina en una posición que corresponde a la posición del aminoácido 158 de la SEQ ID NO: 21; y
(d) alanina, histidina, treonina o prolina en una posición que corresponde a la posición del aminoácido 162 de la SEQ ID NO: 21; o
(v). la O-RS es idéntica en al menos un 95 % a la tirosil-ARNt sintetasa de tipo silvestre de Methanococcus jannaschii que tiene la secuencia de aminoácidos de la SEQ ID NO: 2, en donde la O-RS tiene una secuencia de aminoácidos que comprende:
(a) alanina en una posición que corresponde a la posición del aminoácido 32 de la SEQ ID NO: 21;
(b) prolina o glutamina en una posición que corresponde a la posición del aminoácido 17 de la SEQ ID NO: 21;
(c) alanina en una posición que corresponde a la posición del aminoácido 158 de la SEQ ID NO: 21; y
(d) alanina o prolina en una posición que corresponde a la posición del aminoácido 162 de la SEQ ID NO: 21; o
(vi), la O-RS comprende una secuencia de aminoácidos seleccionada entre las SEQ ID NO: 4, 6, 8, 1, 12, 14, 16, 18, y sus variantes conservativas.
5. La célula de la reivindicación 1 o el método de la reivindicación 2, en donde la célula comprende un polinucleótido que codifica la O-RS, en donde la O-RS comprende una secuencia de aminoácidos seleccionada entre las SEQ ID NO: 4, 6, 8, 1,12, 14,16, 18, y sus variantes conservativas.
6. La célula o el método de la reivindicación 5, en donde el polinucleótido se selecciona entre las secuencias de nucleótidos de las SEQ ID NO: 5, 7, 9, 11, 13, 15, 17 o 19.
7. La célula de la reivindicación 1 o el método de la reivindicación 2, en donde:
(a) el O-ARNt es un ARNt supresor ámbar; o
(b) el O-ARNt comprende o está codificado por una secuencia de polinucleótidos expuesta en la SEQ ID NO: 1.
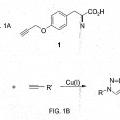
8. La célula de la reivindicación 1 o el método de la reivindicación 2, en donde dicho alquinil aminoácido es para- propargiloxifenilalanina.
9. La célula de la reivindicación 1 o el método de la reivindicación 2, que comprende al menos un codón selector, en donde dicho codón selector es reconocido por dicho primer O-ARNt.
1. La célula o el método de la reivindicación 9, que comprende una segunda O-RS y un segundo O-ARNt, en donde la segunda O-RS preferentemente aminoacila el segundo O-ARNt con un segundo aminoácido no natural que es diferente del primer aminoácido no natural, y en donde el segundo O-ARNt reconoce un codón selector que es diferente del codón selector reconocido por el primer O-ARNt.
11. La célula de la reivindicación 1 que comprende dicho alquinil aminoácido.
12. La célula de la reivindicación 11 en la que dicho alquinil aminoácido es para-propargiloxifenilalanina.
13. La célula de la reivindicación 1 que comprende un sistema de traducción.
14. La célula de la reivindicación 13, comprendiendo dicho sistema de traducción:
(a) dicha O-RS;
(b) dicho O-ARNt;
(c) un ácido nucleico que codifica un polipéptido de interés, comprendiendo el ácido nucleico al menos un codón selector, en donde el codón selector es reconocido por dicho O-ARNt; y
(d) un alquinil aminoácido, en donde dicha O-RS es capaz de cargar dicho O-ARNt con dicho alquinil aminoácido.
15. El método de la reivindicación 2, en el que dicha proteína comprende una proteína terapéutica de tipo silvestre, una proteína diagnóstica, una enzima industrial, o un fragmento de las mismas.
16. El método de la reivindicación 15, en el que, habiendo obtenido dicha proteína, la proteína se formula en una composición con un vehículo farmacéuticamente aceptable.
17. El método de la reivindicación 2, en el que dicha proteína se modifica en dicha posición específica.
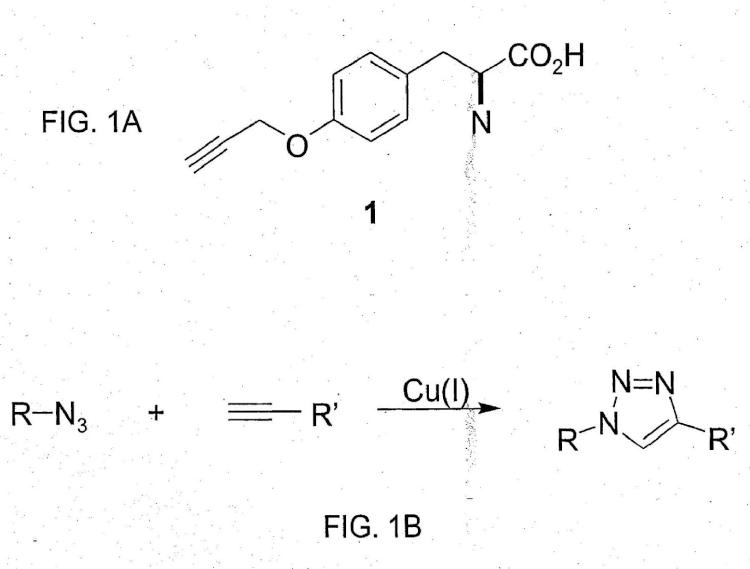
18. El método de la reivindicación 17, en el que dicha proteína comprende una unión triazol en dicha posición específica.
19. Un polipéptido que es al menos idéntico en un 9 % a la tirosil aminoacil-ARNt sintetasa de Methanococcus jannaschiide la SEQ ID NO: 2, en donde el polipéptido tiene una secuencia de aminoácidos que comprende:
(a) alanina, histidina, serina o treonina en una posición que corresponde a la posición del aminoácido 32 de la SEQ ID NO: 21;
(b) prolina, glutamina, lisina, arginina, serina o alanina en una posición que corresponde a la posición del aminoácido 17 de la SEQ ID NO: 21;
(c) alanina, leucina, prolina o serina en una posición que corresponde a la posición del aminoácido 158 de la SEQ ID NO: 21; y
(d) alanina, histidina, treonina o prolina en una posición que corresponde a la posición del aminoácido 162 de la
SEQ ID NO: 21;
y en donde el polipéptido es una aminoacil-ARNt sintetasa capaz de aminoacilar preferentemente un ARNt ortogonal (O-ARNt) con un alquinil aminoácido, alquinil aminoácido que es una tirosina para-sustituida o una fenilalanina para-sustituida, en donde la tirosina o la fenilalanina están sustituidas en la posición para con un grupo etinilo o propinilo; y
en donde la O-RS aminoacila el O-ARNt con el primer aminoácido no natural con una eficiencia de supresión de al menos el 5 % de la eficiencia de supresión de un par sintetasa ortogonal, par que es el O-ARNt de la SEQ ID NO: 1 y una polipéptido sintetasa que comprende una secuencia de aminoácidos seleccionada entre una cualquiera de las SEQ ID NO: 4, 6, 8, 1, 12,14, 16,18, 21,22, 23, 24 y 25.
2. El polipéptido de la reivindicación 19, que tiene una secuencia de aminoácidos que comprende:
(a) alanina en una posición que corresponde a la posición del aminoácido 32 de la SEQ ID NO: 21;
(b) prolina o glutamina en una posición que corresponde a la posición del aminoácido 17 de la SEQ ID NO: 21;
(c) alanina en una posición que corresponde a la posición del aminoácido 158 de la SEQ ID NO: 21; y
(d) alanina en una posición que corresponde a la posición del aminoácido 162 de la SEQ ID NO: 21.
21. Un polipéptido que comprende una secuencia de aminoácidos expuesta en las SEQ ID NO: 4, 6, 8, 1, 12, 14,
16, 18, o una de sus variantes conservativas, en donde dicho polipéptido es una aminoacil-ARNt sintetasa capaz de aminoacilar preferentemente un ARNt ortogonal (O-ARNt) en una célula eubacteriana con un alquinil aminoácido, alquinil aminoácido que es una tirosina que está sustituida en posición para con un grupo etinilo o propinilo, o el alquinil aminoácido es una fenilalanina que está sustituida en posición para con un grupo etinilo o propinilo; y
en donde la O-RS aminoacila el O-ARNt con el primer aminoácido no natural con una eficiencia de supresión de al menos el 5 % de la eficiencia de supresión de un par sintetasa ortogonal, par que es el O-ARNt de la SEQ ID NO: 1 y una polipéptido sintetasa que comprende una secuencia de aminoácidos seleccionada entre una cualquiera de las SEQ ID NO: 4, 6, 8, 1, 12, 14, 16, 18, 21,22, 23, 24 y 25.
22. Un polinucleótido que codifica un polipéptido de una cualquiera de las reivindicaciones 19 a 21.
23. El polinucleótido de la reivindicación 22, en donde el polinucleótido se selecciona entre las SEQ ID NO: 5, 7, 9, 11, 13, 15, 17 y 19.
24. Un vector que comprende un polinucleótido de la reivindicación 22.
25. El vector de la reivindicación 24, en donde el vector es un vector de expresión.
26. Una célula que comprende un vector de la reivindicación 24.
Patentes similares o relacionadas:
Sistema de expresión bacteriano cistrónico doble, del 25 de Marzo de 2020, de LUPIN LIMITED: Un procedimiento para la producción de un anticuerpo o un fragmento del mismo que comprende las etapas de: (i) transformar una célula hospedadora bacteriana […]
Partículas de transducción no replicativas y sistemas indicadores basados en partículas de transducción, del 15 de Enero de 2020, de Geneweave Biosciences Inc: Un sistema de empaquetamiento de células bacterianas para empaquetar una molécula de ácido nucleico indicadora en una partícula de transducción no replicativa (NRTP) para su introducción […]
Método de construcción de biblioteca de péptidos y vectores relacionados, del 1 de Enero de 2020, de Hunan Zonsen Peplib Biotech Co., Ltd: Método integrado de construcción de una biblioteca de péptidos completa que contiene todos los péptidos posibles para el tamaño específico, comprendiendo el método: (i) […]
 Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Producción recombinante de péptidos, del 25 de Septiembre de 2019, de BASF SE: Proteina precursora que comprende una secuencia repetitiva escindible de elementos de peptido (Pep) deseados y elementos de peptido auxiliar […]
Transporte de proteínas basado en bacterias, del 14 de Agosto de 2019, de Universität Basel: Un vector que comprende, en la dirección 5' a 3': un promotor; una primera secuencia de ADN que codifica una señal de transporte procedente […]
Sistema de expresión y secreción, del 26 de Junio de 2019, de F. HOFFMANN-LA ROCHE AG: Una molécula de ácido nucleico que codifica un primer polipéptido y un segundo polipéptido, enlazada de forma funcional a una secuencia señal que codifica […]
 Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Producción de anticuerpos híbridos que contienen regiones variables humanas y regiones constantes de roedor, del 26 de Junio de 2019, de REGENERON PHARMACEUTICALS, INC.: Un procedimiento para modificar genéticamente un locus génico endógeno de la región variable de cadena pesada de inmunoglobulina en una célula madre embrionaria (ES) de […]
Síntesis microbiana de aldehídos y alcoholes correspondientes, del 17 de Junio de 2019, de Easel Biotechnologies, LLC: Un método para producir un alcohol, que comprende: cultivar una pluralidad de células microbianas en un medio de fermentación, en donde las […]