Fosfolipasas, ácidos nucleicos que las codifican, y métodos para obtenerlas y usarlas.
Un ácido nucleico aislado, sintético o recombinante que comprende una secuencia de ácido nucleico:
(a) que codifica un polipéptido que tiene actividad de fosfolipasa, y
(i) que tiene al menos 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%,94%, 95%, 96%, 97%, 98%, 99%, o más, o 100% de una identidad de secuencia con SEC ID NO:177,y que tiene una mutación que codifica E41W, E41F o E41Y,
(ii) se hibrida en condiciones restrictivas al complemento de un ácido nucleico que comprende SEC IDNO:177, y que tiene una mutación que codifica E41W, E41F, o E41Y,
en el que las condiciones restrictivas incluyen una etapa de lavado que comprende un lavado en 0,2XSSC a una temperatura de alrededor de 65ºC durante alrededor de 15 minutos;
(b) la secuencia de ácido nucleico de (a) que codifica un polipéptido que tiene actividad de fosfolipasa peroque carece de: una secuencia señal o secuencia proproteínica, o una secuencia promotora homóloga;
(c) el ácido nucleico de (a) o (b) que codifica un polipéptido que tiene una actividad de fosfolipasa y quecodifica adicionalmente una secuencia de aminoácidos, o el ácido nucleico de (a) o (b) que comprende unasecuencia nucleotídica heteróloga;
(d) el ácido nucleico de (c), en el que la secuencia de aminoácidos heteróloga comprende, o consiste, en unasecuencia que codifica una secuencia señal heteróloga, o una etiqueta o un epítopo, o la secuencianucleotídica heteróloga comprende una secuencia promotora heteróloga;
(e) el ácido nucleico aislado, sintético o recombinante de uno cualquiera de (a) a (d), en el que los restosnucleotídicos en un sitio de comienzo transcripcional críptico se modifican para eliminar la mayoría o toda laproducción de un transcrito truncado;
(f) el ácido nucleico aislado, sintético o recombinante de (e), en el que las modificaciones de restosnucleotídicos en el sitio de comienzo transcripcional críptico comprenden una alteración en un sitio de uniónal ribosoma (RBS); o
(g) una secuencia de ácido nucleico completamente complementaria a la secuencia nucleotídica decualquiera de (a) a (f).
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2007/079109.
Solicitante: DSM IP ASSETS B.V..
Nacionalidad solicitante: Países Bajos.
Dirección: HET OVERLOON 1 6411 TE HEERLEN PAISES BAJOS.
Inventor/es: O'DONOGHUE,EILEEN, BARTON,NELSON R.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12P7/64 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12P PROCESOS DE FERMENTACION O PROCESOS QUE UTILIZAN ENZIMAS PARA LA SINTESIS DE UN COMPUESTO QUIMICO DADO O DE UNA COMPOSICION DADA, O PARA LA SEPARACION DE ISOMEROS OPTICOS A PARTIR DE UNA MEZCLA RACEMICA. › C12P 7/00 Preparación de compuestos orgánicos que contienen oxígeno. › Grasas; Aceites; Ceras de tipo éster; Acidos grasos superiores, es decir, con una cadena lineal de al menos siete átomos de carbono unida a un grupo carboxilo; Aceites o grasas oxidadas.
PDF original: ES-2451266_T3.pdf
Fragmento de la descripción:
Fosfolipasas, ácidos nucleicos que las codifican, y métodos para obtenerlas y usarlas REFERENCIA AL LISTADO DE SECUENCIAS PRESENTADO VÍA EFS-WEB
Esta solicitud se presentó electrónicamente vía el servidor de USPTO EFS-WEB, como se autoriza y se expone en MPEP §1730 II.B.2. (a) (A) , y esta presentación electrónica incluye un listado de secuencias (SEC ID) presentado electrónicamente. El listado de secuencias se identifica en el archivo .txt presentado electrónicamente como sigue:
Nombre del archivo Fecha de creación Tamaño
564462015940seqlist.txt 20 de septiembre de 2007 422.053 bites
CAMPO DE LA INVENCIÓN
Esta invención se refiere generalmente a enzimas fosfolipasas, a polinucleótidos que codifican las enzimas, a métodos para obtener y usar estos polinucleótidos y polipéptidos. En particular, la invención proporciona nuevos polipéptidos que tienen actividad de fosfolipasa, ácidos nucleicos que los codifican y anticuerpos que se unen a ellos. También se proporcionan métodos industriales y productos que comprenden el uso de estas fosfolipasas.
ANTECEDENTES
Las fosfolipasas son enzimas que hidrolizan los enlaces de éster de los fosfolípidos. Correspondiente a su importancia en el metabolismo de fosfolípidos, estas enzimas están ampliamente extendidas entre procariotas y eucariotas. Las fosfolipasas afectan al metabolismo, construcción y reorganización de las membranas biológicas, y están implicadas en las cascadas de señales. Se conocen varios tipos de fosfolipasas que difieren en su especificidad según la posición del enlace atacada en la molécula fosfolipídica. La fosfolipasa A1 (PLA1) elimina el ácido graso de la posición 1 para producir ácido graso libre y 1-liso-2-acilfosfolípido. La fosfolipasa A2 (PLA2) elimina el ácido graso de la posición 2 para producir ácido graso libre y 1-acil-2-lisofosfolípido. Las enzimas PLA1 y PLA2 pueden ser intra- o extracelulares, unidas a la membrana o solubles. PLA2 intracelular se encuentra en casi cualquier célula de mamífero. La fosfolipasa C (PLC) elimina el resto de fosfato para producir 1, 2-diacilglicerol y una éster de fosfato. La fosfolipasa D (PLD) produce 1, 2-diacilglicerofosfato y un grupo base. PLC y PLD son importantes en la función celular y en la señalización. PLD ha sido la fosfolipasa dominante en la biocatálisis (véase, por ejemplo, Godfrey, T. y West S. (1996) Industrial enzymology, 299-300, Stockton Press, Nueva York) . Las patatinas son otro tipo de fosfolipasa, que se piensa que trabajan como una PLA (véase, por ejemplo, Hirschberg HJ, et al., (2001) , Eur J Biochem 268 (19) :5037-44) .
Las oleaginosas habituales, tales como habas de soja, colza, semillas de girasol, aceite de salvado de arroz, sésamo y cacahuetes, se usan como fuentes de aceites y materias primas. En el proceso de extracción del aceite, las semillas se tratan mecánica y térmicamente. El aceite se separa y se divide de la harina mediante un disolvente. Usando destilación, el disolvente se separa entonces del aceite y se recupera. El aceite se “desgoma” y se refina. El contenido de disolvente en la harina se puede evaporar mediante tratamiento térmico en una “tostadora desolventizadora”, seguido del secado y enfriamiento de la harina. Después de que el disolvente se ha separado por destilación, el aceite bruto producido se procesa en aceite comestible, usando procedimientos de desgomado especiales y refinado físico. También se puede utilizar como materia prima para la producción de ácidos grasos y éster metílico. La harina se puede usar para raciones para animales.
El desgomado es la primera etapa en el refinado del aceite vegetal, y se diseña para eliminar fosfátidos contaminantes que se extraen con el aceite pero que interfieren con el procesamiento subsiguiente del aceite. Estos fosfátidos son solubles en el aceite vegetal solamente en una forma anhidra, y se pueden precipitar y eliminar si se hidratan simplemente. La hidratación se logra habitualmente mezclando una pequeña proporción de agua continuamente con aceite sustancialmente seco. Típicamente, la cantidad de agua es 75% del contenido de fosfátidos, que es típicamente 1 a 1, 5%. La temperatura no es muy crítica, aunque la separación de las gomas hidratadas es mejor si la viscosidad del aceite se reduce a 50ºC hasta 80ºC.
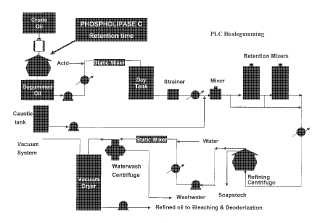
Actualmente se usan muchos métodos para el desgomado del aceite. El procedimiento de desgomado del aceite se puede ayudar enzimáticamente usando enzimas fosfolipasas. Las fosfolipasas A1 y A2 se han usado para el desgomado de aceites en diversos procedimientos comerciales, por ejemplo “desgomado ENZYMAX™” (Lurgi Life Science Technologies GmbH, Alemania) . La fosfolipasa C (PLC) también se ha considerado para el desgomado de aceites debido a que el resto de fosfato generado por su acción sobre los fosfolípidos es muy soluble en agua y fácil de eliminar, y el diglicérido permanecería con el aceite y reduce pérdidas; véanse, por ejemplo, Godfrey, T. y West
S. (1996) Industrial Enzymology, p. 299-300, Stockton Press, Nueva York; Dahlke (1998) “An enzymatic process for the physical refining of seed oils”, Chem. Eng. Technol. 21:278-281; Clausen (2001) “Enzymatic oil degumming by a novel microbial phospholipase”, Eur. J. Lipid Sci. Technol. 103:333-340.
5
Los aceites con contenido elevado de fosfátidos, tales como soja, cánola y girasol, se procesan de forma diferente a otros aceites tales como el de palma. A diferencia del procedimiento de vapor o “refinado físico” para aceites con bajo contenido de fosfátidos, estos aceites con un contenido elevado de fósforo requieren tratamientos químicos y mecánicos especiales para eliminar los fosfolípidos que contienen fósforo. Estos aceites se refinan típicamente de forma química en un procedimiento que supone neutralizar los ácidos grasos libres para formar jabón y una fracción de goma insoluble. El proceso de neutralización es muy eficaz a la hora de eliminar los ácidos grasos libres y los fosfolípidos, pero este proceso también da como resultado pérdidas significativas de rendimiento y sacrificios en la calidad. En algunos casos, el aceite bruto con contenido elevado de fosfátidos se desgoma en una etapa anterior a la neutralización cáustica. Este es el caso para aceite de soja utilizado para lecitina, en el que el aceite se desgoma en primer lugar con agua o ácido.
El aceite de haba de soja se usa ampliamente y es una materia alimentaria importante, dando cuenta de !30% de la producción de aceite a partir de semillas y frutas. Las habas de soja contienen sólo 20% de aceite, y la extracción se realiza habitualmente usando un disolvente tal como hexano a escala comercial. La calidad reconocida de su aceite y el valor nutritivo de la proteína de la harina hacen al haba de soja una oleaginosa principal. Antes de la extracción, las habas de soja se deben limpiar, romper y convertir en copos ya que la extracción eficiente del aceite con disolventes requiere que cada célula de aceite se rompa para mejorar la transferencia de masa. Las paredes celulares compuestas mayoritariamente de celulosa, asociada con hemicelulosas, sustancias pécticas y lignina) , también se podrían romper por medios enzimáticos, para lograr una mejora significativa en los rendimientos y velocidades de extracción.
El aceite de diacilglicerol (DAG) es un aceite comestible que contiene una cantidad de 80% o mayor de DAG que los ácidos grasos naturales. Se ha demostrado en seres humanos que la elevación postprandial de triglicérido en quilomicrones es notablemente menor tras la ingestión de una emulsión de aceite de DAG en comparación con un aceite de TAG con una composición de ácidos grasos similar. En estudios que usan hombres japoneses y hombres y mujeres americanos, el consumo a largo plazo de aceite de DAG promovió la pérdida de peso y la reducción de la grasa corporal. Un estudio mostró que la sustitución del aceite habitual de cocinar por aceite de DAG reduce la incidencia de obesidad y otros factores de riesgo.
SUMARIO DE LA INVENCIÓN
La invención proporciona ácidos nucleicos aislados, sintéticos o recombinantes que comprenden una secuencia de ácido nucleico que tiene al menos 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, o más, o una identidad de secuencia completa (100%) con SEC ID NO:177, y que tiene una mutación E41W, E41F o E41Y y preferiblemente una o más mutaciones que codifican E94R, D100L, D100M, D100Y, D100F, D100W, A104L, D111R, T112R, Y116W, I117W, P118W, E125K, S168N, D171V, D171E, M176W, D230H, D230R, D234W, D234V, D234G, D234R, D234K, o Q265R a lo largo de una región de al menos... [Seguir leyendo]
Reivindicaciones:
1. Un ácido nucleico aislado, sintético o recombinante que comprende una secuencia de ácido nucleico:
(a) que codifica un polipéptido que tiene actividad de fosfolipasa, y
(i) que tiene al menos 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, o más, o 100% de una identidad de secuencia con SEC ID NO:177, y que tiene una mutación que codifica E41W, E41F o E41Y,
(ii) se hibrida en condiciones restrictivas al complemento de un ácido nucleico que comprende SEC ID NO:177, y que tiene una mutación que codifica E41W, E41F, o E41Y,
en el que las condiciones restrictivas incluyen una etapa de lavado que comprende un lavado en 0, 2X SSC a una temperatura de alrededor de 65ºC durante alrededor de 15 minutos;
(b) la secuencia de ácido nucleico de (a) que codifica un polipéptido que tiene actividad de fosfolipasa pero que carece de: una secuencia señal o secuencia proproteínica, o una secuencia promotora homóloga;
(c) el ácido nucleico de (a) o (b) que codifica un polipéptido que tiene una actividad de fosfolipasa y que codifica adicionalmente una secuencia de aminoácidos, o el ácido nucleico de (a) o (b) que comprende una secuencia nucleotídica heteróloga;
(d) el ácido nucleico de (c) , en el que la secuencia de aminoácidos heteróloga comprende, o consiste, en una secuencia que codifica una secuencia señal heteróloga, o una etiqueta o un epítopo, o la secuencia nucleotídica heteróloga comprende una secuencia promotora heteróloga;
(e) el ácido nucleico aislado, sintético o recombinante de uno cualquiera de (a) a (d) , en el que los restos nucleotídicos en un sitio de comienzo transcripcional críptico se modifican para eliminar la mayoría o toda la producción de un transcrito truncado;
(f) el ácido nucleico aislado, sintético o recombinante de (e) , en el que las modificaciones de restos nucleotídicos en el sitio de comienzo transcripcional críptico comprenden una alteración en un sitio de unión al ribosoma (RBS) ; o
(g) una secuencia de ácido nucleico completamente complementaria a la secuencia nucleotídica de cualquiera de (a) a (f) .
2. El ácido nucleico aislado, sintético o recombinante de la reivindicación 1, en el que el ácido nucleico comprende una o más mutaciones que codifican E94R, D100L, D100M , D100Y, D100F, D100W, A104L, D111R, T112R, Y116W, I117W, P118W, E125K, S168N, D171V, D171E, M176W, D230H, D230R, D234W, D234V, D234G, D234R, D234K y/o Q265R o una combinación de las mismas.
3. Un vector, casete de expresión o vehículo de clonación: (a) que comprende la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2; o, (b) el vector, casete de expresión o vehículo de clonación de (a) que comprende o contenido en vector vírico, un plásmido, un fago, un fagómido, un cósmido, un fósmido, un bacteriófago, un cromosoma artificial, un vector adenovírico, un vector retrovírico o un vector vírico adenoasociado, o un cromosoma artificial bacteriano (BAC) , un plásmido, un vector derivado del bacteriófago P1 (PAC) , un cromosoma artificial de levadura (YAC) , o un cromosoma artificial de mamífero (MAC) .
4. Una célula hospedante o una célula transformada: (a) que comprende la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2, o el vector, casete de expresión o vehículo de clonación de la reivindicación 3; o,
(b) la célula hospedante o una célula transformada de (a) , en el que la célula es una célula bacteriana, una célula de mamífero, una célula fúngica, una célula de levadura, una célula de insecto o una célula vegetal.
5. Un animal no humano transgénico: (a) que comprende la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2; el vector, casete de expresión o vehículo de clonación de la reivindicación 3; o la célula hospedante o una célula transformada de la reivindicación 4; o (b) el animal no humano transgénico de (a) , en el que el animal es un ratón, una rata, una cabra, un conejo, una oveja, un cerdo o una vaca.
6. Una planta o semilla transgénica: (a) que comprende la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2; el vector, casete de expresión o vehículo de clonación de la reivindicación 3; o la célula hospedante o una célula transformada de la reivindicación 4; (b) la planta transgénica de (a) , en el que la planta es una planta de maíz, una planta de sorgo, una planta de patata, una planta de tomate, una planta de trigo, una planta oleaginosa, una planta de colza, una planta de haba de soja, una planta de arroz, una planta de cebada, un pasto, una semilla de algodón, una palma, una planta de sésamo, una planta de cacahuete, una planta de girasol o una planta de tabaco; (c) la semilla transgénica de (a) , en el que la semilla transgénica es una semilla de maíz, una pepita de trigo, una semilla oleaginosa, una colza, una semilla de haba de soja, una pepita de palma, una semilla de girasol, una semilla de sésamo, un arroz, una cebada, un cacahuete, una semilla de algodón, una palma, un cacahuete, una semilla de sésamo, una semilla de girasol, o una semilla de planta de tabaco.
7. Un polipéptido aislado, sintético o recombinante que tiene actividad de fosfolipasa, y:
(a) que comprende una secuencia de aminoácidos (i) que tiene al menos 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, o más, o 100% de identidad de secuencia con SEC ID NO:176, y que tiene al menos una de las mutaciones de aminoácidos de E41W, E41F o E41Y, en el que las identidades de secuencia se pueden determinar mediante análisis con un algoritmo de comparación de secuencias, o mediante inspección visual; o, (ii) codificado por el ácido nucleico de la reivindicación 1;
(b) que comprende el polipéptido de (a) pero que carece de una secuencia señal o secuencia proproteínica;
(c) que comprende además una secuencia de aminoácidos heteróloga;
(d) que comprende el polipéptido de (c) , en el que la secuencia de aminoácidos heteróloga comprende, o consiste, en una secuencia señal heteróloga, o una etiqueta o un epítopo;
(e) (i) que comprende el polipéptido de uno de (a) a (d) , en el que el polipéptido está glucosilado, o el polipéptido comprende al menos un sitio de glucosilación, (ii) el polipéptido de (i) , en el que la glucosilación es una glucosilación enlazada mediante N o una glucosilación enlazada mediante O; (iii) el polipéptido de (i) o (ii) , en el que el polipéptido se glucosila después de ser expresado en una célula de levadura; o (iv) el polipéptido de (iii) , en el que la célula de levadura es una P. pastoris o una S. pombe; o
(f) el polipéptido de uno cualquiera de (a) a (e) , en el que (i) el polipéptido comprende además restos de aminoácidos adicionales entre una secuencia señal y la enzima, o (ii) el polipéptido de (i) , en el que los restos de aminoácidos adicionales comprenden Glu-Ala.
8. El polipéptido aislado, sintético o recombinante de la reivindicación 7, en el que el polipéptido comprende una o más mutaciones E94R, D100L, D100M , D100Y, D100F, D100W, A104L, D111R, T112R, Y116W, I117W, P118W, E125K, S168N, D171V, D171E, M176W, D230H, D230R, D234W, D234V, D234G, D234R, D234K y/o Q265R, o una combinación de las mismas.
9. El polipéptido aislado, sintético o recombinante de la reivindicación 7, que comprende el aminoácido de SEC ID NO: 176 y que tiene las siguientes mutaciones: E41Y, D100F, S168N, D171N y M176W.
10. Un método para producir un polipéptido recombinante, que comprende:
(A) (a) proporcionar un ácido nucleico, en el que el ácido nucleico comprende la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2; y (b) expresar el ácido nucleico de la etapa (a) en condiciones que permiten la expresión del polipéptido, produciendo de ese modo un polipéptido recombinante; o
(B) el método de (A) , que comprende además transformar una célula hospedante con el ácido nucleico de la etapa (a) , seguido de la expresión del ácido nucleico de la etapa (a) , produciendo de ese modo un polipéptido recombinante en una célula transformada.
11. Un método para generar una variante de un ácido nucleico que codifica un polipéptido con actividad de fosfolipasa, que comprende:
(A)
(a) proporcionar un ácido nucleico molde que comprende la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2; y
(b) modificar, suprimir o añadir uno o más nucleótidos en la secuencia molde, o una combinación de los mismos, para generar una variante del ácido nucleico molde.
(B) el método de (A) , que comprende además expresar el ácido nucleico variante para generar un polipéptido de fosfolipasa variante;
(C) el método de cualquiera de (A) o (B) , en el que el método se repite iterativamente hasta que se produce una fosfolipasa que tiene una actividad alterada o diferente, o una estabilidad alterada o diferente de la de un polipéptido codificado por el ácido nucleico molde, o se produce una estructura secundaria alterada o diferente de la de un polipéptido codificado por el ácido nucleico molde, o se produce una modificación posttraduccional alterada o diferente de la de un polipéptido codificado por el ácido nucleico molde, o se produce un uso de codones alterado o diferente del de un polipéptido codificado por el ácido nucleico molde;
(D) el método de (A) , (B) o (C) , en el que el polipéptido de fosfolipasa variante tiene mayor glucosilación en comparación con la fosfolipasa codificada por un ácido nucleico molde; o
(E) el método de (A) , (B) o (C) , en el que el polipéptido de fosfolipasa variante tiene mayor expresión en comparación con la fosfolipasa codificada por un ácido nucleico molde.
12. Un método para modificar codones en un ácido nucleico que codifica un polipéptido de fosfolipasa, comprendiendo el método:
(a) proporcionar un ácido nucleico que codifica un polipéptido con actividad de fosfolipasa que comprende la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2; y
(b) identificar un codón en el ácido nucleico de la etapa (a) y sustituirlo por un codón diferente que codifica el mismo aminoácido que el codón sustituido, modificando de ese modo codones en un ácido nucleico que codifica una fosfolipasa.
13. Un método para hidrolizar, romper, licuar o disgregar una composición que comprende fosfolípidos, que comprende:
(A)
(a) proporcionar el polipéptido de cualquiera de las reivindicaciones 7 a 9, o el polipéptido que tiene actividad de fosfolipasa codificado por la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2;
(b) proporcionar una composición que comprende un fosfolípido; y
(c) poner en contacto el polipéptido de la etapa (a) con la composición de la etapa (b) en condiciones en las que el polipéptido hidroliza, rompe, licua o disgrega la composición que comprende fosfolípidos.
(B) el método de (A) , en el que la composición comprende una bicapa lipídica o membrana que comprende fsofolípido; o
(C) el método de cualquiera de (A) o (B) , en el que la composición comprende una célula vegetal, una célula bacteriana, una célula de levadura, una célula de insecto, o una célula de animal.
14. Una composición o un producto de fabricación (a) que comprende el polipéptido de cualquiera de las reivindicaciones 7 a 9, o el polipéptido que tiene actividad de fosfolipasa codificado por la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2, (b) la composición de (a) , en la que la composición una sustancia farmacéutica, un detergente, una biomasa, un biocombustible, un producto a base de petróleo, un soluble desecado de destilería (DDS) , un grano desecado de destilería (DDG) , un soluble de destilería condensado (CDS) , un grano húmedo de destilería (DWG) , o un grano desecado de destilería con solubles (DDGS) .
15. Un método para desgomar un aceite o una grasa, que comprende:
(A)
(a) proporcionar un polipéptido de cualquiera de las reivindicaciones 7 a 9, o el polipéptido que tiene actividad de fosfolipasa codificado por la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2;
(b) proporcionar una composición que comprende una grasa o un aceite; y
(c) poner en contacto el polipéptido de la etapa (a) y la composición de la etapa (b) ;
(B) el método de (A) , en el que el polipéptido (a) está unido a un filtro, y la grasa o aceite se hace pasar a través del filtro; o (b) se añade a una disolución que comprende la grasa o aceite, y después la disolución se hace pasar a través de un filtro;
(C) el método de (A) o (B) , en el que el polipéptido incrementa (a) la cantidad de aceites neutros en la composición tratada que comprende una grasa o aceite; o (b) la producción de diacilglicerol (DAG) , para contribuir a la fase oleosa, o en el que el producto de aceite desgomado final está enriquecido en 1, 3-DAG, o en el que el producto de grasa o aceite desgomado final comprende no menos de 1, 0% de 1, 3-DAG;
(D) el método de cualquiera de (A) a (C) , que comprende además la adición de uno o más polipéptidos que tienen una proteasa, una amilasa, una lipasa, una cutinasa, otra fosfolipasa, una carbohidrasa, una celulasa, una pectinasa, una mananasa, una arabinasa, una galactanasa, una xilanasa, una oxidasa, por ejemplo una lactasa, y/o una peroxidasa, o polipéptidos con actividad equivalente, o una combinación de las mismas, para romper adicionalmente la masa de goma y potenciar los rendimientos de aceite; o
(E) el método de cualquiera de (A) a (D) , que comprende además la eliminación física de la goma producida por el método de desgomado mediante adición de una sustancia endurecedora, en el que la sustancia endurecedora es o comprende un talco.
16. Un procedimiento según la reivindicación 15, que comprende
(F) el método de (A) en el que, en la etapa (c) , el polipéptido de la etapa (a) y la composición de la etapa (b) se ponen en contacto en condiciones en las que el polipéptido puede catalizar la hidrólisis de un fosfolípido en la composición durante un tiempo suficente para reducir la masa de goma e incrementar los aceites neutros; or
(G) el método de (F) , que comprende usar mezclamiento de cizallamiento elevado de la composición, seguido del mezclamiento sin cizallamiento o con poco cizallamiento con el polipéptido de (a) para permitir la puesto en contacto adecuada del fosfolípidos con el polipéptido.
17. Un método para convertir un fosfolípido no hidratable en una forma hidratable, que comprende:
(a) proporcionar un polipéptido de cualquiera de las reivindicaciones 7 a 9, o el polipéptido que tiene actividad de fosfolipasa codificado por la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2;
(b) proporcionar una composición que comprende un fosfolípido no hidratable; y
(c) poner en contacto el polipéptido de la etapa (a) y la composición de la etapa (b) en condiciones en las que el polipéptido convierte el fosfolípido no hidratable en una forma hidratable.
18. Un método para refinar un aceite o grasa, que comprende:
(A)
(a) proporcionar un polipéptido de cualquiera de las reivindicaciones 7 a 9, o el polipéptido que tiene actividad de fosfolipasa codificado por la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2;
(b) proporcionar una composición que comprende un aceite o una grasa; y
(c) poner en contacto el polipéptido de la etapa (a) con la composición de la etapa (b) ;
(B) el método de (A) , en el que el refinado es un refinado físico;
(C) el método de (A) , en el que el refinado es un refinado caústico;
(D) el método de (A) , (B) , o (C) , en el que la composición de (b) se añade antes, durante o después del refinado;
(E) el método de cualquiera de (A) a (D) , que comprende la adciión de un ácido, una sustancia cáustica, un emulsionante o un desemulsionante;
(F) el método de cualquiera de (A) a (E) , en el que el polipéptido se añade (a) durante el refinado caústico, y se añaden niveles variables de ácido o sustancia cáustica, dependiendo de los niveles de fósforo y de los niveles de ácidos grasos libres; (b) tras el refinado caústico: en una mezcladora intensa o mezcladora de retención antes de la separación; tras una etapa de calentamiento; en una centrifugadora; en una materia prima para jabón; en un agua de lavado; o durante las etapas de blanqueamiento o desodorización; (c) durante la trituración de una semilla u otra parte de la planta, o el polipéptido se añade tras la trituración o antes del refinado; o
(G) el método de cualquiera de (A) a (F) , en el que el polipéptido es una disolución acuosa que se añade a la composición, en el que opcionalmente el nivel de agua está entre alrededor de 0, 5 y 5%;
(H) el método de cualquiera de (A) a (G) , que comprende la adición de calor o enfriamiento para promover la separación de una fase acuosa; o
(I) el método de cualquiera de (A) a (H) , que comprende desgomar antes de la etapa de puesta en contacto, para recoger lecitina mediante lecitina mediante centrifugación, y después añadir una PLC, una PLC y/o una PLA para eliminar fosfolípidos no hidratables.
19. Un método para desacilar una cadena de ácido graso 2’ o 3’ de un lípido A, que comprende poner en contacto el lípido A con el polipéptido de cualquiera de las reivindicaciones 7 a 9, o el polipéptido que tiene actividad de fosfolipasa codificado por la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2.
20. Un método para obtener un biocombustible, que comprende:
100 101 102
(A)
(a) proporcionar el polipéptido de cualquiera de las reivindicaciones 7 a 9, o el polipéptido que tiene actividad de fosfolipasa codificado por la secuencia de ácido nucleico de la reivindicación 1 o reivindicación 2;
(b) proporcionar una composición de biomasa;
(c) poner en contacto el polipéptido de (a) con la composición de biomasa de (b) ;
(B) el método de (A) , en el que el biocombustible es o comprende un biodiésel; o
(C) el método de (A) o (B) , en el que la composición de biomasa comprende un alga, un aceite vegetal, un
aceite vegetal puro, un aceite vegetal virgen, un aceite vegetal residual, una grasa animal, una grasa, un 10 sebo, una manteca o una grasa amarilla.
103 104 105 106 107 108 109 110 111 112 113
ººººººººººººººº
SEC ID RESULTADOPÚBLICO MÁSELEVADO EVALORPÚBLICO DEFINICIÓNPÚBLICA ORGANISMO PÚBLICO NÚMEROECPÚBLICO %IDPROTEÍNAPÚBLICA %ID NUCLEÓTIDOPROTEÍNAPÚBLICO LONGITUD DESECUENCIADIVERSA LONGITUD DE SECUENCIAPÚBLICA
1, 2 gi|2815227|emb|CAA76148.1| e-94 Fosfolipasa C quedegrada fosfatidilo Bacillusthuringiensis N/A 80, 5 79, 3 287 283
3, 4 gi|2815227|emb|CAA76148.1| e-94 Fosfolipasa C quedegrada fosfatidilo Bacillusthuringiensis N/A 77, 3 77 283 283
5.6 gi|130081|spP09598| e-134 Fosfolipasa C quedegrada fosfatidilo Bacillus cereus N/A 77 76 280 272
7, 8 gi|3044072|gbAAC 13276.1 e-97 Beta-hemolisina Staphylococcusschleiferi N/A 54 33 330 329
FIGURA 11A FIGURA 11B FIGURA 11C FIGURA 11D FIGURA 11E FIGURA 11F
SEC ID NO: Definición Genbank Organismo Número EC Número de Proteína Gl Evalor NR Número de ADN Gi Longitud de ADN Longitud de proteína Longitud de ADN público Longitud deproteínapública %ID Pública de Proteína
9, 10 beta-hemolisina -Staphylococcus aureus Staphylococcusaureus 3.1.4.3 97786 9E-34 14647263 999 332 993 331 32
11, 12 3.4.21 23474722 3E-52 14276066 1041 346 N/A 339 38
13, 14 3.4.21. 23474722 7E-55 14530226 1038 345 N/A 339 38
15, 16 Proteína hipotética[Pseudomonasaeruginosa PA01] Pseudomonasaeruginosa PA01 15598123 1E-102 9949021 1344 447 1329 443 46
17, 18 3.1.1.4 23127917 4E-24 14670061 1137 378 N/A 391 29
19, 20 27367835 0 3746408 1248 415 N/A 417 72
21, 22 fosfolipasa C[Aeromonas hydrophila] Aeromonashydrophila 3746953 1E-169 3746952 1716 571 1716 572 53
23, 24 proteína hipotéticaconservada[Xanthomonascampestris pv.campestris str. ATCC33913] 21232725 2E-51 4753846 1473 490 1344 448 31
25, 26 3.4.21. 23474722 4E-50 13938818 1098 365 N/A 339 36
27, 28 23474722 4E-26 13173617 1287 428 N/A 339 27
29, 30 proteína hipotéticaconservada[Xanthomonascampestris pv.campestris str. ATCC33913] 21233052 0.00007 14589793 753 250 1311 437 20
31, 32 fosfolipasa C (EC3.1.4.3) precursora -Bacillus cereus Bacillus cereus 3.1.4.3 2126777 2E-48 11558558 1422 473 1776 592 32
33, 34 proteína hipotética[Xanthomonascampestris] Xanthomonascampestris 6689533 1E-18 1160468 792 263 855 285 30
35, 36 Secuencia 2 dedcoumento US 5824864parental 5972806 5E-14 2723462 1389 462 1230 410 20
37, 38 Fosfolipasa C[Aeromonas hydrophila] Aeromonashydrophila 3746953 1E-149 3746952 1329 443 1716 572 59
39, 40 proteína hipotéticaconservada[Pseudomonasaeruginosa PA01] Pseudomonasaeruginosa PA01 15598123 1E-106 9949021 1335 444 1329 443 49
41, 42 proteína hipotéticaconservada[Xanthomonasaxonopodis pv. citri str.306] Xanthomonasaxonopodis pv.citri str. 306 21241345 2E-63 3885908 1419 472 1320 440 35
43, 44 23474722 4E-26 13529454 1287 428 N/A 339 27
45, 46 3.4.21. 23474722 4E-55 14530226 1038 345 N/A 339 38
47, 48 proteína hipotética[Nostoc sp. PCC 7120] Nostoc sp. PCC7120 17230632 6E-25 6322016 1476 491 1365 455 23
49, 50 27367835 1E-166 10998526 1257 418 N/A 417 65
51, 52 proteína hipotéticaconservada[Xanthomonascampestris pv.campestris str. ATCC33913] 21232725 2E-50 13122711 1482 493 1344 448 30
53, 54 proteína hipotética[Pseudomonasaeruginosa PA01] Pseudomonasaeruginosa PA01 15598123 1E-16 13928640 1491 496 1329 443 22
55, 56 3.4.21. 23474722 2E-53 8896126 1041 346 N/A 339 39
57, 58 proteína hipotética (Pseudomonasaeruginosa PA01 Pseudomonasaeruginosa PA01 15598123 1E-18 3337392 1413 470 1329 443 22
59, 60 3.4.21. 23474722 9E-56 14280415 1038 345 N/A 339 39
61, 62 lecitinasa [Vibrio mimicus] 3746409 0 3746408 1257 418 1410 470 77
63, 64 27367835 1E-173 3746408 1242 413 N/A 417 67
65, 66 3.1.1.4 23041851 3E-19 14993667 1164 387 N/A 337 32
67, 68 proteína hipotética[Pseudomonasaeruginosa PA01] Pseudomonasaeruginosa PA01 15598123 9E-25 13880526 1419 472 1329 443 26
69, 70 3.4.21. 23474722 9E-56 14530226 1038 345 N/A 339 38
71, 72 quitinasa (EC3.2.1.14) A-Pseudoaiteromonas sp. (cepa S9) Pseudoalteromonassp. 3.2.1.14 7521919 2E-28 7340814 3264 1088 3162 1054 11
73, 74 proteína hipotéticaconservada[Xanthomonascampestris pv.campestris str. ATCC33913] 21233052 0.00007 14589793 753 250 1311 437 20
75, 76 proteína hipotética[Pseudomonasaeruginosa PA01] Pseudomonasaeruginosa PA01 15598123 1E-22 13157620 1335 444 1329 443 21
77. 78 3.4.21. 23474722 4E-52 14550312 1026 341 N/A 339 36
79, 80 fosfolipasa C[Aeromonas hydrophila] Aeromonashydrophila 3746953 1E-168 15022386 1701 566 1716 572 52
81, 82 FOSFOLIPASA CPRECURSORA (PLC) (FOSFATIDILCOLINACOLINAFOSFOHIDROLASA) (CEREOLISINA A) Bacillus cereus 3.1.4.3 130081 1E-49 8570206 1422 473 849 283 26
83, 84 hemolisina [Vibrio harveyi] Vibrio harveyi 10998525 5E-61 10716598 1290 429 1254 418 33
85, 86 3.4.21. 23474722 1E-55 14530226 1038 345 N/A 339 38
87, 88 3.4.21. 23041851 3E-12 11121040 870 289 N/A 337 28
89, 90 FOSFOLIPASA CPRECURSORA (PLC) (FOSFATIDILCOLINACOLINAFOSFOHIDROLASA) (CEREOLISINA A) Bacillus cereus 3.1.4.3 130081 1E-49 8570206 1422 473 849 283 26
91, 92 3.4.21. 23474722 4E-54 3947685 1035 344 N/A 339 38
93, 94 beta-hemolisina -Staphylococcus aureus Staphylococcus aureus 3.1.4.3 97786 6E-95 3044071 963 320 993 331 54
95, 96 3.4.21. 23474722 2E-52 13518253 1038 345 N/A 339 38
97, 98 fosfolipasa C (EC 3.1.4.3) precursora - Bacillus cereus Bacillus cereus 3.1.4.3 2126777 2E-49 3766134 1422 473 1776 592 32
99, 100 3.1.1.4 23129073 4E-43 7544033 1053 350 N/A 698 34
101, 102 3.1.4.3 27466926 3E-73 5802872 996 331 N/A 334 45
103, 104 Y4il [Rhizobium sp. NGR234] Rhizobium sp.NGR234 16519782 3E-07 6755291 2205 734 2109 703 15
105, 106 Proteína relacionada confosfolipasa C [Clostridium acetobutylicum] Clostridiumacetobutylicum 15894306 0.005 2182412 756 251 735 245 18
SEC ID NO: DescripciónNR CódigodeAccesoNR EvalorNR OrganismoNR Descripción deProteína deGeneseq Código deAcceso deProteínaGeneseq EvalordeProteínadeGeneseq Descripciónde ADN de Geneseq Código deAcceso deADNGeneseq Evalorde ADN deGeneseq Nº ECPredicho Longitudde ADNdeConsulta LongituddeProteínadeConsulta Longitudde ADNdeGeneseqo NR LongituddeProteínadeGeneseqo NR %ID deProteínadeGeneseq o NR %ID deADN deGeneseqo NR
1fosfatidilinositolfosfodiesterasaprecursora[Bacilluscereus] 107, 108 ATCC 14579] 30021654 0 BacilluscereusATCC14579 Listeriamonocytogenes proteína #849. ABB47680 3E-18 Arabidopsisyellowstripel-like 4SEC ID NO10. ABN85766 0.25 3.1.4.10 990 329 990 329 92 91
1fosfatidilinositolfosfodiesterasaprecursora[Bacilluscereus] 109, 110 ATCC 14579] 30021854 0 BacilluscereusATCC14579 ListeriamonocytogenesProteína #849 ABB47680 6E-18 Arabidopsisyellowstripel-like 4SEC ID NO 10. ABN85766 0.25 3.1.4.10 990 329 990 329 92 92
111, 112 fosfolipasa C específica de fosfatidilinositol[Listeria seeligeri] 2231002 8E-25 Listeriaseeligeri Listeriamonocytogenes protein #849. ABB47680 2E-24 Secuencia#13de ADNc humanoaumentó en CC-RCC no agresivo ABX74467 3.2 3.1.4.10 828 275 963 320 31 42
113.114 1-fosfatidilinositol fosfodiesterasaprecursora[Bacillus cereusATCC 14579] 30021854 1E180 BacilluscereusATCC14579 Listeria monocytogenesproteína #849. ABB47680 8E-18 Arabidopsisyellow stripellike 4 SEQ ID NO 10. ABN85766 0.24 3.1.4.10 981 326 990 329 92 92
115, 116 1-fosfatidilinositol fosfodiesterasaprecursora[Bacillus cereusATCC 14579] 30021854 1E178 BacilluscereusATCC14579 Listeriamonocytogenesproteína #849. ABB47680 2E-16 Secuencia deADN #201relacionadacon apoptosismurina ABL01358 0.97 3.1.4.10 987 328 990 329 89 90
FIGURA 11G
117, 118 1fosfatidilinositolfosfodiesterasaprecursora[Bacillus cereus ATCC 14579] 30021854 1E-179 BacilluscereusATCC14579 Listeriamonocytogenesproteína #849. ABB47680 8E-16 Sonda 47.08 de receptorcentral decannabinoideshumano, SECID NO:9. ABZ57017 0.97 3.1.4.10 987 328 990 329 90 91
119.120 1fosfatidilinositol fosfodiesterasaprecursora[Bacilluscereus ATCC 14579] 30021854 1E-178 BacilluscereusATCC14579 Listeriamonocytogenesproteína #849. ABB47680 8E-16 Secuencia deADN #201relacionadacon apoptosismurina ABL01358 0.97 3.1.4.10 987 328 990 329 89 90
FIGURA 11H FIGURA 11I
121, 122 1fosfatidilinositolfosfodiesterasaprecursora[Bacilluscereus ATCC 14579] 30021854 0 BacilluscereusATCC14579 Listeriamonocytogenesproteína #849. ABB47680 3E-17 Arabidopsis yellowstripel-like 4 SEC ID NO10. ABN85766 0.016 3.1.4.10 990 329 990 329 92 92
123, 124 Fosfolipasa C [Bacilluscereus ATCC 14579] 30018852 1E-137 BacilluscereusATCC14579 Listeriamonocytogenesproteína #849. ABB47676 1E-49 SEC ID:505Secuencianucleotídicade antígeno asociado acáncer de próstata humano AAF22890 0.053 3.1.4.3 849 282 283 81
125, 126 Y4il[Rhizobium sp.NGR234] 16519782 1E-69 Rhizobiumsp.NGR23 4 ADN que codifica nueva proteína de diagnósticohumana#20574. ABG07933 0.045 Gen de M.capsulatus#766 para chip de ADN. BQ91418A 0.43 1710 569 2112 703 34 54
127, 128 proteínahipotéticaconservada[Porphyromonasgingivalis W83] 34541487 2E96 Porphyromonasgingivalis W83 Mycobacteriumtuberculosisproteína 10. ABJ04710 2E-14 Secuencia deADNc NShumana SECID NO:76. ABL39805 1 34.21. 1038 345 339 52
129, 130 fosfolipasa C (EC 3.1.4.3) precursora -Bacillus cereus. 2126777 2E50 Bacillus cereus Listeriamonocytogenesproteína #849. ABB47676 2E-34 SPTMpolinucleótidosecretorhumano SECID NO 534. ABZ35958 0.36 3.1.4.3 1434 477 592 31
131, 132 alérgeno de látexde Hevea brasiliensis[Chromobacteriumviolaceum ATCC 12472] 34495896 4E51 Chromobacteriumviolaceurn ATCC 12472 Arabidopsisthalianafragmento de proteína SECID NO:76191. AAG42787 4E-22 Arabidopsisthaliana fragmento deproteína SECID NO:76191. AAC42405 0.058 3.1.1.4 927 308 322 37
FIGURA 11J
133, 134 proteínahipotéticaconservada[Porphyromonasgingivalis W83] 34541487 1E90 Porphyromonasgingivalis W83 Mycobacteriumtuberculosisproteína 10. ABJ04710 2E-20 3.4.21. 1053 350 339 50
135, 136 Y4il [Rhizobiumsp. NGR234] 16519782 1E69 Rhizobium sp. NGR234 ADN quecodifica nuevaproteína dediagnósticohumana#20574. ABG07933 1.5 ADN deStaphylococcusaureus paraproteína deproliferacióncelular #1219. AAS51470 1.7 1710 569 2112 703 33 54
FIGURA 11K
137, 138 proteínahipotéticaconservada[Porphyromonasgingivalis W83] 34541487 7E97 Porphyromonas gingivalis W83 Mycobacteriumtuberculosisproteína 10. ABJ04710 2E-13 Secuencia deADNc NShumana SECID NO:76. ABL39805 1 3.4.21. 1038 345 339 53
139, 140 fosfolipasa C[Aeromonas hydrophila]. 3746953 1E180 Aeromonashydrophila Cebador dePCR parasecuenciacodificante deproteína deunión detopoisomerasaII. AAY03183 3.2 Oligonucleotidoanalizador de metilación del gen deangiotensina#2. AAD28391 6.6 1692 563 1719 572 53 58
FIGURA 11L
Agua
127 128 129 130 131 132 133 134 135 136 137 138 139 140 141
Glucosa
Glucosa Glucosa
tanque Condición Caudal de MeOH Alimentación de MeOH total Caudal de glucosa Alimentación de glucosa total Alimentación de base total OD6OO (88 h) pH stat U/ml (de caldo 5x)
1 Colonia #1 de cultivo zeo200, 6x DSD 0, 6-1, 2 ml/h 59 ml 1, 8 ml/h 135, 3 ml 55, 5 ml 436 3119 a 75 h 3987 a 88 h 4138 a 92 h
2 Colonia #2 de cultivo zeo200, 6x DSD 0, 6-1, 2 ml/h 61 ml 1, 8 ml/h 135, 6 ml 56, 1 ml 461 3511 a 92 h
4 Colonia #4 de cultivo zeo200, 6x DSD 0, 6-1, 2 ml/h 58 ml 1, 8 ml/h 135, 6 ml 56, 1 ml 444 3354 a 75 h 3998 a 88 h 4133 a 92 h
6 Colonia #6 de cultivo zeo200, 6x DSD 0, 6-1, 2 ml/h 48 ml 1, 8 ml/h 112, 1 ml 40, 2 ml 445 2117 a 92 h
7 BD15359, 6x DSD control 0, 6-1, 2 ml/h 56 ml 1, 8 ml/h 135, 1 ml 55, 1 ml 436 2369 a 92 h
8 Duplicado del tanque 7 0, 6-1, 2 ml/h 53 ml 1, 8 ml/h 132, 7 ml 51, 9 ml 428 1932 a 92 h
FIGURA 26
142 143 144 145 146 147 148 149 150 FIGUR.
3. 1
151 FIGUR.
3. 2
D234W GAT TGG 0, 045 0, 000 0, 302 0, 000 127 881 6, 937 14, 552 1.382
D234V GAT GTG 0, 051 0, 000 0, 312 0, 000 145 915 6, 310 13, 574 1.349
D234G GAT GGT 0, 057 0, 000 0, 297 0, 000 162 873 5, 389 12, 320 1.377
D234R GAT CGG 0, 043 0, 000 0, 310 0, 000 120 897 7, 475 16, 582 1.344
D234K GAT AAG 0, 045 0, 000 0, 280 0, 000 126 809 6, 421 15, 011 1.336
Q265R CAG CGT 0, 074 0, 000 0, 000 0, 000 209 0 0, 000 #DIV/0! 1.441
progenitora (SEQ ID NO: 176) 0, 072 0, 000 0, 278 0, 000 205 816 3, 980 9, 948 1, 338
progenitora (SEQ ID NO: 176) 0, 082 0, 000 0, 280 0, 000 230 811 3, 526 9, 186 1, 428
Control positivo (E41A) 0, 021 0, 000 0, 340 0, 000 59 985 16, 690 28, 320 1, 470
Control negativo 0, 119 0, 450 0, 357 0, 643 338 1049 3, 104 4, 672 0, 466
Control negativo 0, 096 0, 346 0, 234 0, 517 270 681 2, 522 9, 093 0, 417
152 153 154
Patentes similares o relacionadas:
Proceso de fermentación para la producción y control de productos derivados de piruvato, del 8 de Julio de 2020, de Lanzatech New Zealand Limited: Un método para aumentar la producción de al menos un producto derivado de piruvato durante la fermentación microbiana, comprendiendo el método: a) proporcionar […]
Proceso para la producción de lípidos a partir de biomasa empleando microorganismos oleaginosos, del 1 de Julio de 2020, de ENI S.P.A.: Proceso para la produccion de lipidos a partir de biomasa que incluye al menos un polisacarido que comprende: - someter dicha biomasa que incluye al menos […]
METODO PARA INCREMENTAR LA PRODUCCION DE BIOMASA Y DE LIPIDOS EN CHLORELLA VULGARIS, del 18 de Junio de 2020, de CENTRO DE INVESTIGACIÓN CIENTÍFICA Y DE EDUCACIÓN SUPERIOR DE ENSENADA, BAJA CALIFORNIA (CICESE): Un método para incrementar la producción de biomasa y de ácidos grasos en el alga Chlorella vulgaris Beyerinck y obtener una biomasa […]
Métodos para producir ácido graso poliinsaturado y lípido que contiene ácido graso poliinsaturado, del 17 de Junio de 2020, de SUNTORY HOLDINGS LIMITED: Un método para producir ácido araquidónico (ARA) y/o ácido dihomo-gamma-linolénico (DGLA), comprendiendo el método el cultivo Mortierella alpina capaz de producir […]
Preparación de aceite microbiano que contiene ácidos grasos poliinsaturados, del 10 de Junio de 2020, de DSM IP ASSETS B.V.: Un aceite microbiano que comprende al menos el 50% de ácido araquidónico (ARA) basado en el aceite y que tiene un contenido en triglicéridos de al menos el 90%.
Métodos y composiciones para la producción mejorada de ácidos grasos y derivados de los mismos, del 27 de Mayo de 2020, de Genomatica, Inc: Un método para producir un ácido graso o derivado del mismo, que comprende; (a) proporcionar una célula hospedadora bacteriana modificada por ingeniería genética que comprende […]
Procedimiento para la hidrólisis de material lignocelulósico, en el que el hidrolizado se usa para la producción de hidrolasa microbiana, del 6 de Mayo de 2020, de CLARIANT INTERNATIONAL LTD.: Un procedimiento para la hidrólisis enzimática autosuficiente de material que contiene lignocelulosa, que comprende las etapas de: (a) someter un material que contiene lignocelulosa […]
Un proceso continuo de producción de aceite biológico a partir de licor negro usado, del 6 de Mayo de 2020, de SCA Forest Products AB: Un proceso para producir de manera continua aceite biológico que comprende las etapas de a) formar una composición de licor negro en un tanque de mezcla (T) […]