Espectrometria de masas de resistencias por betalactamasas.
Método para la determinación de resistencia a antibióticos betalactámicos de microbios basada en la síntesis microbiana de betalactamasas,
en el cual se añaden los microbios a un sustrato y se mide la degradación enzimática del sustrato causada por las betalactamasas de los microbios mediante espectrometría de masas, obteniendo así un espectro de masa del sustrato restante y del producto de degradación.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/EP2011/059670.
Solicitante: Bruker Daltonik GmbH.
Nacionalidad solicitante: Alemania.
Dirección: Fahrenheitstrasse 4 28359 Bremen ALEMANIA.
Inventor/es: KOSTRZEWA,MARKUS, MICHELMANN,KARSTEN, SPARBIER,KATRIN.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/34 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que interviene una hidrolasa.
- G01N33/68 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › en los que intervienen proteínas, péptidos o aminoácidos.
- G01N33/94 G01N 33/00 […] › en los que intervienen narcóticos.
PDF original: ES-2515466_T3.pdf
Fragmento de la descripción:
Espectrometría de masas de resistencias por betalactamasas
Sector de la invención
[1] La invención se refiere a la determinación de la resistencia de bacterias que producen betalactamasas, incluidas las «betalactamasas de espectro extendido» (BLEE).
Estado de la técnica
[2] Muchos tipos de microbios, en particular las bacterias y los hongos unicelulares, pueden identificarse fácilmente con espectrometría de masas mediante la transferencia de pequeñas cantidades de microbios de una colonia cultivada de la forma habitual sobre o en un medio nutritivo a una placa de soporte de muestras para espectrometría de masas, donde se preparan con una disolución de una matriz y se miden mediante espectrometría de masas tras la ionización por desorción láser asistida por matriz. El espectro de masa representa masas e intensidades de proteínas características en el caso de que estas estén presentes en los microbios en una concentración suficiente. Este espectro, que muestra picos de alrededor de 4 a 8 proteínas de los microbios cada vez, es utilizado para determinar su identidad mediante análisis de similitud con miles de espectros de referencia en las colecciones de espectros correspondientes. El término «identificación» denota aquí una clasificación taxonómica, por ejemplo la determinación de la familia, género y especie. La investigación se lleva a cabo en muchos lugares para cotejar las colecciones fiables y susceptibles de aprobación legal para aplicaciones médicas (las denominadas colecciones «homologadas») con espectros de referencia de miles de microbios.
[3] Este método de identificación de microbios ha demostrado una extraordinaria eficacia tanto para estudios como para la actividad diaria de numerosos laboratorios de microbiología. Es un método rápido, con costes reducidos y tasas de error muy bajas, muy inferiores a los métodos de identificación microbiana convencionales.
[4] Se pueden utilizar versiones especiales de estos métodos para identificar no solo especies microbianas sino a menudo también subespecies e incluso en ocasiones las cepas individuales, siempre que la masa o intensidad de las proteínas más frecuentes de estas cepas sea diferente y, por tanto, se pueda detectar mediante espectrometría de masas. Para una descripción más detallada, consultar la solicitud de patente DE 1 29 32 649 Al (T. Maier y M. Kostrzewa, 29, US
211/1216 Al; GB 2 471 746 A), por ejemplo, que no solo presenta una explicación detallada del método sino también un proceso de identificación más refinado.
[5] La identificación microbiana desempeña una función importante en las enfermedades infecciosas, en particular en el caso de una sepsis. En este caso es importante ser capaz de identificar las especies de microorganismos patógenos muy rápidamente, para así aplicar el tratamiento médico adecuado de forma inmediata. La identificación mediante espectrometría de masas se ha ensayado y probado también en estos casos, y actualmente está logrando aceptación en laboratorios clínicos y de microbiología.
[6] No obstante, en el campo médico no solo se hace frente al problema de una identificación rápida sino también al de la detección de resistencias a los antibióticos de uso habitual. Es imposible lograr una rápida lucha contra enfermedades infecciosas sin conocer las resistencias. Por este motivo no solo es preciso obtener una rápida identificación sino también determinar y caracterizar rápidamente las resistencias de los microorganismos. Se sabe que algunas especies microbianas poseen una resistencia casi total a ciertos antibióticos, por lo que tras una identificación precisa ya no tiene sentido determinar la resistencia. Sin embargo, en la mayoría de los casos las especies tienen algunas cepas que no son resistentes, otras con una resistencia reducida y en particular cepas con una gran resistencia, y además muestran diferentes grados de resistencia a diferentes tipos de antibióticos. Por ese motivo es esencial determinar el tipo y la intensidad de la resistencia.
[7] Parece lógico, por tanto, no limitar el uso de los espectrómetros de masas a la identificación taxonómica, sino utilizarlos también para determinar las resistencias de los microbios, en particular de las bacterias, a ciertos antibióticos. No obstante, esta labor se ha revelado muy complicada. Aunque las resistencias deben manifestarse también por la presencia de proteínas nuevas o modificadas, aún no se ha logrado una identificación directa a partir del perfil proteínico resultante de la espectrometría de masas. Al fin y al cabo, de los cientos o incluso miles de proteínas de los microbios en el espectro de masa solo aparecen de 4 a 8 de ellas. Por lo tanto, la resistencia debe determinarse de forma indirecta. En el documento DE 1 26 21 493 B4 (V. Govorun y J. Franzen; GB 2 438 66 B; US 28/929 Al) se presenta un primer intento de una determinación de la resistencia de este tipo, si bien este método aún no está aceptado. El método se basa fundamentalmente en la modificación de los perfiles proteínicos provocada por la muerte
celular tras la adición de antibióticos o en la detección de una interrupción del crecimiento en comparación con los microbios resistentes de referencia.
[8] El término «resistencia a los antibióticos» se utiliza para referirse a características de microorganismos (aquí principalmente bacterias) que les permiten mitigar o neutralizar por completo el efecto de los principios activos antibióticos. Las resistencias están ahora muy extendidas; en EE. UU. en tomo al 7 % de los microbios infecciosos adquiridos en hospitales son resistentes al menos a un antibiótico. Con frecuencia los pacientes se contagian con cepas bacterianas resistentes a varios antibióticos (multirresistencia). Las denominadas bacterias problemáticas son el Staphylococcus aureus resistente a la meticilina (SARM), el género Pseudomonas, la Escherichia coli con resistencia por BLEE y el Mycobacterium tuberculosis. Los cálculos realizados por los CDC (Centros para el Control y Prevención de Enfermedades) indican que en el año 24 se adquirieron en los hospitales de EE. UU. dos millones de infecciones con alrededor de 9 muertes, una cifra muy superior a la de muertes por accidentes de carretera, domésticos o industriales.
[9] Habitualmente el término «antibiótico» se refiere a fármacos o productos farmacéuticos empleados para el tratamiento de enfermedades infecciosas bacterianas. El enorme éxito de los antibióticos en el campo de la medicina comenzó con la penicilina. El éxito de la penicilina, así como la aparición de las primeras resistencias, llevó a los investigadores a buscar y descubrir muchos más antibióticos: estreptomicina, cloranfenicol, aureomicina y tetraciclina, entre muchos otros. La mayoría de los antibióticos conocidos actualmente derivan de sustancias naturales. En el lenguaje coloquial la penicilina se utiliza ahora como sinónimo de antibiótico.
[1] La penicilina es un antibiótico betalactámico. Estos antibióticos betalactámicos se fijan a la proteína fijadora de la penicilina (PFP), un peptidoglicano con actividad transpeptidasa responsable de la formación de enlaces peptídicos para reforzar las paredes celulares. Los enlaces entre los antibióticos betalactámicos y la PFP inhiben a la PFP. Una cantidad insuficiente de PFP activa provoca lesiones en la pared celular a medida que las bacterias crecen; así, la membrana pierde el control de su permeabilidad y la capacidad de regular la concentración en el citoplasma. Tras un breve intervalo de tiempo la bacteria se vuelve inviable. Bajo condiciones extremas, en el laboratorio se puede observar cómo las células bacterianas «estallan» literalmente. Esta es la forma como los antibióticos betalactámicos actúan como bactericidas.
[11] Desde las primeras aplicaciones de la penicilina las bacterias han ido desarrollando diferentes tipos de resistencias a ritmo creciente. Un tipo importante de resistencia bacteriana a los antibióticos betalactámicos consiste en la síntesis de unas enzimas (las betalactamasas) que catalizan la hidrólisis del anillo betalactámico, rompiéndolo e inhibiendo así su acción. Actualmente se conocen más de 34 variantes de betalactamasas sintetizadas por muchos tipos de bacterias. Pueden dividirse en diferentes clases en función de su estructura general o su forma de actuación. La información genética para la síntesis de la enzima, originada por unas mutaciones, es heredada por cromosomas o por plásmidos. La información plasmídica puede transmitirse entre bacterias mediante diversos mecanismos, incluso entre bacterias de diferentes especies por contacto («transmisión horizontal»). En función de la acción de las betalactamasas... [Seguir leyendo]
Reivindicaciones:
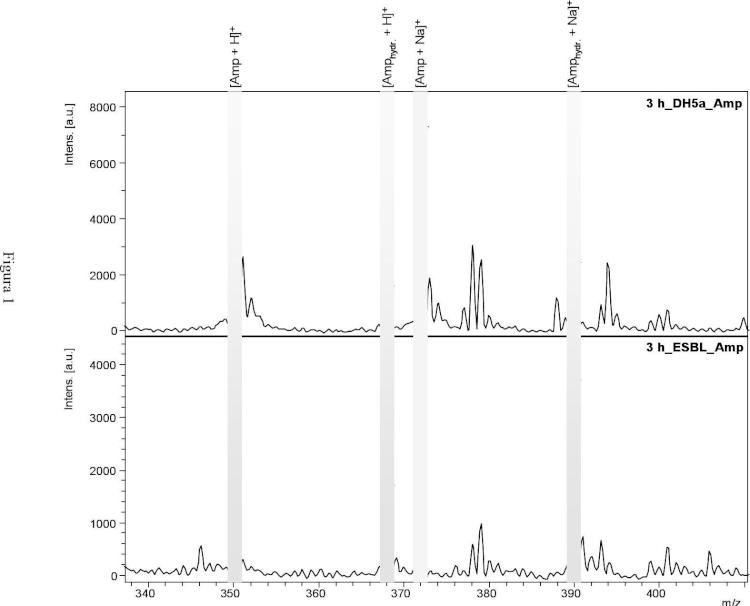
1. Método para la determinación de resistencia a antibióticos betalactámicos de microbios basada en la síntesis microbiana de betalactamasas, en el cual se añaden los microbios a un sustrato y se mide la degradación enzimática del sustrato causada por las betalactamasas de los microbios mediante espectrometría de masas, obteniendo así un espectro de masa del sustrato restante y del producto de degradación.
2. Método según la reivindicación 1, en el cual las moléculas del sustrato contienen un anillo betalactámico.
3. Método según la reivindicación 2, en el cual el sustrato es un antibiótico betalactámico o un derivado betalactámico.
4. Método según la reivindicación 1, en el cual el sustrato tiene una masa molecular de entre 7 y 12 unidades de masa atómica.
5. Método según la reivindicación 1, en el cual el sustrato tiene un efecto antibiótico débil.
6. Método según la reivindicación 1, en el cual las moléculas del sustrato contienen un grupo enlazante que puede utilizarse para extraerlas de disoluciones.
7. Método según la reivindicación 6, en el cual el grupo enlazante es una biotina o un marcador de hexahistidina.
8. Método según la reivindicación 1, en el cual la degradación de varios tipos de sustrato se mide de forma simultánea.
9. Método según la reivindicación 8, en el cual los diferentes tipos de sustrato se preparan a medida para que sus patrones de degradación permitan la identificación de diferentes clases de betalactamasas.
1. Método según la reivindicación 9, en el cual los diferentes sustratos en tomo al anillo betalactámico imitan las formas estéricas de diferentes antibióticos.
11. Método según la reivindicación 1, en el cual se miden las velocidades de degradación de los sustratos.
12. Método según la reivindicación 1, en el cual los microbios se han obtenido a partir de la sangre
o de un hemocultivo.
13. Método según la reivindicación 1, donde se miden las cantidades de sustrato restante y su producto de degradación mediante espectrometría de masas con ionización por desorción láser asistida por matriz.
14. Método para la determinación de una resistencia a antibióticos betalactámicos de microbios
basado en la síntesis microbiana de betalactamasas, que comprende los siguientes pasos:
(a) Añadir los microbios a la disolución de al menos un sustrato que pueda ser degradado por las betalactamasas.
(b) Incubar la disolución a una temperatura especificada durante un tiempo especificado.
(c) Separar la disolución con el sustrato restante y su producto de degradación de los microbios,
(d) Obtener un espectro de masa de la disolución.
Patentes similares o relacionadas:
Inmunomoduladores, del 29 de Julio de 2020, de BRISTOL-MYERS SQUIBB COMPANY: Un compuesto de la fórmula (I) **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo, en donde: A se selecciona de **(Ver fórmula)** en donde: […]
Métodos y composiciones para el diagnóstico y pronóstico de lesión renal e insuficiencia renal, del 29 de Julio de 2020, de Astute Medical, Inc: Un método para evaluar el estado renal en un sujeto, que comprende: realizar una pluralidad de ensayos configurados para detectar una […]
Neuregulina para tratar la insuficiencia cardíaca, del 29 de Julio de 2020, de Zensun (Shanghai) Science & Technology, Co., Ltd: Neuregulina para usar en un método para tratar la insuficiencia cardíaca crónica en un paciente, donde el paciente tiene un nivel plasmático de NT-proBNP […]
Método para llevar a cabo el seguimiento de la enfermedad de Gaucher, del 15 de Julio de 2020, de Centogene GmbH: Un método para determinar la evolución de la enfermedad de Gaucher en un sujeto, que comprende la etapa de determinar en varios puntos en el […]
Procedimiento para evaluación de la función hepática y el flujo sanguíneo portal, del 15 de Julio de 2020, de The Regents of the University of Colorado, a body corporate: Procedimiento in vitro para la estimación del flujo sanguíneo portal en un individuo a partir de una única muestra de sangre o suero, comprendiendo el procedimiento: […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos de monitorización terapéutica de profármacos de ácido fenilacético, del 24 de Junio de 2020, de Immedica Pharma AB: Glicerilo tri-[4-fenilbutirato] (HPN-100) para su uso en un método para tratar un trastorno del ciclo de la urea en un sujeto que tiene discapacidad […]