ENSAYOS DE COMPLEMENTACION DE FRAGMENTOS PROTEINICOS.
Método que utiliza un ensayo de complementación proteínica/sistema indicador universal (PAC/URS) para la detección y la localización de un ligando de un receptor celular,
incluyendo dicho método:
(a) la generación de un primer vector de ácido nucleico que codifique un primer producto de fusión, que comprende:
(i) un primer fragmento de una primera molécula indicadora PCA/URS, y
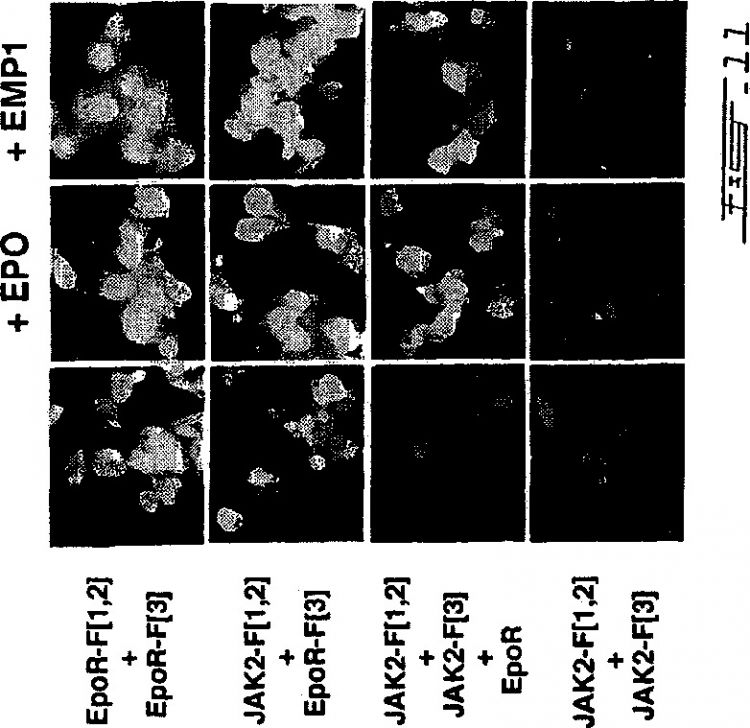
(ii) una segunda molécula, fusionada con dicho primer fragmento, que comprende un primer subdominio del receptor de eritropoietina;
(b) la generación de un segundo vector de ácido nucleico que codifique un segundo producto de fusión, que comprende:
(i) un segundo fragmento de dicha primera molécula indicadora PCA/URS, y
(ii) una tercera molécula, fusionada con dicho segundo fragmento, que comprende un segundo subdominio de dicho receptor de eritropoietina, y en la que dicho segundo subdominio puede ser el mismo que dicho primer subdominio, o diferente de dicho primer subdominio;
(c) la transfección de células procarióticas o eucarióticas con dicho primer y segundo vectores de ácido nucleico; y
(d) verificación en dichas células transfectadas de la actividad indicadora del PCA/URS, indicando dicha actividad la reasociación del primer y del segundo fragmento de la molécula PCA/URS mediada por la interacción de dichos primer y segundo subdominios del receptor de eritropoietina, estando inducida dicha asociación mediante el enlace de dicho receptor con dicho ligando,
en el que dicha molécula indicadora PCA/URS es un polipéptido codificado por un gen que se ha diseccionado racionalmente en al menos dos fragmentos que codifican, respectivamente, dicho primer fragmento y dicho segundo fragmento de dicha molécula indicadora PCA/URS
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/CA99/00702.
Solicitante: UNIVERSITE DE MONTREAL.
Nacionalidad solicitante: Canadá.
Dirección: 6128, SUCCURSALE A,MONTREAL, QUEBEC H3C 3J7.
Inventor/es: MICHNICK, STEPHEN, WILLIAM, WATSON, REMY, INGRID, PELLETIER,JOELLE,NINA.
Fecha de Publicación: .
Fecha Concesión Europea: 20 de Enero de 2010.
Clasificación Internacional de Patentes:
- C12N15/10C6
- G01N33/566 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › utilizando un soporte específico o proteínas receptoras como reactivos para la formación de uniones por ligando.
- G01N33/58B
- G01N33/68 G01N 33/00 […] › en los que intervienen proteínas, péptidos o aminoácidos.
Clasificación PCT:
- G01N33/566 G01N 33/00 […] › utilizando un soporte específico o proteínas receptoras como reactivos para la formación de uniones por ligando.
- G01N33/58 G01N 33/00 […] › en los que intervienen sustancias marcadas (G01N 33/53 tiene prioridad).
- G01N33/68 G01N 33/00 […] › en los que intervienen proteínas, péptidos o aminoácidos.
Clasificación antigua:
- G01W33/68
Fragmento de la descripción:
Ensayos de complementación de fragmentos proteínicos.
Ámbito de la invención
La presente invención se refiere a la determinación de la función de nuevos productos de genes. La invención hace también referencia a ensayos de complementación de fragmentos proteínicos (PCA). Los PCA permiten la detección de una amplia gama de tipos de interacciones proteína-proteína, proteína-ARN, proteína-ADN, proteína-hidrato de carbono o proteína-pequeña molécula orgánica en diferentes contextos celulares adecuados para el estudio de dichas interacciones.
Antecedentes de la invención
Muchos procesos biológicos, incluyendo la transcripción, traducción y las vías de transacción metabólicas o de señales, están mediados por complejos multienzima sin enlaces covalentes1, 101. La formación de complejos ácidos multiproteínicos o con núcleo proteínico producen la maquinaria química más eficaz. Una buena parte de la moderna investigación biológica se refiere a la identificación de proteínas que participan en procesos celulares, determinando sus funciones y cómo, cuándo y dónde interactúan con otras proteínas implicadas en procesos específicos. Asimismo, gracias a los rápidos avances de los proyectos de secuenciación de genomas, se ha dado la necesidad de desarrollar estrategias que definan los "mapas de enlace de proteínas", inventarios detallados de interacciones proteínicas que constituyen conjuntos funcionales de proteínas2, 3. A pesar de la importancia de comprender la agrupación de proteínas en los procesos biológicos, existen pocos métodos convenientes para el estudio in vivo de las interacciones proteína-proteína4, 5. Entre los métodos se incluye la utilización de agentes químicos reticulantes y la transferencia de energía de resonancia entre proteínas acopladas102, 103. Una potente y frecuentemente utilizada estrategia, el sistema de doble híbrido en levadura, se utiliza para identificar nuevas interacciones proteína-proteína y para examinar los determinantes aminoácidos de interacciones proteínicas específicas4, 6, 8. El método permite un rápido filtrado de un gran número de clones, incluyendo bibliotecas cADN. Las limitaciones de esta técnica incluyen el hecho de que la interacción debe tener lugar en un contexto específico (el núcleo de S. cerevisisa), y en general, no puede utilizarse para distinguir interacciones inducidas frente a constitutivas.
Recientemente ha sido demostrada una nueva estrategia para la detección de interacciones proteína-proteína por Johnsson y Varshavsky108 denominado el sensor proteínico partido basado en ubiquitina (USPS)9. La estrategia se basa en la segmentación de proteínas con fusiones en el terminal N en ubiquitina mediante proteasas citosólicas (ubiquitinasas) que reconocen su estructura terciaria. La estrategia depende del reensamblaje de la estructura terciaria de la ubiquitina proteínica a partir de fragmentos complementarios de los terminales N y C, y crucialmente del aumento de este reensamblaje mediante dominios de oligomerización fusionados en estos fragmentos. El reensamblaje se detecta como la proteolisis específica del producto ensamblado mediante proteasas citosólicas (ubiquitinasas). Los autores demostraron que la fusión de un fragmento terminal C de ubiquitina proteínica indicadora, también podía ser segmentada por las ubiquitinasas, pero tan sólo si se expresaba conjuntamente con un fragmento del terminal N de ubiquitina complementario del fragmento del terminal C. La reconstitución de la actividad de la ubiquitinasa observable tan sólo tuvo lugar si los fragmentos de los terminales N y C se enlazaban mediante cremalleras de leucina GCN4109, 110. Los autores sugirieron que esta estrategia de "gen dividido" podría utilizarse como un ensayo in vivo de interacciones proteína-proteína y para el análisis de la cinética del ensamblaje proteínico en las células. Desgraciadamente, esta estrategia requiere factores celulares adicionales (en este caso, ubiquitinasas) y el método de detección no se presta a sí mismo para un filtrado de alto rendimiento de las bibliotecas cADN.
Rossi, F., C. A. Charlton, y H. M. Blau (1997) Proc. Nat. Acad. Sci. (USA) 94,8405-8410) han comunicado un ensayo basado en la complementación clásica de fragmentos a y ? de ß-galactosidasa (ß-gal) y en la inducción de la complementación mediante la oligomerización inducida de las proteínas FKBP12 y del objetivo en mamíferos de rapamicina por rapamicina en líneas de células mioblásticas C2C12 transfectadas. La reconstitución de la actividad de la ß-gal se detecta utilizando un sustrato de fluoresceína di-ß-D-galactopiranosida utilizando diferentes ensayos de detección de la fluorescencia. Aunque este ensayo presenta cierto parecido con la presente invención, existen diversas diferencias importantes. En primer lugar, este método de complementación específico se ha utilizado durante más de treinta años en un gran número de aplicaciones, incluyendo la detección de interacciones proteína-proteína. Krevolin, M. y D. Kates (1993), y la patente estadounidense Nº 5362625) describen la utilización de esta complementación para la detección de interacciones proteína-proteína. Previamente también se ha comunicado la complementación de la ß-gal en células de mamífero (Moosmann, P. and S. Rusconi (1996) Nucl. Acids Res. 24, 1171-1172). El documento WO 98/44350 describe un ensayo de complementación de proteína (PCA) que utiliza ß-galactosidosa como enzima indicadora. El documento WO98/34120 describe un PCA para la detección de las interacciones biomoleculares utilizando dihidrofolato reductosa de ratón (DHFR) como enzima indicadora. Pelletier J N et al: "A Protein Complementation Assay for detection of Protein - Protein interaction in vivo" Protein Engineering, GB, Oxford University Press, Surrey, vol. 10 no. Suppl. 01, -1, Octubre 1997 (1997-10-01), página 89 ISSN: 0269, también describen un PCA que utiliza DHFR como enzima indicadora.
Al igual que en el caso del USPS, la estrategia del doble híbrido en levadura requiere una maquinaria celular adicional para la detección, que existe solamente en compartimentos celulares específicos. Por lo tanto, es necesario un sistema de detección que utilice la reconstitución de una actividad enzimática específica a partir de fragmentos, como la propia prueba, sin necesidad de otras proteínas para la detección de la actividad. Preferiblemente, el ensayo implicaría una complementación asistida por oligomerización de fragmentos de enzimas monoméricas o multiméricas que no precisan otras proteínas para la detección de su actividad. Asimismo, si la estructura de una enzima se conociese, sería posible diseñar fragmentos de la enzima a fin de asegurarse de que los fragmentos reensamblados serían activos, e introducir mutaciones para alterar el rigor de la detección del reensamblaje. No obstante, no sería necesario conocer previamente la estructura para diseñar los fragmentos de complementación. La flexibilidad que permite el diseño de este método lo haría aplicable a situaciones en las que podrían no resultar adecuados otros sistemas de detección.
Un reciente avance de la investigación genómica humana ha llevado a un rápido avance en la identificación de los nuevos genes. En aplicaciones de investigación biológica y farmacéutica, se da actualmente la necesidad apremiante de determinar las funciones de nuevos productos genéticos; por ejemplo, en el caso de genes de fenotipos de enfermedad. La investigación farmacéutica basada en la genómica se estanca a la hora de abordar cuestiones de funcionamiento. Por lo tanto, es necesario que se produzcan avances en el desarrollo de ensayos funcionales sencillos y automatizables. Una primera etapa a la hora de definir el funcionamiento de un nuevo gen consiste en determinar sus interacciones con otros productos genéticos en un contexto apropiado; es decir, ya que las proteínas tienen interacciones específicas con otras proteínas u otros biopolímeros como parte de conjuntos funcionales, una forma adecuada de examinar el funcionamiento de un nuevo gen consiste en determinar sus relaciones físicas con los productos de otros genes.
Las técnicas de filtrado de interacciones proteínicas, como el sistema de doble híbrido en levadura, han transformado la biología molecular, pero tan sólo pueden utilizarse para estudiar tipos específicos de proteínas que interactúan constitutivamente o interacciones de proteínas con otras moléculas, en contextos compartimentales y celulares estrechamente definidos y que requieren una...
Reivindicaciones:
1. Método que utiliza un ensayo de complementación proteínica/sistema indicador universal (PAC/URS) para la detección y la localización de un ligando de un receptor celular, incluyendo dicho método:
(a) la generación de un primer vector de ácido nucleico que codifique un primer producto de fusión, que comprende:
(i) un primer fragmento de una primera molécula indicadora PCA/URS, y
(ii) una segunda molécula, fusionada con dicho primer fragmento, que comprende un primer subdominio del receptor de eritropoietina;
(b) la generación de un segundo vector de ácido nucleico que codifique un segundo producto de fusión, que comprende:
(i) un segundo fragmento de dicha primera molécula indicadora PCA/URS, y
(ii) una tercera molécula, fusionada con dicho segundo fragmento, que comprende un segundo subdominio de dicho receptor de eritropoietina, y en la que dicho segundo subdominio puede ser el mismo que dicho primer subdominio, o diferente de dicho primer subdominio;
(c) la transfección de células procarióticas o eucarióticas con dicho primer y segundo vectores de ácido nucleico; y
(d) verificación en dichas células transfectadas de la actividad indicadora del PCA/URS, indicando dicha actividad la reasociación del primer y del segundo fragmento de la molécula PCA/URS mediada por la interacción de dichos primer y segundo subdominios del receptor de eritropoietina, estando inducida dicha asociación mediante el enlace de dicho receptor con dicho ligando,
en el que dicha molécula indicadora PCA/URS es un polipéptido codificado por un gen que se ha diseccionado racionalmente en al menos dos fragmentos que codifican, respectivamente, dicho primer fragmento y dicho segundo fragmento de dicha molécula indicadora PCA/URS.
2. Método de la reivindicación 1, que comprende las siguientes etapas:
(a) la generación de un primer vector de ácido nucleico que codifique un primer producto de fusión, que comprende:
(iii) un primer fragmento de una primera molécula indicadora PCA/URS, y
(iv) una segunda molécula, fusionada con dicho primer fragmento, que comprende un primer subdominio del receptor de eritropoietina,
(b) la generación de un segundo vector de ácido nucleico que codifique un segundo producto de fusión, que comprende:
i) un segundo fragmento de dicha primera molécula indicadora PCA/URS, y
ii) una tercera molécula, fusionada con dicho segundo fragmento, que comprende un segundo subdominio de dicho receptor de eritropoietina, y en la que dicho segundo subdominio puede ser el mismo que dicho primer subdominio, o diferente de dicho primer subdominio;
(c) la transfección de células procarióticas o eucarióticas con dicho primer y segundo vectores de ácido nucleico; y
(d) obtención de una población clónica de células que exprese dicho primer y dicho segundo producto de fusión; y
(e) verificación en dichas células transfectadas de la actividad indicadora del PCA/URS, indicando dicha actividad la reasociación del primer y del segundo fragmento de la molécula PCA/URS mediada por la interacción de dichos primer y segundo subdominios del receptor de eritropoietina, estando inducida dicha asociación mediante el enlace de dicho receptor con dicho ligando.
3. Método de acuerdo con cualquiera de las reivindicaciones 1 a 2, en el que el segundo subdominio de dicho receptor de eritropoietina es el mismo que el primer subdominio, y la molécula receptora es un homodímero.
4. Método de acuerdo con cualquiera de las reivindicaciones 1 a 2, en el que el segundo subdominio de dicho receptor de eritropoietina es diferente del primer subdominio, y la molécula receptora es un heterodímero.
5. Método de acuerdo con cualquiera de las reivindicaciones 2 a 4, que adicionalmente incluye la etapa de tratamiento de dicha población clónica de células con un compuesto químico antes de proceder a dicha verificación de la actividad PCA/URS en las células, midiendo de este modo la capacidad del compuesto químico para inducir o inhibir dicha actividad.
6. Método de la reivindicación 5, en el que dicho compuesto químico es un compuesto individual o una mezcla de compuestos obtenidos a partir de una librería de compuestos químicos o por síntesis química combinatoria.
7. Método de acuerdo con cualquiera de las reivindicaciones 2 a 4, en el que dicha molécula indicadora es una proteína multimérica.
8. Método de acuerdo con cualquiera de las reivindicaciones 2 a 4, en el que dicha molécula indicadora es un receptor multimérico.
9. Método de acuerdo con cualquiera de las reivindicaciones 2 a 4, en el que dicha molécula indicadora es una proteína de enlace multimérico.
10. Método de acuerdo con cualquiera de las reivindicaciones 2 a 4, en el que dicha molécula indicadora es una molécula catalítica.
11. Método de acuerdo con cualquiera de las reivindicaciones 2 a 4, en el que dicha molécula indicadora es una proteína fluorescente, luminiscente o fosforescente.
12. Método de acuerdo con cualquiera de las reivindicaciones 2 a 4, en el que dicha molécula indicadora es una molécula de electro-transferencia.
13. Método de acuerdo con cualquiera de las reivindicaciones 2 a 4, en el que dicha molécula indicadora es una molécula quimioluminiscente.
14. Método de acuerdo con cualquiera de las reivindicaciones 2 a 4, en el la actividad PCA/URS se detecta mediante un ensayo de fluorescencia, y la actividad se controla mediante microscopía de fluorescencia, clasificación celular por fluorescencia (FACS) o mediante espectroscopia de alícuotas celulares.
15. Método de la reivindicación 5, en el que dicho compuesto químico es un ligando agonista o antagonista.
16. Método de la reivindicación 5, en el que dicho compuesto químico es un ácido nucleico.
17. Método de la reivindicación 5, en el que dicho compuesto químico es un péptido.
18. Método de la reivindicación 5, en el que dicho compuesto químico es un hidrato de carbono.
19. Método de la reivindicación 5, en el que en la etapa de tratamiento, las células se tratan con el compuesto químico a distintas concentraciones en el medio, y la actividad PCA/URS se compara a las distintas concentraciones.
20. Método de la reivindicación 19, en el que los valores de la actividad PCA/URS respecto de la concentración del compuesto del tratamiento se utilizan para calcular la isoterma de enlace del compuesto con el receptor de eritropoietina.
21. Método de la reivindicación 19, en el que dicha molécula indicadora es reductasa de dihidrofolato y dicho método de detección comprende el tratamiento de las células con metotrexato conjugado con fluoresceína antes de comprobar la fluorescencia celular.
22. Método de la reivindicación 6, en el que dicha librería de compuestos es una librería combinatoria de ácidos nucleicos.
23. Método de la reivindicación 6, en el que dicha librería de compuestos es una librería combinatoria de hidratos de carbono.
24. Método de la reivindicación 6, en el que dicha librería de compuestos es una librería combinatoria de péptidos o proteínas.
Patentes similares o relacionadas:
Inmunomoduladores, del 29 de Julio de 2020, de BRISTOL-MYERS SQUIBB COMPANY: Un compuesto de la fórmula (I) **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo, en donde: A se selecciona de **(Ver fórmula)** en donde: […]
Métodos y composiciones para el diagnóstico y pronóstico de lesión renal e insuficiencia renal, del 29 de Julio de 2020, de Astute Medical, Inc: Un método para evaluar el estado renal en un sujeto, que comprende: realizar una pluralidad de ensayos configurados para detectar una […]
Neuregulina para tratar la insuficiencia cardíaca, del 29 de Julio de 2020, de Zensun (Shanghai) Science & Technology, Co., Ltd: Neuregulina para usar en un método para tratar la insuficiencia cardíaca crónica en un paciente, donde el paciente tiene un nivel plasmático de NT-proBNP […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Método para llevar a cabo el seguimiento de la enfermedad de Gaucher, del 15 de Julio de 2020, de Centogene GmbH: Un método para determinar la evolución de la enfermedad de Gaucher en un sujeto, que comprende la etapa de determinar en varios puntos en el […]
Procedimiento para evaluación de la función hepática y el flujo sanguíneo portal, del 15 de Julio de 2020, de The Regents of the University of Colorado, a body corporate: Procedimiento in vitro para la estimación del flujo sanguíneo portal en un individuo a partir de una única muestra de sangre o suero, comprendiendo el procedimiento: […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos de monitorización terapéutica de profármacos de ácido fenilacético, del 24 de Junio de 2020, de Immedica Pharma AB: Glicerilo tri-[4-fenilbutirato] (HPN-100) para su uso en un método para tratar un trastorno del ciclo de la urea en un sujeto que tiene discapacidad […]