Ensayo para la metilación en el gen para la GST-Pi.
Un ensayo de diagnóstico o de pronóstico para el cáncer de próstata en un sujeto,
dicho cáncer de próstata caracterizado por metilación anormal de la citosina en un sitio dentro de la región del gen para la glutatión S-transferasa humana (GST) Pi definido por uno cualquiera de los sitios CpG +1 a +33, en donde dicho ensayo comprende las etapas de:
(i) aislar el ADN de dicho sujeto, y
(ii) determinar la presencia de metilación anormal de la citosina en un sitio individual dentro de la región del gen para la GST-Pi humana definido por uno cualquiera de los sitios CpG +1 a +33.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/AU1999/000306.
Solicitante: COMMONWEALTH SCIENTIFIC AND INDUSTRIAL RESEARCH ORGANISATION.
Nacionalidad solicitante: Australia.
Dirección: LIMESTONE AVENUE CAMPBELL, ACT 2601 AUSTRALIA.
Inventor/es: MOLLOY,PETER,LAURENCE, CLARK,SUSAN JOY, MILLAR,DOUGLAS S.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/09 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Tecnología del ADN recombinante.
- C12N15/54 C12N 15/00 […] › Transferasas (2).
- C12N9/10 C12N […] › C12N 9/00 Enzimas, p. ej. ligasas (6.); Proenzimas; Composiciones que las contienen (preparaciones para la limpieza de los dientes que contienen enzimas A61K 8/66, A61Q 11/00; preparaciones de uso médico que contienen enzimas A61K 38/43; composiciones detergentes que contienen enzimas C11D ); Procesos para preparar, activar, inhibir, separar o purificar enzimas. › Transferasas (2.) (ribonucleasas C12N 9/22).
- C12Q1/48 C12 […] › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que interviene una transferasa.
- C12Q1/68 C12Q 1/00 […] › en los que intervienen ácidos nucleicos.
PDF original: ES-2537423_T3.pdf





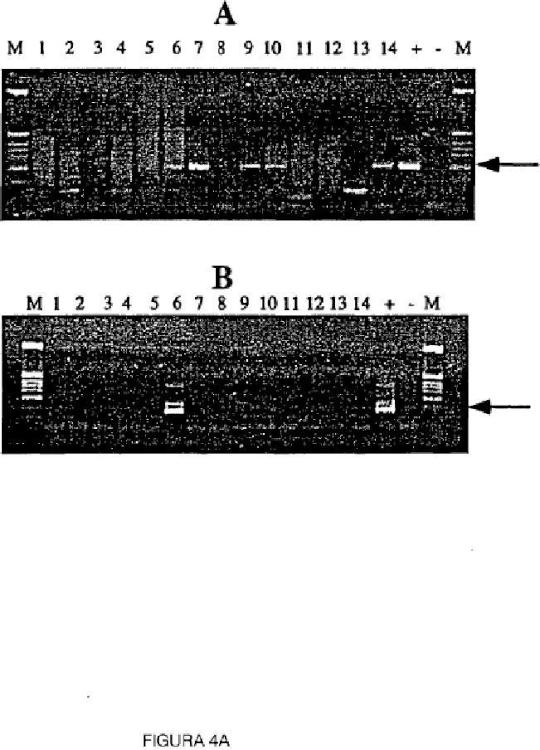
Fragmento de la descripción:
Ensayo para la metilación en el gen para la GST-Pi Campo de la invención:
Esta invención se refiere a un ensayo para el diagnóstico o pronóstico de cáncer de próstata caracterizado por la metilación anormal de la cltoslna en un sitio dentro del gen para la glutatlón S-transferasa (GST) P¡.
Antecedentes de la Invención:
Metilación del ADN en genomas de mamíferos
La única modificación post-sintética establecida del ADN en genomas de animales y plantas superiores es la metilación de la posición 5' de la citosina. La proporción de citosinas que son metiladas puede variar desde un pequeño porcentaje en algunos genomas de animales (1) hasta 3% en algunos genomas de plantas (2). Gran parte de esta metilación se encuentra en los sitios CpG en donde las citosinas simétricamente posicionadas en cada hebra son metiladas. En los genomas de plantas, la metilación simétrica similar de citosinas en CpNpG (donde N puede ser cualquier base) también es común (3). Tales sitios de metilación también se han identificado con baja frecuencia en el ADN de mamífero (4).
Los patrones de metilación son heredables ya que la enzima metilasa reconoce como sustrato, sitios donde un dinucleótido CpG está metilado en una hebra, pero la C correspondiente en la otra hebra no está metilada, y procede a metilarla (5, 6). Los sitios completamente no metilados normalmente no actúan como sustratos para la enzima y por lo tanto permanecen no metilados a través de sucesivas divisiones celulares. Por lo tanto, en ausencia de errores o eventos intervinientes específicos, la enzima metilasa permite la heredabilidad estable de los patrones de metilación.
Amplios estudios de expresión génica en vertebrados han demostrado una fuerte correlación entre la metilación de regiones reguladoras de los genes y su falta de expresión (7). La mayoría de estos estudios han examinado sólo un número limitado de sitios de enzimas de restricción usando enzimas que no logran hacer el corte si están metilados sus sitios objetivo. Un número mucho más limitado han sido examinados en todas las bases de citosina utilizando métodos de secuenciación genómica (8, 9).
Conversión de ADN con bisulfito
El tratamiento de ADN monocatenario con altas concentraciones de bisulfito seguido por álcali conduce a la desaminación selectiva de la citosina, convirtiéndola en uracilo (1, 11). Por el contrario, las 5-metil citosinas (5meC) son resistentes a esta desaminación química. Cuando el ADN tratado con bisulfito es copiado por ADN polimerasas, los uracilos se leen como si fueran timinas y un nucleótido de adenina incorporado, mientras que 5meC todavía se lee como una citosina (siendo G incorporada en frente). Por lo tanto, después de que se amplifica una región de secuencia por reacción en cadena de la polimerasa (PCR), las citosinas en la secuencia que fueron metiladas en el ADN original se leerán como citosinas mientras que las citosinas no metiladas se leerán como timinas (12, 13).
Amplificación por PCR de ADN metilado y no metilado
Con el fin de amplificar el ADN tratado con bisulfito, se diseñan los cebadores para hibridarse con la secuencia producida después del tratamiento con bisulfito del ADN. Dado que las citosinas se convierten en uracilos, la base en el cebador de hibridación será una adenina en lugar de una guanina para la citosina no convertida. Del mismo modo, para el otro cebador del par, timinas reemplazan citosinas. Para permitir la cuantificación de los niveles de metilación en el ADN objetivo, normalmente se escogen los cebadores para evitar los sitios que pueden o no ser metilados (en particular los sitios CpG) y entonces pueden contener ya sea un 5meC o un uracilo después del tratamiento con bisulfito. El uso de tales cebadores no selectivos permite amplificar tanto los ADN metilados como los no metilados por PCR, permitiendo la cuantificación del nivel de metilación en la población de partida de ADN. El ADN amplificado por PCR se puede cortar con una enzima de restricción informativa, se puede secuenciar directamente para proporcionar una medida promedio de la proporción de metilación en cualquier posición o se pueden clonar y secuenciar moléculas (cada clon se deriva de la amplificación de una hebra individual en el ADN inicial). Tales estudios han indicado que, aunque una población de moléculas puede ajustarse a un patrón general de metilación, no todas las moléculas serán idénticas y la metilación pueden encontrarse en sólo una fracción de las moléculas en algunos sitios (13, 16).
Amplificación selectiva de ADN metilado
Recientemente, Hermán et al. (14) describieron una variación del procedimiento de secuenciación con bisulfito para hacerlo selectivo para la amplificación de ADN metilado solamente. En este trabajo, se utilizaron cebadores de PCR que fueron diseñados para discriminar entre las secuencias producidas después del tratamiento con bisulfito de los ADN objetivo metilados y no metilados. Por lo tanto, las citosinas que formaban parte de un sitio CpG no serían convertidas con bisulfito y se mantendrían como citosinas en el ADN metilado pero se convertirían en uracilos en el ADN objetivo no metilado. Los cebadores que utilizan estas diferencias fueron diseñados y utilizados para la amplificación de secuencias de ADN metilado de cuatro genes supresores tumorales, cuatro p16, p15, E-cadherina y von Hippel-Lindau.
Metilación del gen para glutatión S-transferasa pi en cáncer de próstata
Lee et al. (15) (patente de los Estados Unidos No 5.552.277 y la solicitud internacional de patente No PCT/US95/95) demostraron que la expresión del gen para la glutatión S-transferasa (GST) Pi se pierde en casi todos los casos de cáncer de próstata. Ellos mostraron, además, que en veinte casos examinados, utilizando transferencia tipo Southern, que esta pérdida de expresión estaba acompañada por metilación en un sitio específico de la enzima de restricción (BssHIl) en la región promotora del gen. Esta metilación no se observó en tejido prostático normal o en un gran número de otros tejidos normales examinados. En el examen de una línea celular de cáncer de próstata en la que el gen para la GST-Pi está inactivo, también identificaron metilación en otros dos sitios de enzimas de restricción, Not\ y Sacll en la región promotora del gen. La digestión de los ADN de la línea celular con las enzimas Msp\ y Hpall, indicó que la correlación de la metilación del ADN con la falta de expresión no se mantuvo para estos sitios que se encuentran en gran parte secuencia abajo del sitio de inicio de la transcripción. La naturaleza de los datos hace que sea difícil llegar a conclusiones sobre el estado de metilación de sitios individuales de Msp\IHpa\\. Sin embargo, Lee et al. (18) fueron capaces de demostrar que después de la digestión con HpaW (que cortará en todos los sitios no metilados de Hpall), una región de ADN que contiene doce sitios de reconocimiento de Hpall podría ser amplificada por PCR a partir de ADN del tumor, pero no de próstata normal o ADN de leucocitos. Esto Indica que algunas moléculas de ADN en el cáncer de próstata están metiladas en todos estos sitios de Hpall, mientras que los ADN de próstata normal y el ADN de leucocitos deben contener al menos uno de estos sitios no metilados (ya que un solo corte hará que la región no pueda ser amplificada por PCR).
Los presentes Inventores han identificado y desarrollado un método alternativo para la detección de sitios de metilación presentes en el ADN de tejido de cáncer de próstata, pero no presentes en el ADN de tejido normal. El método se basa en la amplificación selectiva de una región objetivo del gen para la GST-Pi pero no requiere restricción previa con una enzima de restricción informativa.
Divulgación de la invención:
Por lo tanto, en un primer aspecto, la presente invención proporciona un ensayo de diagnóstico o pronóstico para cáncer de próstata caracterizado por metilación anormal de la citosina en un sitio dentro de la región del gen para glutatión S- transferasa (GST) Pi humana definido por cualquiera de los sitios CpG +1 a +33, en donde dicho ensayo comprende las
etapas de:
(i) aislar el ADN de dicho sujeto,
(ii) exponer dicho ADN aislado a los reactivos y condiciones para la amplificación de una región objetivo del gen para la GST-Pi y/o sus secuencias de flanqueo reguladoras, que incluyen un sitio o sitios en los que se presenta una metilación anormal de cisteína característica de la enfermedad o condición, siendo la amplificación selectiva porque sólo se amplifica la región objetivo si dicho sitio o sitios en los que se produce metilación anormal de citosina está/están metilados, y
(iii)... [Seguir leyendo]
Reivindicaciones:
1. Un ensayo de diagnóstico o de pronóstico para el cáncer de próstata en un sujeto, dicho cáncer de próstata caracterizado por metilación anormal de la citosina en un sitio dentro de la región del gen para la glutatión S-transferasa humana (GST) Pi definido por uno cualquiera de los sitios CpG +1 a +33, en donde dicho ensayo comprende las etapas de:
(i) aislar el ADN de dicho sujeto, y
(ii) determinar la presencia de metilación anormal de la citosina en un sitio individual dentro de la región del gen para la GST-Pi humana definido por uno cualquiera de los sitios CpG +1 a +33.
2. El ensayo de acuerdo con la reivindicación 1, en donde la región del gen para la GST-Pi dentro de la cual se determina la presencia de citosina metilada en un sitio individual es la región definida por cualquiera de los sitios CpG +9 a +33.
3. El ensayo de acuerdo con la reivindicación 1 o 2, en donde antes de la etapa de determinación, se trata el ADN aislado de tal manera que las citosinas no metiladas se convierten en uracilo u otro nucleótido capaz de formar un par de bases con adenina mientras que las citosinas metiladas permanecen sin cambios o se convierten en un nucleótido capaz de formar un par de bases con la guanina.
4. El ensayo de acuerdo con la reivindicación 3, en donde el tratamiento del ADN aislado implica hacer reaccionar el ADN aislado con bisulfito.
5. El ensayo de acuerdo con una cualquiera de las reivindicaciones 1 a 4, en donde la etapa de determinación implica la hibridación selectiva de una(s) sonda(s) de oligonucleótido / polinucleótido / ácido péptido-nucleico (PNA).
6. El ensayo de acuerdo con una cualquiera de las reivindicaciones 1 a 5, en donde dicho ADN se aísla a partir de células de tejido, sangre (incluyendo suero y plasma), semen, orina o linfa.
7. El ensayo de diagnóstico o pronóstico para el cáncer de próstata en un sujeto de acuerdo con la reivindicación 1, en donde la etapa (ii) de dicho ensayo comprende las etapas de:
(a) exponer dicho ADN aislado a los reactivos y condiciones para la amplificación de una región objetivo del gen para la GST-Pi humana que incluye un sitio individual en el cual ocurre la metilación anormal de citosina característica de cáncer de próstata, siendo la amplificación selectiva ya que sólo se amplifica la región objetivo si dicho sitio en el cual ocurre la metilación anormal de citosina está metilado, y
(b) determinar la presencia de ADN amplificado,
en donde la etapa de amplificación (a) se utiliza para amplificar una región objetivo dentro de la región del gen para la GST- Pi humana definida por cualquiera de los sitios CpG +1 a +33, y
en donde antes de la etapa de amplificación (a) se trata el ADN aislado de tal manera que las citosinas no metiladas se convierten en uracilo u otro nucleótido capaz de formar un par de bases con adenina, mientras que las citosinas metiladas permanecen sin cambios o se convierten en un nucleótido capaz de formar un par de bases con la guanina.
8. El ensayo de acuerdo con la reivindicación 7, en donde la etapa de amplificación implica la amplificación mediante la reacción en cadena de la polimerasa (PCR).
9. El ensayo de acuerdo con la reivindicación 8, en donde dicha amplificación por PCR utiliza un cebador inverso que incluye guanina en al menos un sitio por lo que, después de la hibridación del cebador inverso con el ADN tratado, dicha guanina o bien formará un par de bases con una citosina metilada (u otro nucleótido al cual ha sido convertida la citosina metilada a través de dicho tratamiento) si está presente, o formará una falta de correspondencia con uracilo (o otro nucleótido al cual ha sido convertido la citosina no metilada a través de dicho tratamiento).
1. El ensayo de acuerdo con la reivindicación 9, en donde dicha amplificación por PCR utiliza un cebador directo que incluye citosina en uno sitio correspondiente a un nucleótido citosina que está anormalmente metilado en el ADN.
11. El ensayo de acuerdo con la reivindicación 1, en donde los cebadores son de 12 a 3 nucleótidos de longitud.
12. El ensayo de acuerdo con la reivindicación 7, en donde el tratamiento del ADN aislado implica hacer reaccionar el ADN aislado con bisulfito.
13. El ensayo de acuerdo con la reivindicación 12, en donde la etapa de amplificación implica la amplificación mediante la reacción en cadena de la polimerasa (PCR).
14. El ensayo de acuerdo con la reivindicación 13, en donde dicha amplificación por PCR utiliza un cebador inverso que incluye guanina en al menos un sitio por el que, después de la hibridación del cebador inverso con el ADN tratado, dicha guanina o bien formará un par de bases con una citosina metilada, si está presente, o formará una falta de correspondencia con uracilo.
15. El ensayo de acuerdo con la reivindicación 14, en donde dicha amplificación por PCR utiliza un cebador directo que incluye citosina en al menos un sitio correspondiente a los nucleótidos de citosina que están anormalmente metilados en el ADN aislado.
16. El ensayo de acuerdo con la reivindicación 15, en donde los cebadores son de 12 a 3 nucleótidos de longitud.
17. El ensayo de acuerdo con la reivindicación 16, en donde los cebadores se seleccionan con el fin de hibridar con una secuencia dentro de la región objetivo que incluye dos a cuatro nucleótidos de citosina que están anormalmente metilados en el ADN.
18. El ensayo de acuerdo con una cualquiera de las reivindicaciones 7 a 17, en donde dicho ADN se aisló a partir de células de tejido, sangre (incluyendo suero y plasma), semen, orina o linfa.
19. El ensayo de acuerdo con la reivindicación 7, en donde la etapa de amplificación se utiliza para amplificar una región objetivo dentro de la región del gen para la GST-Pi definido por (e inclusive de) sitios CpG +9 a +33.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
Anticuerpos del OPGL, del 15 de Julio de 2020, de AMGEN FREMONT INC.: Un anticuerpo, que comprende una cadena pesada y una cadena ligera, donde: a) la cadena pesada comprende: 1) una secuencia de aminoácidos recogida […]
Anticuerpo anti-FGF23 y composición farmacéutica que comprende el mismo, del 15 de Julio de 2020, de Kyowa Kirin Co., Ltd: Anticuerpo o fragmento funcional del mismo que se une a la totalidad o a una parte del epítopo de FGF23 humano, al que se une un anticuerpo producido […]
Biblioteca de péptidos y su uso, del 8 de Julio de 2020, de DAIICHI SANKYO COMPANY, LIMITED: Una biblioteca de péptidos que comprende una pluralidad de péptidos diferentes en la que los péptidos comprenden cada uno una secuencia de aminoácidos […]
Producción de vectores de expresión y selección de células de alta capacidad de procesamiento, del 8 de Julio de 2020, de Kymab Limited: Un método para producir células que codifican un repertorio de anticuerpos que comprende cadenas pesadas y cadenas ligeras de anticuerpo cognadas, comprendiendo dicho […]
Vacuna de ADN contra pseudotuberculosis en peces marinos, del 1 de Julio de 2020, de NATIONAL UNIVERSITY CORPORATION TOKYO UNIVERSITY OF MARINE SCIENCE AND TECHNOLOGY: Una vacuna de ADN para peces, caracterizada por: - impartir inmunidad contra la pseudotuberculosis causada por Photobacterium damselae subsp. piscicida - que comprende, […]
Vacuna de ADN que contiene un epítopo específico de VEGF y/o un epítopo específico de angiopoyetina-2, del 1 de Julio de 2020, de OSAKA UNIVERSITY: Un vector de expresión que codifica un polipéptido del antígeno del núcleo del virus de la hepatitis B quimérico con una inserción para uso en el tratamiento o la profilaxis […]
Anticuerpo anti-Notch 4 humano, del 1 de Julio de 2020, de EISAI R&D MANAGEMENT CO., LTD: Un anticuerpo anti-Notch4 o un fragmento de unión a Notch4 de este, donde dicho anticuerpo o un fragmento de unión a Notch4 de este comprende cadenas pesadas y ligeras y […]