Detección de Staphylococcus aureus resistente a meticilina.
Un procedimiento de detección en una muestra de Staphylococcus aureus resistente a meticilina (MRSA) que tiene una inserción de un módulo SCCmec en el ADN cromosómico de Staphylococcus aureus,
que comprende
efectuar en la muestra una reacción de amplificación y detección utilizando:
a) un primer cebador capaz de hibridar específicamente con una región de conexión del extremo del módulo SCCmec,
b) un segundo cebador capaz de hibridar específicamente con una región de conexión del extremo del ADN cromosómico de Staphylococcus aureus, y
c) una sonda capaz de hibridar específicamente con una región del módulo SCCmec entre la región con que el primer cebador es capaz de hibridar y la conexión,
en el que cada uno del primer cebador y el segundo cebador está orientado de tal modo que, en las condiciones de amplificación, se amplifica la conexión, y
en el que si la muestra contiene MRSA, se detecta la hibridación de la sonda.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/US2008/013922.
Solicitante: BIOMERIEUX SA.
Nacionalidad solicitante: Estados Unidos de América.
Dirección: 100 RODOPHE STREET DURHAM, NC 27712 ESTADOS UNIDOS DE AMERICA.
Inventor/es: JAY, CORINNE, DEIMAN,BIRGIT, VAN STRIJP,DIANNE, VAN DE WIEL,PAUL.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2425267_T3.pdf
Fragmento de la descripción:
Detección de Staphylococcus aureus resistente a meticilina Campo de la invención La presente invención se refiere a la detección molecular de Staphylococcus aureus resistente a meticilina (MRSA) . Más particularmente, la presente invención se refiere a una detección mejorada de MRSA que reduce los resultados falsos positivos.
Antecedentes de la invención El Staphylococcus aureus resistente a meticilina (MRSA) es un patógeno intrahospitalario, pero también extrahospitalario, importante que puede causar graves infecciones tales como infecciones de heridas quirúrgicas, neumonía, endocarditis y septicemia. La resistencia a meticilina es debida a la presencia del gen mecA que codifica una proteína de unión a penicilina modificada, PBP2a o PBP2', con afinidad reducida por fármacos 1-lactámicos. El gen mecA está portado por un módulo llamado SCCmec (“Staphylococcal Cassette Chromosome mec”; Ito et al., 2001, Antimicrob. Agents Chemother. 5 (5) : 1323-1336; Hiramatsu, et al., 2001, Trends Microbiol. oct; 9 (10) : 486-93) , un elemento móvil que puede incorporarse al cromosoma de S. aureus y otros estafilococos negativos de coagulasa, principalmente S. epidermidis y S. haemolyticus. SCCmec se caracteriza por la presencia de repeticiones terminales inversas y directas, un conjunto de genes de recombinasa específicos de sitio (ccrA y ccrB) y el complejo del gen mecA (Ito et al., 1999, Antimicrob. Agents Chemother. 43: 1449-1458; Katayama et al., 2000, Antimicrob. Agents Chemother. 44: 1549-1555) . El sitio de inserción de este módulo SCCmec del gen mecA en el genoma de Staphylococcus aureus es conocido y la secuencia está conservada (Ito et al., 2001, Antimicrob. Agents Chemother.
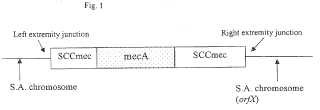
45: 1323-1336) . Después de la inserción en el cromosoma de S. aureus, el SCCmec tiene una región de conexión del extremo izquierdo y una región de conexión del extremo derecho (véase la FIGURA 1) , donde la secuencia de SCCmec es contigua a la secuencia cromosómica de S. aureus. Se ha analizado anteriormente la secuencia nucleotídica de las regiones que rodean los límites izquierdo y derecho del ADN de SCCmec (concretamente, attL y attR, respectivamente) , así como aquellas de las regiones alrededor del sitio de integración de ADN de SCCmec (concretamente, attBscc, el sitio de enlazamiento de cromosoma bacteriano para ADN de SCCmec) . El análisis de secuencia de estos sitios de integración reveló que attBscc está localizado en el extremo 3’ de un marco de lectura abierto (ORF) novedoso, orfX. orfX codifica un presunto polipéptido de 159 aminoácidos que exhibe homología de secuencia con algunos polipéptidos anteriormente identificados de función desconocida (Ito et al., 1999, Antimicrob. Agents Chemother. 43: 1449-1458) . Se ha estudiado adicionalmente la organización de la región mecA de SCCmec (Oliveira, D.C., et al., 2000, Antimicrob. Agents Chemother. 44 (7) : 1906-1910) .
El MRSA puede portarse por personas sanas sin causar la enfermedad, pero estos portadores sanos, cuando entran en un hospital, pueden contaminar a pacientes hospitalizados. Adicionalmente, un paciente puede contaminarse a sí mismo, por ejemplo, si experimenta cirugía aumenta el riesgo de infección. Los portadores sanos de MRSA constituyen un reservorio de MRSA y debe efectuarse el cribado de estos portadores para erradicar las cepas mediante descontaminación local. El cribado de MRSA está reconocido actualmente como una herramienta importante para reducir la prevalencia de MRSA en el mundo. Típicamente, en un ensayo de MRSA en un paciente, se toma un frotis nasal del paciente y se cultiva repetidamente, para determinar si está presente una cepa de MRSA. La necesidad de cultivar podría obviarse mediante un ensayo para identificar MRSA directamente del frotis nasal. Los procedimientos de identificación de cultivo requieren típicamente como mínimo 24 horas, y más típicamente 72 horas, para obtener resultados. Los nuevos medios cromogénicos (que tienen uno o varios sustratos en los medios y, típicamente, antibiótico (por ejemplo cefoxitina) para seleccionar las cepas resistentes a meticilina) pueden limitar potencialmente este tiempo hasta el resultado a un periodo de tiempo de 24-48 horas. Sin embargo, en el caso de infección por MRSA, se requieren resultados en cuestión de horas, puesto que el paciente debería aislarse hasta obtener los resultados. Por lo tanto, es altamente deseable una prueba de MRSA molecular fiable que pueda proporcionar resultados en cuestión de 2-4 horas.
La amplificación es una técnica bien conocida, y se han desarrollado diversos procedimientos que incluyen amplificación basada en la transcripción, tal como amplificación mediada por la transcripción (TMA; patentes de EE.UU. nº 5.766.849, 5.399.491, 5.480.784, 5.766.849 y 5.654.142) y amplificación basada en secuencias de ácido nucleico (NASBA; documentos 5.130.238, 5.409.818, 5.654.142 y 6.312.928) , y tecnologías de amplificación de ácidos nucleicos por ciclado (termociclado) tales como reacción en cadena de la polimerasa (PCR; patentes de EE.UU. nº 4.683.195, 4.965.188, 4.683.202) y reacción en cadena de la ligasa (LCR; patente de EE.UU. nº 5.792.607) . Los procedimientos de amplificación conocidos incluyen también la amplificación por desplazamiento de hebra (SDA) , la replicación de secuencia automantenida (3SR) , la replicasa Q-1 y la amplificación en círculo rodante en cascada (CRCA) .
Los procedimientos de detección que utilizan ácidos nucleicos son también bien conocidos en la materia. Los ácidos nucleicos a menudo se marcan con diversos fines de detección. Por ejemplo, los procedimientos descritos en las patentes de EE.UU. nº 4.486.539 (Kourlisky) , 4.411.955 (Ward) , 4.882.269 (Schneider) y 4.213.893 (Carrico) , ilustran la preparación de sondas de detección marcadas para detectar secuencias de ácido nucleico específicas. Se han descrito también diseños de sonda para diferentes procedimientos de detección, tales como captura de diana, HPA, TAQman, balizas moleculares e hibridación de tipo sándwich (por ejemplo, patentes de EE.UU. nº 4.486.539 y 4.751.177, 5.210.015, 5.487.972, 5.804.375, 5.994.076) . Las técnicas y condiciones de hibridación de ácidos nucleicos son conocidas por el experto en la materia y se han descrito, por ejemplo, en Sambrook et al. “Molecular Cloning A Laborator y Manual”, 2ª Ed. Cold Spring Lab. Press, diciembre de 1989; patentes de EE.UU. nº 4.563.419 (Ranki) y 4.851.330 (Kohne) y en Dunn, et al., Cell 12, pág. 23-26 (1978) entre otras muchas publicaciones. Son también conocidos diseños de sonda para diferentes procedimientos de detección, tales como de captura de diana, HPA, TaqMan, balizas moleculares e hibridación de tipo sándwich (por ejemplo, patentes de EE.UU. nº 4.486.539 y 4.751.177, 5.210.015, 5.487.972, 5.804.375, 5.994.076) .
Se han descrito procedimiento moleculares anteriores para detectar e identificar MRSA basándose en la detección del gen mecA y de secuencias cromosómicas específicas de S. aureus (Saito et al., 1995, J. Clin. Microbiol. 33: 2498-2500; Ubukata et al., 1992, J. Clin. Microbiol. 30: 1728-1733; Murakami et al., 1991, J. Clin. Microbiol. 29: 2240-2244; Hiramatsu et al., 1992, Microbiol. Immunol. 36: 445-453) . Sin embargo, los resultados positivos de la presencia en una muestra tanto del gen mecA como de secuencias cromosómicas de S. aureus no pueden garantizar que el MRSA esté presente puesto que, por ejemplo, en pruebas basadas en la detección de mecA y marcadores específicos de S. aureus, pueden observarse falsos positivos en presencia de MSSA y estafilococos negativos de coagulasa resistentes a meticilina que poseen el gen mecA. Además, en pruebas basadas en la detección de solo la conexión del módulo, se han observado falsos positivos con aislamientos de S. aureus sensibles a meticilina que contienen un pequeño fragmento del extremo derecho de SCCmec (véase Rupp, J. et al.,
J. Clin. Microbiol. 44 (6) : 2317 (2006) ) . Adicionalmente, Ramakrishnan y Riccelli describen un procedimiento para la detección de MRSA que utiliza sondas oligonucleotídicas que tienen secuencias que son complementarias de regiones cercanas a la conexión izquierda del sitio de inserción del módulo SCCmec, incluyendo parte de la secuencia del módulo SCCmec y parte de la secuencia de S. aureus en la región de inserción (la región de conexión del extremo izquierdo) (publicación de patente de EE.UU. nº US20060057613) . Zhang et al. J. Clin. Microbiol. 43 (10) : 5026-5033 (2005) describen un procedimiento de PCR múltiple para detectar MRSA usando cebadores orientados al gen mecA y al módulo SCCmec de tipo I, II, III, IV y V.
Sin embargo, los intentos anteriores de determinar MRSA mediante procedimientos moleculares han tenido dificultades... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento de detección en una muestra de Staphylococcus aureus resistente a meticilina (MRSA) que tiene una inserción de un módulo SCCmec en el ADN cromosómico de Staphylococcus aureus, que comprende efectuar en la muestra una reacción de amplificación y detección utilizando: a) un primer cebador capaz de hibridar específicamente con una región de conexión del extremo del módulo SCCmec,
b) un segundo cebador capaz de hibridar específicamente con una región de conexión del extremo del ADN
cromosómico de Staphylococcus aureus, y c) una sonda capaz de hibridar específicamente con una región del módulo SCCmec entre la región con que el primer cebador es capaz de hibridar y la conexión,
en el que cada uno del primer cebador y el segundo cebador está orientado de tal modo que, en las condiciones de amplificación, se amplifica la conexión, y en el que si la muestra contiene MRSA, se detecta la hibridación de la sonda.
2. El procedimiento de la reivindicación 1, que comprende además la etapa de efectuar en la muestra una reacción de amplificación y detección que detecta la presencia del gen mecA,
en el que, si la muestra contiene MRSA, se detecta la presencia tanto de la conexión diana como de mecA en la muestra.
3. El procedimiento de la reivindicación 2, en el que la reacción de amplificación y detección de la región de conexión del extremo y la reacción de amplificación y detección del gen mecA se efectúan simultáneamente.
4. Un procedimiento de detección en una muestra de Staphylococcus aureus resistente a meticilina (MRSA) que tiene una inserción de un módulo SCCmec en el ADN cromosómico de Staphylococcus aureus, comprendiendo el procedimiento
a) efectuar en una muestra una reacción de amplificación múltiple que puede amplificar tanto (1) una conexión de un módulo SCCmec insertado y ADN cromosómico de Staphylococcus aureus como (2) una región de mecA,
en el que la amplificación de la conexión se efectúa utilizando: 1) un primer cebador capaz de hibridar específicamente con una región de conexión del extremo del módulo SCCmec,
2) un segundo cebador capaz de hibridar específicamente con una región de conexión del extremo del ADN
cromosómico de Staphylococcus aureus, y 3) una primera sonda capaz de hibridar específicamente con una región del módulo SCCmec entre la región con que el primer cebador es capaz de hibridar específicamente y la conexión,
en el que, en las condiciones de amplificación, se amplifica la conexión y b) detectar, en los productos de amplificación, la presencia o ausencia de cada una de la conexión y mecA, en el que, si la muestra contiene MRSA, se detecta la presencia tanto de la conexión como de mecA en la muestra.
5. El procedimiento de la reivindicación 4, que comprende las etapas de: poner en contacto, en un solo recipiente, la muestra con a) un primer conjunto de oligonucleótidos que comprende
(1) un primer oligonucleótido que tiene una secuencia de ácido nucleico que hibrida específicamente con una región de conexión del extremo de un módulo SCCmec, y
(2) un segundo oligonucleótido que tiene una secuencia de ácido nucleico que hibrida específicamente con una región de ADN cromosómico de Staphylococcus aureus que flanquea dicho módulo SCCmec, formando un primer producto de reacción de la muestra y el primer y segundo oligonucleótidos, y
b) un segundo conjunto de nucleótidos que comprende
(3) un tercer oligonucleótido que tiene una secuencia nucleotídica que hibrida específicamente con una primera región del ácido nucleico de mecA, y
(4) un cuarto oligonucleótido que tiene una secuencia nucleotídica que hibrida específicamente con una segunda región del ácido nucleico de mecA, formando un segundo producto de reacción de la muestra y el tercero y cuarto oligonucleótidos; e
identificar la presencia de MRSA detectando tanto el primero como el segundo productos de reacción.
6. El procedimiento de la reivindicación 5, en el que la etapa de puesta en contacto comprende además efectuar una reacción de amplificación usando los conjuntos de oligonucleótidos (a) y (b) como cebadores, y en el que dicha etapa de identificación comprende detectar la presencia o ausencia de un producto de amplificación de ambos conjuntos de oligonucleótidos.
7. El procedimiento de la reivindicación 6, en el que la etapa de identificación comprende poner en contacto el primer y segundo productos de reacción con (1) una primera sonda capaz de hibridar específicamente con el primer producto de reacción y (2) una segunda sonda capaz de hibridar específicamente con el segundo producto de reacción.
8. El procedimiento de una cualquiera de las reivindicaciones 1-7, en el que la sonda o primera sonda comprende más de una sonda, preferiblemente cinco o más sondas, cada una capaz de hibridar específicamente con un tipo diferente de MRSA.
9. El procedimiento de una cualquiera de las reivindicaciones 1-7, en el que la reacción de amplificación se selecciona del grupo consistente en amplificación basada en la transcripción, preferiblemente NASBA, más preferiblemente NASBA-ADN, y PCR.
10. El procedimiento de una cualquiera de las reivindicaciones 1-7, en el que el primer cebador o primer oligonucleótido comprende más de un cebador u oligonucleótido capaz de hibridar con una región de conexión del extremo del módulo SCCmec.
11. El procedimiento de una cualquiera de las reivindicaciones 1-7, en el que la región de conexión del extremo es la región de conexión del extremo derecho.
12. El procedimiento de la reivindicación 11, en el que el segundo cebador o segundo oligonucleótido comprende el ácido nucleico expuesto en la SEQ ID NO 13.
13. El procedimiento de una cualquiera de las reivindicaciones 1-7, en el que la sonda o primera sonda comprende un ácido nucleico seleccionado del grupo consistente en las SEQ ID NO: 7, 8, 9, 10, 11 y 12, preferiblemente al menos cinco ácidos nucleicos, comprendiendo cada uno una secuencia diferente seleccionada del grupo consistente en las SEQ ID NO: 7, 8, 9, 10, 11 y 12.
14. El procedimiento de una cualquiera de las reivindicaciones 1-7, en el que el primer cebador o primer oligonucleótido comprende un ácido nucleico que comprende una secuencia seleccionada del grupo consistente en las SEQ ID NO: 1, 2, 3, 4, 5 y 6, preferiblemente al menos cinco ácidos nucleicos, comprendiendo cada uno una secuencia diferente seleccionada del grupo consistente en las SEQ ID NO: 1, 2, 3, 4, 5 y 6.
15. El procedimiento de una cualquiera de las reivindicaciones 2-7, en el que la amplificación de mecA utiliza un cebador u oligonucleótido de mecA que comprende un ácido nucleico seleccionado del grupo consistente en las SEQ ID NO: 15 y 16.
16. El procedimiento de una cualquiera de las reivindicaciones 2-7, en el que la detección de mecA utiliza una sonda de mecA que comprende el ácido nucleico expuesto en la SEQ ID NO: 14.
17. El procedimiento de una cualquiera de las reivindicaciones 5-7, en el que el segundo oligonucleótido hibrida específicamente con una región de orfX.
18. Un kit de detección de Staphylococcus aureus resistente a meticilina (MRSA) que tiene una inserción de un módulo SCCmec en el ADN cromosómico de Staphylococcus aureus, que comprende:
a) un primer cebador capaz de hibridar específicamente con una región de conexión del extremo derecho del módulo SCCmec,
b) un segundo cebador capaz de hibridar específicamente con una región de conexión del extremo derecho del ADN cromosómico de Staphylococcus aureus, y
c) una sonda seleccionada del grupo consistente en (1) una sonda capaz de hibridar específicamente principalmente con una región del módulo SCCmec entre la región con que el primer cebador es capaz de hibridar y la conexión, y
(2) una sonda capaz de hibridar específicamente del todo con una región del módulo SCCmec entre la región con que el primer cebador es capaz de hibridar y la conexión,
en el que cada uno del primer cebador y el segundo cebador está orientado de tal modo que, en las condiciones de amplificación, se amplifica la conexión.
19. El kit de la reivindicación 18, que comprende: a) un primer conjunto de oligonucleótidos de amplificación y detección que comprende 1) un primer cebador capaz de hibridar específicamente con una región de conexión del extremo derecho del módulo SCCmec,
2) un segundo cebador capaz de hibridar específicamente con el ADN cromosómico de Staphylococcus aureus en la región de conexión del extremo y 3) una primera sonda capaz de hibridar específicamente del todo con una región del módulo SCCmec entre la región con que el primer cebador es capaz de hibridar y la conexión,
en el que cada uno del primer cebador y el segundo cebador está orientado de tal modo que, en las condiciones de amplificación, se amplifica la conexión, y b) un segundo conjunto de oligonucleótidos de amplificación y detección que comprende 4) un tercer cebador capaz de hibridar específicamente con una primera región de mecA, 5) un cuarto cebador capaz de hibridar específicamente con una segunda región de mecA, y 6) una segunda sonda capaz de hibridar específicamente con una región de mecA entre la primera y la segunda regiones de mecA, en el que cada uno del tercer cebador y el cuarto cebador está orientado de tal modo que, en las condiciones de amplificación, se amplifica la región de mecA entre la primera y segunda regiones de mecA.
20. El kit de la reivindicación 18 o 19, en el que el primer cebador comprende más de un cebador capaz de hibridar con la región de conexión del extremo derecho del módulo SCCmec.
21. El kit de la reivindicación 18 o 19, en el que el primer cebador es un ácido nucleico que comprende una secuencia seleccionada del grupo consistente en: SEQ ID NO: 1, 2, 3, 4, 5 y 6.
22. El kit de la reivindicación 18 o 19, en el que el segundo cebador es capaz de hibridar específicamente con orfX.
23. El kit de la reivindicación 18 o 19, en el que el segundo cebador comprende el ácido nucleico expuesto en la SEQ ID NO: 13.
24. El kit de la reivindicación 18 o 19, en el que la sonda o primera sonda comprende más de una sonda, preferiblemente cinco o más sondas, cada una capaz de hibridar específicamente con un tipo diferente de MRSA.
25. El kit de la reivindicación 18 o 19, en el que la sonda o primera sonda es un ácido nucleico que comprende una secuencia seleccionada del grupo consistente en las SEQ ID NO: 7, 8, 9, 10, 11 y 12.
26. El kit de la reivindicación 18 o 19, en el que el primer, segundo, tercer, cuarto y quinto cebadores se proporcionan en un solo recipiente.
27. El kit de la reivindicación 18 o 19, en el que el primer y segundo conjuntos de oligonucleótidos de amplificación y detección se proporcionan en un solo recipiente.
28. El kit de la reivindicación 18 o 19, en el que el tercer y/o cuarto cebadores comprenden un ácido nucleico seleccionado del grupo consistente en las SEQ ID NO: 15 y 16.
29. El kit de la reivindicación 18 o 19, en el que la segunda sonda comprende el conjunto de ácidos nucleicos expuesto en la SEQ ID NO: 14.
30. Una composición oligonucleotídica que comprende:
(1) Un primer cebador oligonucleotídico que tiene una secuencia de ácido nucleico que hibrida específicamente con una región de conexión del extremo de un módulo SCCmec;
(2) Un segundo cebador oligonucleotídico que tiene una secuencia de ácido nucleico que hibrida específicamente con una región de ADN cromosómico de Staphylococcus aureus que flanquea dicho módulo SCCmec; y
(3) Una primera sonda capaz de hibridar específicamente con una región del módulo SCCmec entre la región con que el primer cebador es capaz de hibridar y la conexión,
en el que cada uno del primer cebador y el segundo cebador está orientado de tal modo que, en las condiciones de
amplificación, se amplifica la conexión.
31. La composición oligonucleotídica de la reivindicación 30, que comprende además:
(4) un tercer oligonucleótido que tiene una secuencia nucleotídica que hibrida específicamente con una primera región del ácido nucleico de mecA;
(5) un cuarto oligonucleótido que tiene una secuencia nucleotídica que hibrida específicamente con una segunda región del ácido nucleico de mecA; y
(6) una segunda sonda capaz de hibridar específicamente con una región de mecA entre la primera y segunda regiones de mecA.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]