Constructos de ADN para la inhibición específica de la expresión génica mediante interferencia de ARN.
Constructo de expresión de ADN para la inhibición selectiva de la expresión génica por medio de interferencia deARN constituido por los siguientes componentes
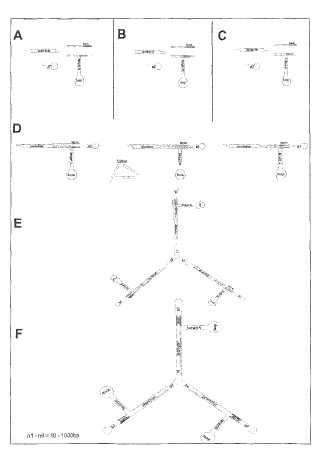
a) una primera estructura de horquilla de terminación de ADN,
en la que una cadena sencilla dedesoxirribonucleótidos está replegada para dar una región bicatenaria de manera que ésta presenta en unextremo una proyección cohesiva y en el extremo opuesto un bucle monocatenario,
b) una segunda estructura de horquilla de terminación, que está estructurada de manera análoga a la primeraestructura de horquilla de terminación,
c) al menos una estructura de ADN en forma de T con tres brazos de ADN bicatenario que están unidos através de uniones Holliday, en la que
i. un brazo contiene las secuencias que van a transcribirse y las cadenas sencillas de este brazo de cadenadoble de ADN en el extremo que está opuesto a la unión con los otros brazos, están unidos entre símediante un bucle de ADN monocatenario, y
ii. los otros dos brazos presentan extremos cohesivos, presentando un brazo una secuencia de terminaciónadecuada y el otro brazo una secuencia de promotor adecuada.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/DE2006/002083.
Solicitante: MOLOGEN AG.
Nacionalidad solicitante: Alemania.
Dirección: FABECKSTRASSE 30 14195 BERLIN ALEMANIA.
Inventor/es: SCHROFF, MATTHIAS, OSWLAD,DETLEF.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12N15/11 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12N MICROORGANISMOS O ENZIMAS; COMPOSICIONES QUE LOS CONTIENEN; PROPAGACION, CULTIVO O CONSERVACION DE MICROORGANISMOS; TECNICAS DE MUTACION O DE INGENIERIA GENETICA; MEDIOS DE CULTIVO (medios para ensayos microbiológicos C12Q 1/00). › C12N 15/00 Técnicas de mutación o de ingeniería genética; ADN o ARN relacionado con la ingeniería genética, vectores, p. ej. plásmidos, o su aislamiento, su preparación o su purificación; Utilización de huéspedes para ello (mutantes o microorganismos modificados por ingeniería genética C12N 1/00, C12N 5/00, C12N 7/00; nuevas plantas en sí A01H; reproducción de plantas por técnicas de cultivo de tejidos A01H 4/00; nuevas razas animales en sí A01K 67/00; utilización de preparaciones medicinales que contienen material genético que es introducido en células del cuerpo humano para tratar enfermedades genéticas, terapia génica A61K 48/00; péptidos en general C07K). › Fragmentos de ADN o de ARN; sus formas modificadas (ADN o ARN no empleado en tecnología de recombinación C07H 21/00).
- C12N15/113 C12N 15/00 […] › Acidos nucleicos no codificantes que modulan la expresión de genes, p.ej. oligonucleótidos antisentido.
PDF original: ES-2421314_T3.pdf
Fragmento de la descripción:
Constructos de ADN para la inhibición específica de la expresión génica mediante interferencia de ARN
La invención se refiere a procedimientos para la preparación de vectores que son adecuados para inhibir tras su transfección en células eucariotas de manera selectiva la formación de una o varias proteínas mediante interferencia de ARN así como a vectores de este tipo.
Una posibilidad recientemente probada de la inhibición de la expresión génica se basa en la generación de moléculas de ARN bicatenarias. Con este ARN bicatenario (ARNbc) pueden desactivarse de manera selectiva genes individuales de manera altamente eficaz y más rápidamente que con cualquier otro procedimiento, sin alterar la formación de proteínas de genes adyacentes. El principio subyacente se designa como interferencia de ARN, de manera abreviada iARN. La secuencia de ARNbc que causa este fenómeno como ARNip (small interference RNA, ARN de interferencia pequeño) .
El ARNip no impide la lectura del gen, sino que conecta un mecanismo propio de la célula que impide los ARNm leídos por el gen y así la formación de correspondientes proteínas (post-transcriptional gene silencing, silenciamiento génico postranscripcional) .
Esta degradación de ARNm selectiva se activa mediante moléculas cortas de ARNip con 19-23 bases de ARN de longitud, que son homólogas al ARNm diana, cuya conversión en una proteína debe impedirse. Las moléculas de ARNip se acoplan con endorribonucleasas especiales para dar un complejo de ARN-proteína propio de la célula con la denominación “RISC” (RNA induced silencing complexes, complejos de silenciamiento inducidos por ARN) . En la construcción de estos complejos se disocian las dos cadenas de ARN una de otra, de manera que se producen los denominados RISC activados que contienen respectivamente una cadena sencilla de la molécula de ARNip. Los RISC activados que contienen la cadena antisentido, que es complementaria al ARNm diana, se unen a éste y la endorribonucleasa del complejo ARN-proteína se ocupa ahora de la degradación de ARNm específica de secuencia.
El ARNip puede generarse en la célula de manera experimental o puede introducirse mediante infiltración desde fuera. Por un lado se logra esto a través de moléculas de ARNip preparadas sintéticamente que pueden administrarse tanto in-vitro como in-vivo.
Este procedimiento tiene sin embargo límites técnicos. Además de la inestabilidad general del ARNip sintético en el medio como también en la célula, es posible la inhibición por medio de ARNip sintético en principio sólo de manera temporal y muchas células (por ejemplo células neuronales) solo pueden transfectarse de manera muy ineficaz. Los estudios que se basan en la transfección de ARNip sintético están limitados, por tanto, por regla general tanto temporalmente a 1 - 5 días como específicamente al tipo de célula. Además son desventajosos los altos costes de producción y la larga duración de producción.
El documento WO 2004/044135 da a conocer compuestos oligoméricos para la modulación de la expresión génica por medio de interferencia de ARN. A este respecto, los compuestos oligoméricos comprenden dos oligómeros que hibridan entre sí al menos parcialmente y de los cuales uno puede hibridar a su vez con ácidos nucleicos diana para suprimir la expresión génica. Al menos uno de los oligómeros tiene una estructura secundaria no lineal o es parte de una disposición oligomérica múltiple.
Sin embargo, ARNip puede generarse también mediante vectores en la célula. A este respecto se trata de vectores virales o a base de plásmido que conducen solamente en la célula mediante expresión a la formación de las secuencias de ARNip. Las ventajas en comparación con la transfección con ARNip sintético se encuentran en la transcripción más estable y eventualmente más regulada de la correspondiente secuencia de ARNip.
Sin embargo, los vectores a base de plásmido muestran además de una baja eficacia de transfección igualmente un procedimiento de producción costoso. Así es necesario seleccionar clones estables. En este procedimiento con frecuencia de larga duración, que puede durar semanas e incluso también meses, se llega con frecuencia a la aparición de numerosas dificultades potenciales que son inherentes de experimentos de clonación. Para la comprobación del producto son necesarias secuenciaciones que igualmente son laboriosas y caras.
Además, los vectores a base de plásmido contienen genes resistentes a antibióticos que son necesarios para su selección. Por este motivo no son adecuados vectores de este tipo para su uso en organismos vivos. La posible recombinación con bacterias que se producen de manera ubicua en el organismo, encierra el riesgo de una aparición creciente de bacterias resistentes a antibióticos. La extensión de resistencias a antibióticos es un problema grave y no asumible.
Dado que los vectores virales pueden realizar la transfección de manera eficaz y selectiva, éstos ofrecen una ventaja en comparación con moléculas de ARNip sintéticas como también vectores a base de plásmido.
Para la aplicación terapéutica pueden usarse vectores virales de este tipo, aunque con reservas. También en este caso la recombinación de secuencias virales con virus que se producen en la naturaleza representa un riesgo de seguridad inherente, dado que puede temerse la generación de nuevos virus híbridos patógenos. Además, su
preparación es también costosa y cara.
Dado que ya únicamente la inhibición de la expresión de sólo un producto génico usando sistemas de expresión está unida con una multiplicidad de complicaciones, se entiende casi por sí mismo que la desactivación simultánea de la expresión de varios productos génicos en una célula o un tejido ha de obtenerse claramente de manera más compleja y por consiguiente más difícil.
La baja probabilidad de que una célula se transfecte únicamente por solo un constructo representa uno de los problemas principales en la inhibición múltiple de la expresión génica mediante interferencia de ARN, para el caso del uso de varios constructos independientes. Esta baja probabilidad se potencia negativamente con el número de constructos usados. Con frecuencia, sin embargo, es necesaria para determinados planteamientos terapéuticos genéticos una inhibición controlada, simultánea de la expresión génica.
Existe una multiplicidad de procedimientos que permiten la cotranscripción de varios ARN en una célula. El procedimiento más sencillo es la cotransfección de dos constructos de expresión independientes (a continuación también: vectores) . Otro procedimiento consiste en la transfección con un vector que porta dos casetes de expresión independientes. Los constructos de este tipo pueden usarse también para la generación de ARNip.
Sin embargo, en las dos posibilidades existe el riesgo de que los transcriptos se diferencien significativamente con respecto a su número, procesamiento, tiempo de vida medio y efectividad traduccional y por consiguiente en la cantidad de la proteína expresada, de modo que su uso esté caracterizado por la inefectividad y la mala reproducibilidad. Por tanto, el uso de sistemas de expresión de este tipo conduciría también a una transcripción de distinta intensidad de ARNip, de manera que por último se obtendría una inhibición desigual de los respectivos genes.
La construcción de ARN di y policistrónicos mediante el uso de elementos IRES (Internal Ribosome Entr y Site, sitio interno de entrada en el ribosoma) representa otra posibilidad para la coexpresión de varios genes. Estos permiten la iniciación de la traducción mediante ribosomas independientemente de la estructura de caperuza de ARNm mediante elementos de secuencia. Por primera vez se descubrieron elementos IRES en el ARNm del picornavirus (Pelletier y Sonenberg, 1988, Nature 334: 320-325, Jang et al., 1988, Journal Virology 62: 2636-2643) .
Para la construcción de vectores bicistrónicos se usan lo más frecuentemente los elementos IRES del poliovirus y del VEMC (virus de la encefalomiocarditis) (Dirks et al., 1993, Gene 128: 247-249) . De manera desventajosa varía su efectividad mucho dependiendo de la línea celular usada (Borman et al., 1997, Nucleic Acids Res. 25:925-932) .
La mayoría de los casetes de expresión bicistrónicos disponibles están constituidos por un marcador que puede seleccionarse o un gen indicador, que están dispuestos en el lado 3’ del elemento IRES y un MCS (Multiple Cloning Site, sitio de clonación múltiple) para la inserción del gen deseado (Dirks et al., 1993, Gene 128: 247-249) . Igualmente se conocen sistemas de expresión tri y policistrónicos... [Seguir leyendo]
Reivindicaciones:
1. Constructo de expresión de ADN para la inhibición selectiva de la expresión génica por medio de interferencia de ARN constituido por los siguientes componentes a) una primera estructura de horquilla de terminación de ADN, en la que una cadena sencilla de desoxirribonucleótidos está replegada para dar una región bicatenaria de manera que ésta presenta en un extremo una proyección cohesiva y en el extremo opuesto un bucle monocatenario, b) una segunda estructura de horquilla de terminación, que está estructurada de manera análoga a la primera estructura de horquilla de terminación, c) al menos una estructura de ADN en forma de T con tres brazos de ADN bicatenario que están unidos a través de uniones Holliday, en la que i. un brazo contiene las secuencias que van a transcribirse y las cadenas sencillas de este brazo de cadena doble de ADN en el extremo que está opuesto a la unión con los otros brazos, están unidos entre sí mediante un bucle de ADN monocatenario, y
ii. los otros dos brazos presentan extremos cohesivos, presentando un brazo una secuencia de terminación adecuada y el otro brazo una secuencia de promotor adecuada.
2. Constructo de expresión de ADN para la inhibición selectiva de la expresión génica por medio de interferencia de ARN constituido por los siguientes componentes
a) una primera estructura de horquilla de terminación de ADN, en la que una cadena sencilla de desoxirribonucleótidos está replegada para dar una región bicatenaria de manera que ésta presenta un extremo con proyección cohesiva y en el extremo opuesto las cadenas sencillas que forman la cadena doble están unidas entre sí mediante un bucle monocatenario, b) una segunda estructura de horquilla de terminación, que está estructurada de manera análoga a la primera estructura de horquilla de terminación y cuya región bicatenaria y monocatenaria está constituida por la secuencia que va a transcribirse c) una secuencia de promotor adecuada y una secuencia de terminación complementaria inversa adecuada que se encuentran entre las dos estructuras de horquilla de terminación descritas en a) y b) en una cadena doble de ADN.
3. Constructo de expresión de ADN según la reivindicación 1, en el que varias estructuras de ADN en forma de T están unidas respectivamente en un extremo mediante un adaptador por medio de uniones Holliday.
4. Constructo de expresión de ADN según la reivindicación 2, en el que varias estructuras de ADN lineales están unidas respectivamente en un extremo mediante un adaptador por medio de uniones Holliday.
5. Constructo de expresión de ADN según al menos una de las reivindicaciones anteriores, en el que los extremos cohesivos de componentes individuales están seleccionados de manera específica y distinta para la unión selectiva con otros componentes.
6. Constructo de expresión de ADN para la inhibición selectiva de la expresión de uno o varios genes según al menos una de las reivindicaciones 1 a 4, en el que las secuencias que van a transcribirse codifican para genes idénticos o distintos.
7. Constructo de expresión de ADN para la inhibición selectiva de la expresión de uno o varios genes según al menos una de las reivindicaciones 1 a 4, en el que la protección frente a degradación por exonucleasas se consigue mediante una región de cadena sencilla corta de tres a ocho desoxinucleótidos, que enlaza covalentemente entre sí las dos cadenas de la cadena doble lineal.
8. Constructo de expresión de ADN para la inhibición selectiva de la expresión de uno o varios genes según al menos una de las reivindicaciones 1 a 4, en el que éste está enlazado covalentemente en la región de cadena sencilla corta con uno o varios péptidos, proteínas, hidratos de carbono, anticuerpos o esteroides.
9. Procedimiento de preparación para vectores que tras su transfección en células eucariotas inhiben de manera selectiva la formación de proteínas definidas mediante interferencia de ARN, caracterizado por las siguientes etapas de procedimiento
a) mezclar una cadena doble de ADN que contiene una copia singular d.
19. 23 bases de longitud de una secuencia génica o su secuencia complementaria inversa así como una señal de terminación para ARNpolimerasas y está terminada mediante un bucle de secuencia de 8-12 bases de longitud en un extremo, que se selecciona de modo que bases opuestas no sean en ningún caso complementarias una con respecto a otra y que las cadenas sencillas de ADN flanqueantes estén unidas entre sí para dar una cadena doble de ADN que en el otro extremo presenta una cadena sencilla de ADN corta que sobresale, con b) un oligodesoxinucleótido en forma de horquilla que en su extremo presenta extremos cortos de ADN monocatenario que sobresalen y
c) una cadena doble de ADN de 10 a 1000 bases (= n) de longitud de una secuencia no codificante con extremos cortos de ADN monocatenario que sobresalen, pudiéndose aparear el extremo 3’ monocatenario con el extremo 5’ monocatenario del promotor y siendo el extremo 5’ monocatenario complementario al extremo 3’ monocatenario del oligodesoxinucleótido en forma de horquilla, y d) un promotor con extremos cortos de ADN monocatenario que sobresalen, pudiéndose aparear el extremo 5’ monocatenario del promotor con el oligodesoxinucleótido en forma de horquilla o la cadena doble de 1, 0 a 1000 bases de longitud y siendo el extremo 3’ monocatenario del promotor complementario al extremo 5’ monocatenario de la cadena doble de ADN descrita en a) y e) ligar posteriormente los fragmentos de ADN, f) así como purificar finalmente los vectores preparados.
10. Procedimiento de preparación para vectores que tras su transfección en células eucariotas inhiben la formación de proteínas definidas de manera selectiva mediante interferencia de ARN, caracterizado por las siguientes etapas de procedimiento
a) mezclar una cadena doble de ADN en forma de T que contiene una copia singular d.
19. 23 bases de longitud de una secuencia génica en dirección 5’ - 3’ así como una señal de terminación para ARN-polimerasas y porque está terminada mediante un bucle de secuencia d.
8. 12 bases de longitud en un extremo, que se selecciona de modo que bases opuestas no sean en ningún caso complementarias una con respecto a otra y que las regiones de cadena doble flanqueantes estén unidas entre sí así mediante dos cadenas sencillas de ADN y en el otro extremo presenta dos cadenas sencillas de ADN que sobresalen, y un oligodesoxinucleótido cuya secuencia es complementaria a las dos cadenas sencillas de ADN cortas que sobresalen y forma dos extremos cortos de ADN monocatenario que sobresalen, con b) un oligodesoxinucleótido en forma de horquilla que en el extremo presenta extremos cortos de ADN monocatenario que sobresalen y c) otro oligodesoxinucleótido en forma de horquilla que en el extremo presenta extremos cortos de ADN monocatenario que sobresalen, de manera complementaria a la cadena doble de ADN en forma de T de a) y d) un promotor con extremos cortos de ADN monocatenario que sobresalen, pudiéndose aparear el extremo 5’ monocatenario del promotor con el oligodesoxinucleótido en forma de horquilla de b) y siendo el extremo 3’ monocatenario del promotor complementario al extremo 5’ monocatenario de la cadena doble de ADN en forma de T y e) ligar posteriormente los fragmentos de ADN, así como f) purificar finalmente los vectores preparados.
11. Procedimiento de preparación según la reivindicación 10, en el que tras la etapa de procedimiento c) se añade una cadena doble de ADN de 10 a 1000 bases (= n) de longitud de una secuencia no codificante con extremos cortos de ADN monocatenario que sobresalen, pudiéndose aparear el extremo 3’ monocatenario con el extremo 5’ monocatenario del promotor y siendo el extremo 5’ monocatenario complementario al extremo 3’ monocatenario del oligodesoxinucleótido en forma de horquilla de la etapa de procedimiento b) y/o la adición de una cadena doble de ADN de 10 a 1000 bases (= n) de longitud de una secuencia no codificante con extremos cortos de ADN monocatenario que sobresalen, pudiéndose aparear el extremo 5’ monocatenario con el extremo 3’ monocatenario de la cadena doble de ADN en forma de T y siendo el extremo 3’ monocatenario complementario al extremo 5’ monocatenario del oligodesoxinucleótido en forma de horquilla de la etapa de procedimiento c) .
12. Procedimiento de preparación según al menos una de las reivindicaciones 9 a 11, en el que al inicio del procedimiento se hibridan oligodesoxinucleótidos monocatenarios parcialmente complementarios uno con respecto a otro para dar una molécula de unión (adaptador) , en el que la hibridación se realiza de manera incompleta de tal modo que dos o más regiones bicatenarias con extremos cohesivos con secuencia de bases igual o distinta se unen entre sí mediante uniones Holliday y realización posterior de las etapas de procedimiento indicadas.
13. Procedimiento según al menos una de las reivindicaciones 9 a 12, en el que el promotor es parte de un plásmido que puede amplificarse por vía bacteriana, que antes del mezclado de los componentes respectivos se corta con una endonucleasa de restricción que reconoce un sitio de corte que flanquea al promotor en el plásmido y que no está presente en el constructo de expresión que va a prepararse.
14. Procedimiento según la reivindicación 13, en el que la respectiva etapa de ligación se realiza en presencia de la endonucleasa de restricción con la que se escindió el promotor del plásmido.
15. Procedimiento según al menos una de las reivindicaciones 13 ó 14, en el que antes de la etapa de purificación final se realiza una digestión de la mezcla de reacción por medio de una exonucleasa específica exclusivamente para extremos de ADN 3’ o 5’.
16. Procedimiento según al menos una de las reivindicaciones 13 a 15, en el que en el caso de la endonucleasa de restricción se trata de una enzima del grupo de las endonucleasas de restricción clase II, preferentemente una enzima del grupo BbsI, BbvI, BbvII, BpiI; Bp1I, BsaI, BsmAI, BsmBI, BsmFI, BspMI, Eam1104I, EarI, Echo31I, Esp3I, FokI, HgaI, SfaNI o sus isoesquizómeros.
17. Procedimiento según al menos una de las reivindicaciones 9 a 16, en el que durante el mezclado de los componentes se añade una cadena doble de ADN que resulta del apareamiento parcial de un oligodesoxinucleótido parcialmente autocomplementario o al menos dos oligodesoxinucleótidos.
18. Procedimiento según al menos una de las reivindicaciones 9 a 17, en el que los oligodesoxinucleótidos en forma
de horquilla presentan en su región bicatenaria la secuencia de reconocimiento para una endonucleasa de restricción.
19. Procedimiento según al menos una de las reivindicaciones 9 a 18, en el que la purificación se realiza mediante cromatografía y/o por medio de electroforesis en gel.
20. Kit para la preparación de un constructo de expresión de acuerdo con una de las reivindicaciones 1 a 4, que
contiene al menos oligodesoxinucleótidos adecuados para la formación de estructuras de horquilla de terminación, oligodesoxinucleótidos adecuados para la formación de un adaptador, estructuras de ADN en forma de T en las que pueden insertarse las secuencias que van a transcribirse así como enzimas y medios para la realización de etapas de ligación, escisión, degradación de ADN y purificación.
diana adaptador
Patentes similares o relacionadas:
Proteínas del factor VIII que tienen secuencias ancestrales, vectores de expresión, y usos relacionados con ellos, del 15 de Julio de 2020, de EMORY UNIVERSITY: Una proteína FVIII recombinante o quimérica que comprende una o más mutaciones ancestrales y una eliminación del dominio B, y en donde la secuencia […]
Método para romper un ácido nucleico y añadir un adaptador por medio de transposasa y reactivo, del 1 de Julio de 2020, de MGI Tech Co., Ltd: Un metodo para romper un acido nucleico y anadir un adaptador por medio de una transposasa, que comprende las siguientes etapas: interrumpir […]
Vacuna subunitaria contra Mycoplasma spp., del 1 de Julio de 2020, de Agricultural Technology Research Institute: Una composición para prevenir una infección por Mycoplasma spp., que comprende: un principio activo, que comprende una proteína de PdhA; y un adyuvante […]
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Oligonucleótidos para tratar una enfermedad ocular, del 13 de Mayo de 2020, de ProQR Therapeutics II B.V: Un oligonucleótido antisentido (OAS) que es capaz de inducir omisión del pseudoexón 40 (PE40) de pre-ARNm de USH2A humano, donde dicho OAS comprende una secuencia […]
Composiciones promotoras, del 6 de Mayo de 2020, de UNIVERSITY OF IOWA RESEARCH FOUNDATION: Una secuencia promotora aislada que comprende un ácido nucleico de entre 500 y 1700 nucleótidos de longitud que tiene por lo menos un 98% de identidad con la SEQ […]
Transfección mediada por células apoptóticas de células de mamífero con ARN de interferencia, del 22 de Abril de 2020, de LOMA LINDA UNIVERSITY: Una célula de mamífero apoptótica o preapoptótica para su uso en la prevención del rechazo de trasplantes, en la que la célula de mamífero […]
Ácidos ribonucleicos con nucleótidos modificados con 4-tio y procedimientos relacionados, del 15 de Abril de 2020, de Translate Bio, Inc: Una molécula de ARNm que tiene una región codificante y opcionalmente, una o más regiones no codificantes, en donde al menos el 25% de los residuos de nucleótidos del […]