Cebadores, sondas y kits para la detección de especies bacterianas que pertenecen al género Rickettsia.
Un par de cebadores que tienen un cebador que comprende SEQ ID NO:
36 y un cebador que comprende SEQ ID NO: 37, y capaces de amplificar SEQ ID NO: 55, 56, 82-93 de especies bacterianas que provocan zoonosis, que pertenecen al género Rickettsia.
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E10173990.
Solicitante: INSTITUTO DE SALUD CARLOS III..
Nacionalidad solicitante: España.
Inventor/es: ANDA FERNANDEZ,PEDRO, ESCUDERO NIETO,RAQUEL, JADO GARCIA,ISABEL, RODRIGUEZ MORENO,ISABEL, JIMENEZ ALONSO,MARIA ISABEL.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2522523_T3.pdf
Fragmento de la descripción:
Cebadores, sondas y kits para la detección de especies bacterianas que pertenecen al género Rickettsia
La presente invención se refiere a la detección e identificación de especies bacterianas que provocan zoonosis, basada en el análisis de ADN. Más específicamente, la invención proporciona cebadores y kits para la detección de bacterias pertenecientes al género Rickettsia basados en la amplificación de ADN.
Antecedentes
Hasta la fecha hay descritas unas 200 enfermedades zoonóticas (bartonelosis, leptospirosis, borreliosis de Lyme...) que afectan a seres humanos. En los países en vías de desarrollo, son una importante causa de mortandad y suponen cuantiosas pérdidas económicas. La convivencia con animales, la ausencia de infraestructuras sanitarias y el bajo nivel cultural continúan siendo los principales aliados de estas enfermedades.
Determinados tipos de zoonosis se están difundiendo ahora en países desarrollados como consecuencia del aumento de la población en zonas urbanas y periurbanas, así como del aumento del tráfico de animales a nivel internacional, que conlleva el riesgo de introducir enfermedades exóticas en nuestro entorno.
Estas circunstancias, unidas al hallazgo frecuente de artrópodos infectados por más de uno de los patógenos incluidos en la presente invención, aumentan la posibilidad de que se transmita más de una de las especies bacterianas incluidas en la presente invención en una única picadura.
Como resultado, cada vez son más comunes las hospitalizaciones debidas a cuadros clínicos producidos por el contacto de seres humanos con animales o con diferentes clases de artrópodos, tales como mosquitos, garrapatas, pulgas, piojos, ácaros, etc., que actúan como vectores o como reservónos de patógenos. Dichos cuadros clínicos, debido a su gran similitud, no permiten una identificación rápida y fiable del agente patógeno, por lo que no es posible un tratamiento específico y rápido, y en ocasiones se administra demasiado tarde. Esto justifica, sin dejar lugar a dudas, la necesidad de un método integrado de detección.
Los métodos diagnósticos disponibles actualmente se limitan a la detección de anticuerpos que, por lo general, es retrospectiva y de escasa ayuda en el tratamiento de los pacientes en estados de fase aguda. El cultivo no se considera un método diagnóstico, tanto por su dificultad tecnológica, que lo excluye de las prácticas habituales en un laboratorio de microbiología de hospital, como por la necesidad de instalaciones P3.
El diagnóstico molecular por amplificación de genoma mediante PCR supone una opción diagnóstica de gran valor. Sin embargo, no siempre se dispone de suficiente cantidad de muestras clínicas para ensayos de patógenos o de la metodología necesaria para realizar diferentes ensayos.
VITORINO LILIANA ETAL "rRNA intergenic spacer regions for phylogenetic analysis of Rickettsia speciesTANNALS OF THE NEW YORK ACADEMY OF SCIENCES JUN 2003 LNKD- PUBMED:12860714, vol. 990, Junio 2003 (2003- 06), páginas 726-733, ISSN: 0077-8923 desvela un par de cebadores para amplificar el espaciador intergénico rrl-rrf en varias especies de Rickettsia. Los amplicones producidos comprender las secuencias espaciadoras 23S - 5S de las especies R.bellii, R.slovaca, R.conorii, R.aeschlimannii, R.ricketsii, R.helvetica, R.typhiy R.prowazekii.
Recientemente se ha publicado un trabajo (Blaskovic D. ef al. 2005. Oligo-based detection of tick-borne bacteria. FEMS Microbiology Letters 243:273-8), que describe un método para la detección de 5 de los 6 patógenos propuestos por la presente invención. Dicho método está basado en el análisis de ADN ribosómico y emplea cebadores universales, que amplifican material genético tanto de bacterias diana como de otras que no los son por lo que su sensibilidad es bastante reducida.
Otros métodos, tales como el descrito por las patentes americanas US 6.300.072 y 6.518.020, son capaces de detectar e identificar bacterias del género Bartonella, mediante el empleo de la misma región de ADN (espacio intergénico 16S-23S). Sin embargo, el número de especies dentro de este género ha aumentado sensiblemente desde la presentación de dichas patentes y su aproximación, que consiste en diferenciar entre especies según el tamaño del amplicón obtenido en una PCR, no es útil para determinadas especies conocidas dentro del género que son de un tamaño similar al fragmento amplificado.
Aunque el método aportado por la presente invención propone también el empleo de la región intergénica 16S-23S para la detección de especies pertenecientes al género Bartonella, se han introducido mejoras respecto de los procedimientos descritos previamente, ya que es capaz de detectar un grupo mucho más amplio de especies dentro del mismo y otros géneros, utilizando sondas y cebadores completamente novedosos y con niveles de sensibilidad máximos.
Para la detección de Coxiella burnetii, se emplean los mismos cebadores y la misma región de ADN (secuencia de inserción IS1111) que los métodos anteriormente descritos. Se ha mejorado dicha detección combinándola con otra
serie de ensayos completamente novedosos para la identificación de otras especies bacterianas, que pueden ser transmitidas por los mismos vectores, y además aporta una nueva sonda de hibridación para la detección de Coxiella burnetii.
Descripción de la invención
Definiciones: PCR Múltiple o PCR Multiplex: la PCR (Reacción en Cadena de la Polimerasa) es un sistema mediante el que se amplifica o aumenta el número de copias de una secuencia de nucleótidos concreta de un organismo, mediante el uso de dos cebadores. La PCR múltiple o PCR multiplex es una variante de la PCR, que permite la amplificación simultanea de más de una secuencia diana empleando más de un par de cebadores.
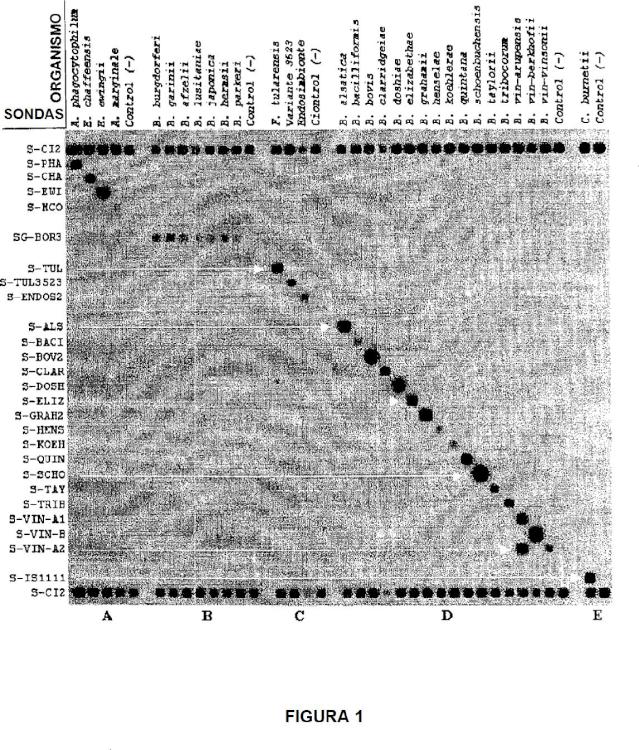
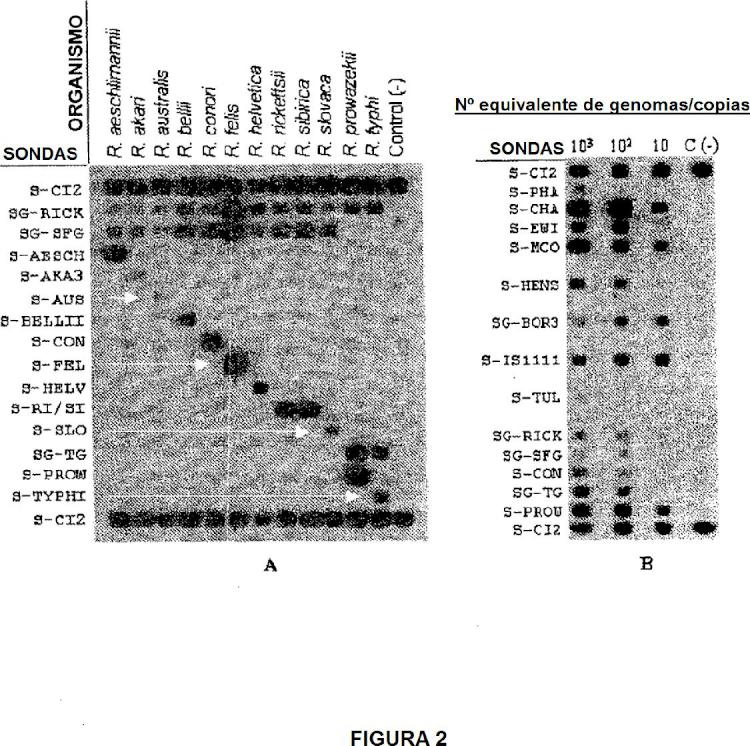
La presente invención resuelve el problema de lo tedioso y complicado que resulta detectar un elevado número de bacterias, que causan zoonosis que pueden ser clínica y/o epidemiológicamente indistinguibles, mediante el desarrollo de un método y un Kit para la detección de especies bacterianas causantes de zoonosis pertenecientes al género Rickettsia.
La solución encontrada por la presente invención consiste en analizar una región del ADN bacteriano para determinar que especies se encuentran presentes. En concreto se analiza el ARNr de espacio intergénico 23S-5S para detectar la presencia de Rickettsia.
De acuerdo con lo anterior, se describe un método para la detección de bacterias a partir de una muestra, que comprende las siguientes etapas:
i) poner en contacto la muestra que se analiza con una mezcla de reacción, que contiene cebadores específicos para la realización de PCR Multiplex.
ii) amplificar mediante reacción en cadena de la polimerasa.
iii) Identificar la formación de los productos de la etapa anterior, siendo dicha información indicativa de la presencia o ausencia de bacterias causantes de zoonosis.
En relación con esto, se describe un método para detectar:
El género Rickettsia, y el grupo causante de la fiebre maculosa y el grupo causante del tifus, las especies Rickettsia akari, R. bellii, R. slovaca, R. conorii, R. aeschlimannii, R. ricketsii, R. sibirica, R. helvética, R. felis, R. australis, R. prowazekii, y R. typhy (R. mooserii),
siendo todas ellas capaces de provocar zoonosis, infectar conjuntamente a un individuo y difíciles de identificar por la simple observación de los cuadros clínicos, basándose en la amplificación y análisis de los genes o regiones génicas concretas presentadas en las tabla 6.
De acuerdo con una realización específica, se amplifican fragmentos de ADN incluidos o comprendidos dentro de las secuencias cuyos números de referencia se muestran en la tabla 6.
De acuerdo con una realización más específica, las regiones amplificadas tienen un tamaño de entre 99 y 686 nucleótidos y contienen regiones variables empleadas para la identificación. De acuerdo con una realización aún más específica, las regiones variables contienen o están incluidas dentro de las SEQ ID NO: 55, SEQ ID N°: 56 y SEQ ID N°: 82 a SEQ ID NO: 93 o secuencias complementarias, cuyas posiciones se muestran en la tabla 6.
De acuerdo con otra realización, los productos de amplificación, que permiten identificar los diferentes especies y grupos bacterianos se detectan mediante el empleo de sondas. De acuerdo con una realización más preferida, estas sondas tienen una longitud de entre 15 y 25 nucleótidos. Y de acuerdo con una realización aún más preferida, las sondas tienen secuencias SEQ ID NO:38-51; o secuencias complementarias (Tabla 6).
Los cebadores pueden diseñarse mediante alineamiento... [Seguir leyendo]
Reivindicaciones:
1. Un par de cebadores que tienen un cebador que comprende SEQ ID NO: 36 y un cebador que comprende SEQ ID NO: 37, y capaces de amplificar SEQ ID NO: 55, 56, 82-93 de especies bacterianas que provocan zoonosis, que
pertenecen al género Rickettsia.
2. El par de cebadores de acuerdo con la reivindicación 1 que tienen la secuencia SEQ ID NO: 36 y SEQ ID NO: 37.
3. Sonda que consiste en la secuencia seleccionada de SEQ ID NO: 38-51, y capaz de hibridar con SEQ ID NO: 55,
56, 82-93 de especies bacterianas que provocan zoonosis, que pertenecen al género Rickettsia.
4. Un kit que comprende el par de cebadores de acuerdo con cualquiera de las reivindicaciones 1 o 2.
5. El kit de acuerdo con la reivindicación 4, que comprende además los cebadores SEQ ID NO: 52 y SEQ ID 53.
6. Un kit que comprende la sonda de acuerdo con la reivindicación 3.
7. El kit de acuerdo con la reivindicación 6, que comprende además la sonda SEQ ID NO: 54.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]