Determinación de resistencia con espectrometría de masas mediante la medición de la proliferación microbiana.
Método para la determinación mediante espectrometría de masas de la resistencia microbiana en el cual los microbios son cultivados en un medio con un antibiótico y se obtiene un espectro de masas EMcum de los microbios tras su cultivo,
caracterizado por que la proliferación microbiana que, en su caso, tiene lugar durante el cultivo se determina con ayuda de una sustancia de referencia añadida de forma dosificada y medida en el espectro de masas EMcum, de forma que una proliferación microbiana indica resistencia frente al antibiótico.
Tipo: Patente Europea. Resumen de patente/invención. Número de Solicitud: E13002699.
Solicitante: Bruker Daltonik GmbH.
Nacionalidad solicitante: Alemania.
Dirección: Fahrenheitstrasse 4 28359 Bremen ALEMANIA.
Inventor/es: LANGE,CHRISTOPH, SPARBIER,KATRIN.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/04 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › Determinación de la presencia o del tipo de microorganismo; Empleo de medios selectivos para la investigación o análisis de antibióticos o bactericidas; Composiciones para este fin que contienen un indicador químico.
- C12Q1/18 C12Q 1/00 […] › Investigación o análisis de la actividad antimicrobiana de un material.
- G01N33/68 FISICA. › G01 METROLOGIA; ENSAYOS. › G01N INVESTIGACION O ANALISIS DE MATERIALES POR DETERMINACION DE SUS PROPIEDADES QUIMICAS O FISICAS (procedimientos de medida, de investigación o de análisis diferentes de los ensayos inmunológicos, en los que intervienen enzimas o microorganismos C12M, C12Q). › G01N 33/00 Investigación o análisis de materiales por métodos específicos no cubiertos por los grupos G01N 1/00 - G01N 31/00. › en los que intervienen proteínas, péptidos o aminoácidos.
PDF original: ES-2545873_T3.pdf
Fragmento de la descripción:
Determinación de resistencia con espectrometría de masas mediante la medición de la proliferación microbiana La invención se refiere a un proceso de espectrometría de masas para la determinación de las resistencias microbianas frente a antibióticos.
Definiciones En este documento se utiliza, en lugar de la «unidad de masa atómica unificada» (u) establecida legalmente, la unidad «dalton» (Da) , que en la última (octava) edición de 2006 del documento «The International System of Units (SI) » del Bureau International des Poids et Mesures se equiparó a la unidad de masa atómica; en particular, tal y como allí se apuntó, para poder utilizar las unidades kilodalton, milidalton y similares.
Por razones de simplificación en este documento solo se empleará el término «proteína», aunque en el intervalo de masas de 3.000 a 15.000 dalton con frecuencia sería preferible referirse a las «proteínas» como «péptidos». La transición de los péptidos más ligeros a las proteínas más pesadas es difusa y no se ha definido de forma inequívoca.
Como abreviatura de «microorganismos» se utiliza aquí con frecuencia el término «microbios». Bajo el término singular «microbio» se entiende, tal y como es habitual en el uso general del lenguaje, tanto una célula microbiana por separado como la especie microbiana. Bajo el término plural «microbios» se entienden las células microbianas objeto de análisis.
Estado de la técnica
Muchas especies de microorganismos, en particular las bacterias y hongos unicelulares como las levaduras, se pueden identificar actualmente mediante espectrometría de masas de forma rápida y con tasas de error muy bajas. Según el proceso habitual, la identificación se realiza mediante el cálculo de los índices de similitud entre un espectro de masas de los microbios sometidos a lisis celular, en particular de sus proteínas solubles, y similares espectros de masas de referencia de microorganismos conocidos. Si los índices de similitud superan determinados valores límite, se pueden identificar familia, género, especie o incluso cepa. Este método de identificación de microorganismos, muy rápido y con costes reducidos, ha tenido un extraordinario éxito tanto en estudios de gran envergadura como en la aplicación diaria en numerosos laboratorios de microbiología. Según el aparato se pueden determinar de 48 a 384 muestras microbianas de forma simultánea; la identificación tiene lugar tan solo unos minutos tras el final del cultivo de una colonia. El método tiene tasas de error muy reducidas, más reducidas que las tasas de error de los métodos de identificación microbiológica convencionales, incluso más reducida que los análisis de ADN. Existen ya en el mercado espectrómetros de masas, programas de evaluación y bibliotecas con espectros de referencia que están admitidos como productos para el diagnóstico in vitro (DIV) conforme a la ley alemana de productos médicos y otros reglamentos y directrices nacionales o internacionales.
El término «antibiótico» se refiere, tal y como es habitual en el uso general del lenguaje, tanto a un principio activo farmacológico para el tratamiento de enfermedades infecciosas microbianas como a sustancias desinfectantes. Los éxitos de la penicilina condujeron a la búsqueda y descubrimiento de muchos otros antibióticos. Existen antibióticos de amplio espectro, que son eficaces frente a muchas familias microbianas, y antibióticos de espectro reducido, que actúan de forma selectiva contra especies microbianas concretas.
Desde que la penicilina se aplicó como primer principio activo farmacológico las cepas microbianas han desarrollado con frecuencia creciente diferentes resistencias frente a diferentes tipos de antibióticos o las han adquirido de otros microbios, obteniendo así propiedades que les permiten atenuar o neutralizar por completo el efecto de los principios activos antibióticos. Desafortunadamente, las resistencias son ya frecuentes; actualmente la mayoría de los microbios que aparecen en los hospitales son resistentes. En algunos casos puede preverse la resistencia de un microbio transmitido en el hospital frente a los antibióticos habituales en el hospital; pero esto no rige en el caso de las infecciones adquiridas fuera del hospital.
El éxito del tratamiento de las infecciones microbianas, que en situaciones agudas como en una sepsis o una infección secundaria a una enfermedad (o infección) primaria ya existente con frecuencia suponen una amenaza para la vida, depende con frecuencia de la eficacia de la primera administración de un antibiótico. Una administración dirigida no solo requiere una identificación del microorganismo patógeno lo más rápida y correcta posible, sino también una determinación lo más rápida posible de su resistencia frente a los diferentes antibióticos. La determinación clásica de la resistencia consiste en un cultivo con un antibiótico, pero por desgracia lleva mucho tiempo: dura de 24 a 48 horas.
Aparte del cultivo con antibióticos existen también métodos genéticos para determinar las resistencias. En ellos la determinación de una resistencia se realiza mediante la comprobación de los genes de resistencia conocidos en el genoma del microorganismo patógeno correspondiente. Una ventaja del método genético es que los genes de
resistencia pueden reproducirse mediante técnicas como la reacción en cadena de la polimerasa (RCP) , de forma que la duración del análisis ya no está determinada por la tasa de proliferación de las bacterias. Las desventajas consisten en que estos métodos tienen costes más elevados que los métodos de rutina y no permiten la realización de pruebas funcionales. Así, puede haber un gen de resistencia que no esté expresado, de forma que la cepa bacteriana objeto de análisis no sea resistente, pero el proceso la identifica como resistente.
En la patente DE 10 2006 021 493 B4 (V. M. Govorun y J. Franzen, 2006, correspondiente a GB 2438066 B, US 8, 293, 496 B2; de aquí en adelante denominada «Govorun») se presentan métodos de espectrometría de masas para la determinación de la resistencia microbiana.
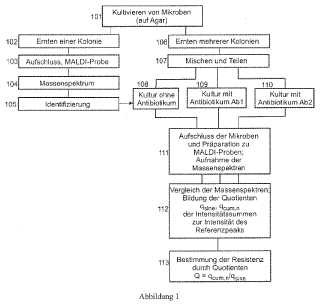
En una versión se miden mediante espectrometría de masas, por ejemplo, los perfiles proteínicos de los microbios tras el cultivo en medios con y sin adición de antibióticos y se comparan. Para ello los microbios se cultivan, por ejemplo, en tubos de centrifugadora con y sin antibióticos. Tras el cultivo los microbios se centrifugan, se lavan y a continuación se someten a lisis con ácidos y acetonitrilo en los tubos de centrifugadora para que liberen sus proteínas solubles. Una mínima cantidad (aprox. 1 µl) de este líquido de lisis se prepara en el soporte de muestras, se seca y a continuación se cubre con una pequeña cantidad de solución matriz (también aprox. 1 µl) . Las proteínas liberadas se incorporan a los cristales que se forman al secar la matriz. Las muestras con cristales de matriz y proteínas incorporadas se someten a pulsos de luz de láser en el espectrómetro de masas, de manera que los iones de las moléculas de las proteínas forman un plasma de evaporación. La medición de su tiempo de vuelo en un espectrómetro de masas con analizador de tiempo de vuelo da como resultado el espectro de masas del microbio, que consiste principalmente en picos de proteínas.
Pero a partir de la similitud de ambos espectros de masas no se puede concluir con certeza la resistencia del microbio. Si se inhibe la proliferación de los microbios susceptibles o se los elimina sin descomponerlos, por ejemplo bacterias del género Klebsiella de la familia de las enterobacterias, el resultado es un espectro de masas casi idéntico al de los microbios en medios sin antibiótico. El motivo es que la formación de los espectros de masas, que se obtienen con una ionización por desorción láser asistida por matriz (MALDI) , solo depende mínimamente de la cantidad de microbios de la muestra. Las preparaciones de muestras con diez mil microbios proporcionan prácticamente los mismos espectros de masas que las preparaciones con diez millones de microbios, lo cual es ideal para una identificación, pero no para la detección de la resistencia. Si únicamente se detiene la proliferación de los microbios se obtienen los mismos espectros de masas, ya que solo existe una diferencia cuantitativa que no es visible en los espectros de masas.
Tal y como se detalla en la patente Govorun, la resistencia también se puede detectar mediante la adición a los microbios objeto de análisis de un segundo tipo de microbios que proporcione un espectro de masas muy diferente, que sea resistente y que en todo caso continúe proliferando en presencia del antibiótico. El espectro de masas con las proteínas superpuestas... [Seguir leyendo]
Reivindicaciones:
1. Método para la determinación mediante espectrometría de masas de la resistencia microbiana en el cual los microbios son cultivados en un medio con un antibiótico y se obtiene un espectro de masas EMcum de los microbios tras su cultivo, caracterizado por que la proliferación microbiana que, en su caso, tiene lugar durante el cultivo se determina con ayuda de una sustancia de referencia añadida de forma dosificada y medida en el espectro de masas EMcum, de forma que una proliferación microbiana indica resistencia frente al antibiótico.
2. Método según la reivindicación 1 caracterizado por que la cantidad de microbios tras el cultivo se determina a partir del espectro de masas EMcum y los microbios se identifican como resistentes cuando la cantidad de microbios es superior a un valor límite fijado previamente.
3. Método según una de las reivindicaciones 1 o 2 caracterizado por el cultivo adicional de los microbios en el medio sin el antibiótico y la obtención de un espectro de masas EMsine de los microbios tras su cultivo sin antibiótico, en el que también se mide la sustancia de referencia añadida de forma dosificada y se identifican los microbios como resistentes cuando la diferencia relativa o absoluta entre las cantidades de microbios resultantes de los espectros de masas EMcum y EMsine es menor que un valor límite fijado.
4. Método según una de las reivindicaciones 1 a 3 caracterizado por que antes del cultivo se obtiene adicionalmente un espectro de masas EM0 de los microbios en el que también se mide la sustancia de referencia añadida de forma dosificada y se identifican los microbios como resistentes cuando la cantidad de microbios obtenida a partir del espectro de masas EMcum es considerablemente mayor que la obtenida a partir del espectro de masas EM0.
5. Método según una de las reivindicaciones 2 a 4 caracterizado por que la cantidad de microbios se averigua como un cociente a partir de un espectro de masas, donde -el cociente se obtiene a partir de la intensidad de una señal microbiana y la intensidad de una señal de referencia o la suma de las intensidades de varias señales de referencia, -el cociente se obtiene a partir de la suma de las intensidades de varias señales microbianas y la intensidad de una señal de referencia o la suma de las intensidades de varias señales de referencia, o bien -el cociente se obtiene a partir de la suma de las intensidades de todas las señales de un intervalo o de todo el espectro de masas y la intensidad de una señal de referencia o la suma de las intensidades de varias señales de referencia.
6. Método según una de las reivindicaciones 1 a 5 caracterizado por que la sustancia de referencia se añade de forma dosificada tras un cultivo de los microbios.
7. Método según una de las reivindicaciones 1 a 5 caracterizado por que los microbios se preparan para obtener
una muestra para espectrometría de masas y la substancia de referencia se añade de forma dosificada durante la preparación.
8. Método según la reivindicación 7 caracterizado por que los espectros de masas se obtienen mediante ionización por desorción láser asistida por matriz (MALDI) y la sustancia de referencia se añade durante la preparación de las 45 muestras para MALDI en un soporte de muestras.
9. Método según una de las reivindicaciones 1 a 8 caracterizado por que los microbios son cultivados simultáneamente en varios cultivos, cada uno con un antibiótico diferente, y para cada cultivo se obtiene un espectro de masas de los microbios y de la sustancia de referencia.
10. Método según una de las reivindicaciones 1 a 9 caracterizado por que los microbios son cultivados simultáneamente en varios cultivos cada uno con una concentración diferente del antibiótico y para cada cultivo se obtiene un espectro de masas de los microbios y de la sustancia de referencia.
11. Método según una de las reivindicaciones 1 a 10 caracterizado por que la afinidad protónica de la sustancia de referencia es superior a la afinidad protónica de la mayoría de las proteínas de los microbios.
12. Método según la reivindicación 11 caracterizado por que la sustancia de referencia es una ribonucleasa o una lisozima. 60
13. Método según una de las reivindicaciones 1 a 12 caracterizado por que antes o durante la preparación de una muestra para espectrometría de masas se concentran las proteínas de los microbios.
14. Método según una de las reivindicaciones 1 a 13 caracterizado por que se añade de forma dosificada una 65 mezcla de sustancias de referencia y se mide en los espectros de masas de los microbios.
15. Método según una de las reivindicaciones 1 a 14 caracterizado por la identificación taxonómica de los microbios antes de la determinación de la resistencia y la selección de la sustancia de referencia, los valores límite para el establecimiento de la resistencia, el medio y/o las condiciones de cultivo, en particular el tiempo de cultivo, a partir de la clasificación taxonómica.
Patentes similares o relacionadas:
Inmunomoduladores, del 29 de Julio de 2020, de BRISTOL-MYERS SQUIBB COMPANY: Un compuesto de la fórmula (I) **(Ver fórmula)** o una sal farmacéuticamente aceptable del mismo, en donde: A se selecciona de **(Ver fórmula)** en donde: […]
Métodos y composiciones para el diagnóstico y pronóstico de lesión renal e insuficiencia renal, del 29 de Julio de 2020, de Astute Medical, Inc: Un método para evaluar el estado renal en un sujeto, que comprende: realizar una pluralidad de ensayos configurados para detectar una […]
Neuregulina para tratar la insuficiencia cardíaca, del 29 de Julio de 2020, de Zensun (Shanghai) Science & Technology, Co., Ltd: Neuregulina para usar en un método para tratar la insuficiencia cardíaca crónica en un paciente, donde el paciente tiene un nivel plasmático de NT-proBNP […]
Método para llevar a cabo el seguimiento de la enfermedad de Gaucher, del 15 de Julio de 2020, de Centogene GmbH: Un método para determinar la evolución de la enfermedad de Gaucher en un sujeto, que comprende la etapa de determinar en varios puntos en el […]
Procedimiento para evaluación de la función hepática y el flujo sanguíneo portal, del 15 de Julio de 2020, de The Regents of the University of Colorado, a body corporate: Procedimiento in vitro para la estimación del flujo sanguíneo portal en un individuo a partir de una única muestra de sangre o suero, comprendiendo el procedimiento: […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Biomarcadores de pronóstico y predictivos y aplicaciones biológicas de los mismos, del 1 de Julio de 2020, de INSTITUT GUSTAVE ROUSSY: Un método para evaluar la sensibilidad o la resistencia de un tumor frente a un agente antitumoral, que comprende evaluar la cantidad de complejo eiF4E-eiF4G (complejo Cap-ON) […]
Métodos de monitorización terapéutica de profármacos de ácido fenilacético, del 24 de Junio de 2020, de Immedica Pharma AB: Glicerilo tri-[4-fenilbutirato] (HPN-100) para su uso en un método para tratar un trastorno del ciclo de la urea en un sujeto que tiene discapacidad […]