Procedimiento para la determinación de la fragmentación del ADN en microorganismos.
La presente invención se relaciona con un procedimiento para la determinación de la integridad del ADN en microorganismos,
y un kit para la evaluación de la integridad del ADN en los mismos. Debido a que la muerte celular se traduce en fragmentación del ADN, con el presente procedimiento se puede discriminar claramente, y de forma sencilla, rápida y precisa, los niveles de fragmentaciónde ADN en microorganismos.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/ES2007/000637.
Solicitante: UNIVERSIDAD AUTONOMA DE MADRID.
Nacionalidad solicitante: España.
Inventor/es: FERNANDEZ GARCIA,JOSE LUIS, GOYANES VILLAESCUSA,VICENTE, MURIEL RIOS,LOURDES, CARTELLE GESTAL,MONICA, GOSÁLVEZ BERENGUER,Jaime, BAU ARÉVALO,GERMÁN.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2509879_T3.pdf


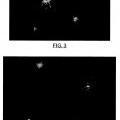

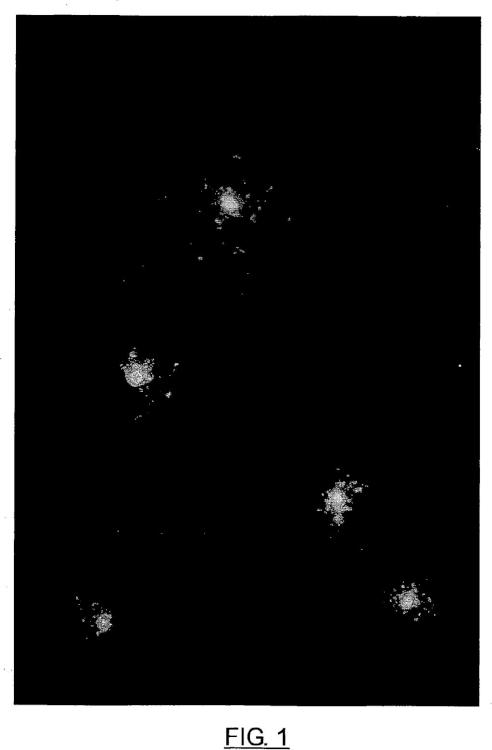
Fragmento de la descripción:
Procedimiento para la determinación de la fragmentación del ADN en microorganismos La presente invención se encuadra dentro del campo de la industria biotecnológica y principalmente en aquel relacionado con la microbiología, cuyo ámbito de aplicación se encuentra dentro del sector sanitario (humano, veterinario, medioambiental y básico) .
En concreto, se refiere a un procedimiento de determinación de la integridad del ADN en microorganismos, dado que la muerte celular se traduce en fragmentación del ADN y de un kit para la evaluación de la integridad del ADN en microorganismos.
Estado de la técnica Los microbios pueden morir por diversas causas. En el caso de las bacterias, que son organismos de especial interés sanitario, el mecanismo final de muerte por acción de agentes antibióticos es prácticamente desconocido, seguramente por lo obvio del problema. Los antibióticos afectan procesos celulares importantes, que tarde o temprano llevarán a la muerte de la célula. A pesar del conocimiento del mecanismo inicial de acción de un antibiótico concreto, a veces no es posible discernir claramente un efecto bacteriostático o bactericida. Este cuadro acerca de la muerte celular es especialmente complicado debido a la reciente descripción de la presencia de una pequeña proporción de células que persisten invulnerables a los antibióticos bactericidas, a pesar de ser mutantes y no crecer en presencia del antibiótico. Dichas células persistentes parecen explicar la alta resistencia de los biofilms y de los cultivos estacionarios a la muerte por agentes quimioterápicos.
Además, los estudios de perfiles de transcripción de la totalidad de los genes de Escherichia coli, han demostrado la existencia de un grupo de genes que se inducen y otros que se reprimen de forma común tras la acción de antibióticos, siendo el mecanismo de acción muy diferente. Esto se comprobó con la ampicilina, inhibidora de la síntesis de la pared celular y con la ofloxacina, una fluoroquinolona que bloquea la ADN girasa y la topoisomerasa IV, induciendo daño directo en el ADN (Kaldalu N, Mei R, Lewis K. Killing by ampicillin and ofloxacin induces overlapping changes in Escherichia coli transcription profile. Antimicrob Agents Chemother 2004; 48:890-896.)
Estos conocimientos sugieren que la muerte celular en bacterias, por ejemplo tras la acción de un antibiótico bactericida, podría ser un proceso programado, al igual que el fenómeno de apoptosis, presente en los organismos superiores. Un fenómeno similar ha sido descrito en levaduras unicelulares, como respuesta la acción de agentes fungicidas. La autolisis de las células bacterianas por autodigestión de la pared celular por autolisinas tras la exposición a antibióticos o condiciones ambientales adversas, puede ser una expresión de la muerte celular programada de organismos defectuosos (Lewis K. Programmed Death in Bacteria. Microbiol Mol Biol Rev 2000; 64: 503-514.)
La mayor parte de estudios de acción quimioterápica se vienen evaluando de modo rutinario, desde los inicios de la microbiología, valorando el crecimiento celular como la capacidad de producir colonias en un medio de cultivo semisólido o de producir turbidez en un medio líquido. Este sistema, además de ser relativamente largo, no evalúa el comportamiento de cada célula sino el conjunto en general y solo es aplicable a aquellos microorganismos con capacidad de ser cultivables in vitro. Para estudiar el estado vital de cada célula, es necesario el uso de técnicas de microscopía o citometría (Lecoeur H. Nuclear apoptosis detection by flow cytometr y : influence of endogenous nucleases. Exp Cell Res 2002; 277:1-14; Steensma DP, Timm M, Witzig TE. Flow cytometric methods for detection and quantification of apoptosis. Methods Mol Med 2003; 85:323-332) .
Una posible evaluación, aunque no habitual, es valorar la permeabilidad de la pared celular y de la membrana celular, usando colorantes vitales. La célula solo se tiñe con el colorante vital si posee una alteración de la barrera que la aísla del exterior, lo que suele estar ligado a la lisis por choque osmótico. Sea por daño directo, a través de sistemas enzimáticos, o a través de la pérdida de la integridad de la membrana, el ADN del cromosoma del microorganismo debe fragmentarse en el proceso de muerte celular. Sin embargo, la integridad del ADN cromosómico no se ha venido evaluando como parámetro de muerte microbiana, en estudios in situ célula a célula. Esto se debe a la ausencia de una técnica asequible, fiable y reproducible, para determinar la integridad de un ADN cromosómico de pequeño tamaño en relación con el de las células de organismos superiores.
Existen diferentes metodologías in situ bien establecidas para evaluar la integridad del ADN de las células de organismos superiores en relación con el daño inducido y con la muerte celular por apoptosis o necrosis. Entre tales metodologías se destacan el etiquetado de roturas del ADN in situ introduciendo nucleótidos marcados en las mismas utilizando enzimas como la transferasa terminal (TUNEL) o la ADN polimerasa (traslado de la mella in situ ISNT) (Didenko V, ed. In situ detection of DNA damage. Humana Press, Totowa, Nueva Jersey, 2002.)
Estas metodologías se basan en el empleo de enzimas sobre las células fijadas en portaobjetos, que actúan sobre extremos 3'-OH de las roturas, es decir sin modificaciones químicas. Por dicho motivo, su eficacia es irregular, resultando solo marcadas aquellas roturas que son accesibles a la enzima, lo que se traduce en una reproducibilidad relativamente baja de los resultados. Existe un único trabajo en el que se aplica de la técnica de TUNEL para la detección de roturas del ADN bacteriano, en Escherichia coli y la arquea Haloferax volcanii (Rohwer F and Azam F.
Detection of DNA Damage in Prokar y otes by Terminal Deoxyribonucleotide Transferase-Mediated dUTP Nick End Labeling Appl Environ Microbiol 2000; 66:1001-1006.)
Este estudio demostró la capacidad de detección de la fragmentación del ADN bacteriano tras infección por un fago. Sin embargo, las roturas originadas directamente por el peróxido de hidrógeno, en ciertas condiciones, no se detectaban. Esto puede deberse a la imposibilidad de la enzima de marcar roturas con extremo 3 '-OH modificados, lo que es una dificultad de esta técnica. Además, las células deben fijarse para efectuar estas técnicas, lo que afecta a la capacidad de marcaje. Además, los reactivos son caros, por lo que estas técnicas solo se aplican en estudios de investigación, no siendo posible utilizarlas para la valoración rutinaria del daño del ADN y su degradación. Estas técnicas son relativamente largas y complejas, por lo que no se han usado de modo habitual en microbiología y no se ha descrito ningún otro trabajo en relación a las mismas.
Otra técnica de microscopía para el estudio in situ célula a célula de la integridad del ADN es el ensayo de cometas o electroforesis de célula única (Olive PL, Durand RE. Heterogeneity in DNA damage using the comet assay. Cytometr y 2005; 66:1-8.)
Las células eucariotas se incluyen en un microgel de agarosa sobre un portaobjetos y se someten a soluciones lisantes para extraer las membranas y las proteínas. Se obtienen así nucleoides es decir núcleos desproteinizados, en los que los bucles de ADN se han relajado debido a la descompactación. Los nucleoides se someten a una electroforesis en una cubeta rellena de solución tampón, de tal modo que las fibras de ADN migran hacia el ánodo, constituyendo una imagen de cometa con una cabeza y una cola en la dirección de la migración electroforética. Estos cometas se tiñen con un colorante fluorescente para ser observados mediante microscopía de fluorescencia. Si el núcleo presenta fragmentación del ADN, gran cantidad de fragmentos del mismo habrán migrado, concentrándose en la cola del cometa. Se trata de un test bastante sensible pero relativamente caro y complicado para un laboratorio clínico convencional. De hecho, requiere cierto instrumental no común: fuente y cubeta de electroforesis y un sistema de captación de las imágenes y de análisis de las mismas. Por todo ello, solo se utiliza con fines de investigación. Existe un único trabajo publicado en el que la técnica de cometas se aplica a un pH neutro en la bacteria Escherichia coli (Singh NP, Stephens RE, Singh H, Lai H. Visual quantification of DNA doublestrand breaks in bacteria. Mutat Res 1999; 429:159-168) .
La técnica es larga y complicada, requiriendo múltiples incubaciones y la interpretación de las imágenes en relación con las roturas del ADN no está clara. Por lo tanto, tampoco se ha descrito ningún otro trabajo con esta técnica en bacterias.
De lo descrito anteriormente se... [Seguir leyendo]
Reivindicaciones:
1. Un procedimiento para evaluar la integridad del ADN de los microorganismos que comprende las siguientes etapas secuenciales:
a) inmovilización del microorganismo sobre un portaobjetos, sin fijación, mediante inclusión del mismo en 5 un medio inerte;
b) tratamiento con una solución de lisis no desnaturalizante del ADN, que tiene un pH comprendido entre 6, 5 y 10, 5 para extraer paredes, membranas y proteínas celulares, en el que el detergente comprendido en dicha solución de lisis es un detergente desnaturalizante de proteínas iónico;
c) estabilización del nucleoide de ADN del microorganismo sobre el portaobjetos secando la muestra por 10 medio de calor seco, incubando el portaobjetos con la muestra lisada en un horno de microondas y d) tinción con un colorante fluorescente altamente sensible y evaluación de la integridad del ADN.
2. Un procedimiento de acuerdo con la reivindicación 1, en el que el detergente iónico es un detergente seleccionado del grupo del dodecilsulfato de sodio (SDS) , sulfonato de alquilbenceno, laurilsarcosina (sarcosil) , sal hidratada del acido glicocólico y sus mezclas.
3. Un procedimiento de acuerdo con cualquiera de las reivindicaciones 1 y 2, en el que la solución de lisis comprende ditiotreitol (DTT) entre 0, 001 y 2M; 2-amino-2 (hidroximetil) -1, 3-propanodiol (Tris) entre 0, 001 y 2M; ácido etilendiamino tetraacético (EDTA) entre 0, 001 y 2M y dodecilsulfato de sodio (SDS) entre 0, 1 y 3%.
4. Un procedimiento de acuerdo con la reivindicación 3, en el que la solución de lisis comprende preferentemente dithiothreitol (DTT) 0, 1M; (hidroximetil) -1, 3-propanodiol (Tris) 0, 01M; ácido etilendiamino tetraacético (EDTA) 0, 05M 20 y dodecilsulfato de sodio (SDS) 2%.
5. Un procedimiento de acuerdo con la reivindicación 4, en el que la solución de lisis es ajustada a pH aproximadamente 10 con NaOH.
6. Un procedimiento de acuerdo con la reivindicación 1, en el que la muestra que contiene los microorganismos está incluida en un microgel inerte, preferentemente en un microgel de agarosa.
8. Un procedimiento de evaluación de la integridad del ADN de los microorganismos de acuerdo con la reivindicación 1, en el que la evaluación se realiza en una forma automatizada mediante la aplicación de software para analizar imágenes digitalizadas, obtenidas mediante cámaras acopladas a plataformas de microscopía. 10. El uso de acuerdo con la reivindicación 9, en el que la solución de lisis comprende ditiotreitol (DTT) entre 0, 001 y 2M; 2-amino-2 (hidroximetil) -l, 3-propanodiol (Tris) entre 0, 001 y 2M; ácido etilendiamino tetraacético (EDTA) entre 0, 001 y 2M y dodecilsulfato de sodio (SDS) entre 0, 1 y 3%. 11. El uso de acuerdo con cualquiera de las reivindicaciones 9 y 10, en el que la solución de lisis es ajustada a pH entre 6, 5 y 10, 5. 12. El uso de acuerdo con cualquiera de las reivindicaciones 9 a 11, en el que la solución de lisis comprende preferente ditiotreitol (DTT) 0, 1M; (hidroximetil) -1, 3-propanediol (Tris) 0, 01M; ácido etilendiamino tetraacético (EDTA) 0, 05M y dodecilsulfato de sodio (SDS) 2%. 13. El uso de acuerdo con cualquiera de las reivindicaciones 9 a 12, en el que la solución de lisis es ajustada a pH de aproximadamente 10 con NaOH. 7. Un procedimiento de evaluación de la integridad del ADN de los microorganismos de acuerdo con la reivindicación 1, en el que la evaluación se realiza mediante un análisis visual directo.
9. El uso de un kit para llevar a cabo el procedimiento de la reivindicación 1, comprendiendo dicho kit: a) portaobjetos pretratados; b) solución de agarosa; c) solución de lisis que comprende un agente desnaturalizante de proteínas iónico; y d) fluorocromo.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]