Detección no invasiva de anormalidades genéticas fetales.
Un método implementado por ordenador para determinar una anormalidad genética fetal la cual es una aneuploidía cromosómica,
método que comprende:
(a) obtener la información de secuencia de múltiples fragmentos de polinucleótidos a partir de una muestra, siendo dicha muestra una muestra de sangre periférica derivada de un sujeto femenino en embarazo y que contiene ADN tanto materno como fetal;
(b) asignar dichos fragmentos a cromosomas con base en dicha información de secuencia comparando dichos fragmentos con las lecturas únicas de referencia del mismo tamaño para cada uno de dichos cromosomas, en donde las lecturas únicas de referencia son fragmentos de un cromosoma que tiene una secuencia única la cual puede ser asignada de manera no ambigua a una única localización cromosómica individual con base en una secuencia genómica de referencia;
(c) determinar la profundidad de cobertura y el contenido de GC de un cromosoma con base en la información de secuencia para aquellos fragmentos que han sido asignados a las lecturas únicas de referencia de dicho cromosoma en la etapa (b), en donde la profundidad de cobertura es la relación entre el número de fragmentos asignados de manera única a dicho cromosoma y el número de lecturas únicas de referencia para dicho cromosoma del mismo tamaño del fragmento con base en dicha secuencia genómica humana de referencia;
(d) determinar la profundidad de cobertura ajustada de dicho cromosoma utilizando dicho contenido de GC de dicho cromosoma y la relación establecida entre la profundidad de cobertura y el contenido de GC para dicho cromosoma en la ausencia de aneuploidía, en donde dicha relación establecida ha sido determinada por un método que comprende las etapas de:
(i) obtener información de secuencia de múltiples fragmentos de polinucleótidos que cubren dicho cromosoma a partir de una pluralidad de muestras de sangre periférica euploide que contienen ADN genómico, en donde el tamaño del fragmento es el mismo que el tamaño del fragmento de los fragmentos de polinucleótido múltiples de la etapa (a) anterior;
(ii) asignar dichos fragmentos a cromosomas con base en dicha información de secuencia como en la etapa (b) más arriba;
(iii) determinar la profundidad de cobertura y el contenido de GC de dicho cromosoma con base en dicha información de secuencia para cada muestra euploide como en la etapa (c) más arriba; y
(iv) utilizar la profundidad de cobertura y el contenido de GC determinados para cada muestra en la etapa (iii) para determinar la relación entre la profundidad de cobertura y el contenido de GC de dicho cromosoma en la ausencia de aneuploidía; y
(e) comparar dicha profundidad de cobertura ajustada con la profundidad de cobertura de dicho cromosoma determinada en la etapa (c), en donde una diferencia entre ellos indica aneuploidía cromosómica fetal.
Tipo: Patente Internacional (Tratado de Cooperación de Patentes). Resumen de patente/invención. Número de Solicitud: PCT/CN2011/001070.
Solicitante: BGI Diagnosis Co., Ltd.
Nacionalidad solicitante: China.
Dirección: 2F, 3F, 11 Buildings, Beishan Industrial Zone, Beishan Road 146, Yantian District, Shenzhen Guangdong 518083 CHINA.
Inventor/es: CHEN,FANG, JIANG,FUMAN, CHEN,HUIFEI, CHAI,XIANGHUA, YUAN,YUYING, ZHANG,XIUQING.
Fecha de Publicación: .
Clasificación Internacional de Patentes:
- C12Q1/68 QUIMICA; METALURGIA. › C12 BIOQUIMICA; CERVEZA; BEBIDAS ALCOHOLICAS; VINO; VINAGRE; MICROBIOLOGIA; ENZIMOLOGIA; TECNICAS DE MUTACION O DE GENETICA. › C12Q PROCESOS DE MEDIDA, INVESTIGACION O ANALISIS EN LOS QUE INTERVIENEN ENZIMAS, ÁCIDOS NUCLEICOS O MICROORGANISMOS (ensayos inmunológicos G01N 33/53 ); COMPOSICIONES O PAPELES REACTIVOS PARA ESTE FIN; PROCESOS PARA PREPARAR ESTAS COMPOSICIONES; PROCESOS DE CONTROL SENSIBLES A LAS CONDICIONES DEL MEDIO EN LOS PROCESOS MICROBIOLOGICOS O ENZIMOLOGICOS. › C12Q 1/00 Procesos de medida, investigación o análisis en los que intervienen enzimas, ácidos nucleicos o microorganismos (aparatos de medida, investigación o análisis con medios de medida o detección de las condiciones del medio, p. ej. contadores de colonias, C12M 1/34 ); Composiciones para este fin; Procesos para preparar estas composiciones. › en los que intervienen ácidos nucleicos.
PDF original: ES-2512448_T3.pdf



Fragmento de la descripción:
Detección no invasiva de anormalidades genéticas fetales Campo técnico
La invención se relaciona con métodos no invasivos para la detección de anormalidades genéticas fetales, más particularmente aneuploidía cromosómica, mediante secuenciación de ADN de muestras de sangre periférica tomadas de mujeres embarazadas que contienen ADN tanto materno como fetal. Más particularmente, esta invención se relaciona con el análisis de datos que tiene en cuenta el sesgo de GC introducido por amplificación y secuenciación de muestras de ADN a través de bases sobre relaciones preestablecidas entre el contenido de GC y la profundidad de cobertura para un cromosoma en ausencia de aneuploidía y en el tamaño de fragmentación de ADN seleccionado como se discute en más detalle más adelante. Puede utilizarse análisis estadístico para comparar la profundidad de cobertura calculada para un cromosoma en una muestra con respecto a la profundidad de cobertura esperada de acuerdo con tal relación establecida con lo cual puede lograrse la detección de aneuploidía cromosómica fetal.
Técnica antecedente
Los métodos de diagnóstico prenatales convencionales con procedimientos invasivos, tales como muestreo de vilus coriónico y amniocentesis, conllevan riesgos potenciales tanto para los fetos como para las madres. La selección no invasiva de aneuploidía fetal utilizando marcadores en el suero materno y ultrasonido está disponible, pero tiene sensibilidad y especificidad limitadas (Kagan, et al., Human Reproduction (28) 23:1968-1975; Malone, et al., N Engl J Med (25) 353:21-211).
Estudios recientes han demostrado que la detección no invasiva de aneuploidía fetal mediante secuenciación paralela masiva de moléculas de ADN en el plasma de mujeres embarazadas es factible. El ADN fetal ha sido detectado y cuantificado en plasma y suero maternos (Lo, et al., Lancet (1997) 35:485 487; Lo, et al., Am. J. hum. Genet. (1998) 62:768-775). Se presentan múltiples tipos de células fetales en la circulación materna, incluyendo granulocitos, linfocitos, glóbulos rojos nucleados y células de trofoblastos fetales (Pertl y Bianchi, Obstetrics and Gynecology (21) 98:483-49). El ADN fetal puede ser detectado en el suero en la séptima semana de gestación, y se incrementa con el término del embarazo. El ADN fetal presente en el suero y en el plasma materno es comparable a la concentración de ADN obtenida de los protocolos de aislamiento de células fetales.
El ADN fetal en circulación ha sido utilizado para determinar el sexo del feto (Lo, et al., Am. J. hum. Genet. (1998) 62:768-775). También se ha detectado el genotipo D de Rhesus fetal utilizando ADN fetal. Sin embargo, el diagnóstico y las aplicaciones clínicas del ADN fetal en circulación están limitados a genes que están presentes en el feto pero no en la madre (Pertl y Bianchi, Obstetrics and Gynecology (21) 98:483-49). Así, existe una necesidad por un método no invasivo que pueda determinar la secuencia del ADN fetal y proveer un diagnóstico definitivo de las anormalidades cromosómicas en un feto.
El descubrimiento de las células fetales y de los ácidos nucleicos fetales libres de células en material sanguíneo en las últimas décadas y la aplicación de la secuenciación biolística de alto rendimiento de ADN libre de células en plasma materno lo hace disponible para detectar pequeños cambios en la representación de cromosomas aportados por un feto aneuploide en una muestra de plasma materno. Se ha alcanzado la detección no invasiva de embarazos portrisomía 13, 18 y 21.
Sin embargo, como lo muestran algunos estudios, el sesgo de GC introducido por la amplificación y secuenciación ha puesto un límite práctico sobre la sensibilidad de la detección de aneuploidía. El sesgo de GC podría ser introducido durante la preparación de la muestra y el proceso de secuenciación, bajo diferentes condiciones tales como composición de reactivos, densidad y temperatura de la aglomeración, lo cual lleva a un muestreo diferencial de las moléculas de ADN con diferente composición de GC y variación significativa en los datos de secuenciación para los cromosomas que son ricos en GC o pobres en GC.
Para mejorar la sensibilidad, se han desarrollado protocolos para la eliminación del efecto del sesgo de GC. Fan y Quake desarrollaron un método para eliminar computacionalmente el sesgo de GC aplicando peso a cada densidad de GC con base en el contenido GC genómico local, para mejorar el número de lecturas mapeadas en cada conjunto basura multiplicando el peso correspondiente (Fan y Quake PLoS ONE (21) 5: e1439). Sin embargo, el método tiene dificultades al tratar con trastornos en cromosomas sexuales, especialmente trastornos relevantes del cromosoma Y por la razón de que el proceso puede producir una ligera distorsión de datos la cual interfiere con la precisión de la detección.
Aquí, hemos descubierto un método que hace uso del sesgo de GC con el fin de obtener una alta sensibilidad en la detección de aneuploidía fetal así como para evitar la distorsión de los datos. Este método permite la definición de parámetros usados para pruebas estadísticas de acuerdo con el contenido de GC. Además, los inventores introdujeron la fracción fetal estimada en el diagnóstico mediante una hipótesis binaria la cual muestra sensibilidad y
especificidad más altas. Nuestro método también muestra que debe ser posible incrementar la sensibilidad de la detección no invasiva de anormalidades genéticas fetales para prefijar la precisión para muestras maternas que contienen una baja fracción de ADN fetal secuenciando más fragmentos de polinucleótidos. El remuestreo del plasma materno en las últimas semanas de la gestación también puede Incrementar la sensibilidad del diagnóstico.
Resumen de la invención
La presente invención está dirigida a métodos para la detección no invasiva de aneuploidía cromosómica fetal en los cuales se lleva a cabo un secuenciación a gran escala de nucleótidos a partir de muestras de sangre periférica materna. En un aspecto, la presente invención provee un método ¡mplementado por ordenador para determinar una anormalidad genética fetal la cual es una aneuploidía cromosómica o aneuploidía parcial, método que comprende:
(a) obtener la información de secuencia de múltiples fragmentos de pollnucleótldos a partir de una muestra, siendo dicha muestra una muestra de sangre periférica derivada de un sujeto femenino en embarazo y que contiene ADN tanto materno como fetal;
(b) asignar dichos fragmentos a cromosomas con base en dicha información de secuencia comparando dichos fragmentos con las lecturas únicas de referencia del mismo tamaño para cada uno de dichos cromosomas, en donde las lecturas únicas de referencia son fragmentos de un cromosoma que tiene una secuencia única la cual puede ser asignada de manera no ambigua a una localización cromosómica individual con base en una secuencia genómica de referencia;
(c) determinar la profundidad de cobertura y el contenido de GC de un cromosoma con base en la información de secuencia para aquellos fragmentos que han sido asignados a las lecturas únicas de referencia de dicho cromosoma en la etapa (b), en donde la profundidad de cobertura es la relación entre el número de fragmentos asignados de manera única a dicho cromosoma y el número de lecturas únicas de referencia para dicho cromosoma del mismo tamaño del fragmento con base en dicha secuencia genómica humana de referencia;
(d) determinar la profundidad de cobertura ajustada de dicho cromosoma utilizando dicho contenido de GC de dicho cromosoma y la relación establecida entre la profundidad de cobertura y el contenido de GC para dicho cromosoma en la ausencia de aneuploidía, en donde dicha relación establecida ha sido determinada por un método que comprende las etapas de:
(i) obtener información de secuencia de múltiples fragmentos de polinucleótidos que cubren dicho cromosoma a partir de una pluralidad de muestras de sangre periférica euploide que contienen ADN genómico, en donde el tamaño del fragmento es el mismo que el tamaño del fragmento de los fragmentos de polinucleótido múltiples de la etapa (a) anterior;
(¡i) asignar dichos fragmentos a cromosomas con base en dicha información de secuencia como en la etapa (b) más arriba;
(iii) determinar la profundidad de cobertura y el contenido de GC de dicho cromosoma con base en dicha información de secuencia para cada muestra euploide como en la etapa (c) más arriba; y
(¡v) utilizar la profundidad de cobertura y el contenido de GC determinados para cada muestra en la etapa (iii) para determinar la relación entre la profundidad de cobertura y el contenido de GC de dicho cromosoma... [Seguir leyendo]
Reivindicaciones:
1. Un método implementado por ordenador para determinar una anormalidad genética fetal la cual es una aneuploidía cromosómica, método que comprende:
(a) obtener la información de secuencia de múltiples fragmentos de polinucleótidos a partir de una muestra, siendo dicha muestra una muestra de sangre periférica derivada de un sujeto femenino en embarazo y que contiene ADN tanto materno como fetal;
(b) asignar dichos fragmentos a cromosomas con base en dicha información de secuencia comparando dichos fragmentos con las lecturas únicas de referencia del mismo tamaño para cada uno de dichos cromosomas, en donde las lecturas únicas de referencia son fragmentos de un cromosoma que tiene una secuencia única la cual puede ser asignada de manera no ambigua a una única localización cromosómica individual con base en una secuencia genómica de referencia;
(c) determinar la profundidad de cobertura y el contenido de GC de un cromosoma con base en la información de secuencia para aquellos fragmentos que han sido asignados a las lecturas únicas de referencia de dicho cromosoma en la etapa (b), en donde la profundidad de cobertura es la relación entre el número de fragmentos asignados de manera única a dicho cromosoma y el número de lecturas únicas de referencia para dicho cromosoma del mismo tamaño del fragmento con base en dicha secuencia genómica humana de referencia;
(d) determinar la profundidad de cobertura ajustada de dicho cromosoma utilizando dicho contenido de GC de dicho cromosoma y la relación establecida entre la profundidad de cobertura y el contenido de GC para dicho cromosoma en la ausencia de aneuploidía, en donde dicha relación establecida ha sido determinada por un método que comprende las etapas de:
(i) obtener información de secuencia de múltiples fragmentos de polinucleótidos que cubren dicho cromosoma a partir de una pluralidad de muestras de sangre periférica euploide que contienen ADN genómico, en donde el tamaño del fragmento es el mismo que el tamaño del fragmento de los fragmentos de polinucleótido múltiples de la
etapa (a) anterior;
(¡i) asignar dichos fragmentos a cromosomas con base en dicha información de secuencia como en la etapa (b) más
arriba;
(iii) determinar la profundidad de cobertura y el contenido de GC de dicho cromosoma con base en dicha información de secuencia para cada muestra euploide como en la etapa (c) más arriba; y
(iv) utilizar la profundidad de cobertura y el contenido de GC determinados para cada muestra en la etapa (iii) para determinar la relación entre la profundidad de cobertura y el contenido de GC de dicho cromosoma en la ausencia
de aneuploidía; y
(e) comparar dicha profundidad de cobertura ajustada con la profundidad de cobertura de dicho cromosoma determinada en la etapa (c), en donde una diferencia entre ellos indica aneuploidía cromosómica fetal.
2. El método de la reivindicación 1, en donde la etapa (a) comprende adicionalmente obtener información de secuencia de múltiples fragmentos de polinucleótidos a partir de muestras diferentes y la profundidad de cobertura es normalizada para tener en cuenta las diferencias en el número total de lecturas de secuencia obtenidas para diferentes muestras, por ejemplo contra la profundidad de cobertura promedio de otro cromosoma, preferiblemente contra la profundidad de cobertura promedio de todos los otros autosomas o contra la profundidad de cobertura promedio de todos los otros cromosomas.
3. El método de acuerdo con la reivindicación 1 o reivindicación 2, en donde el contenido de GC del cromosoma se determina como el contenido de GC promedio de todos los fragmentos que se asignan a dicho cromosoma para el propósito de la etapa (c).
4. El método de acuerdo con la reivindicación 2 que comprende adicionalmente determinar el género fetal, por ejemplo de acuerdo con la fórmula
logit(p() = ln(^) = & + fte/\a + facra. 1 ~P¡
donde cr.a.i,* y cr.aiy son la profundidad de cobertura relativa de los cromosomas X y Y de la muestra i, respectivamente.
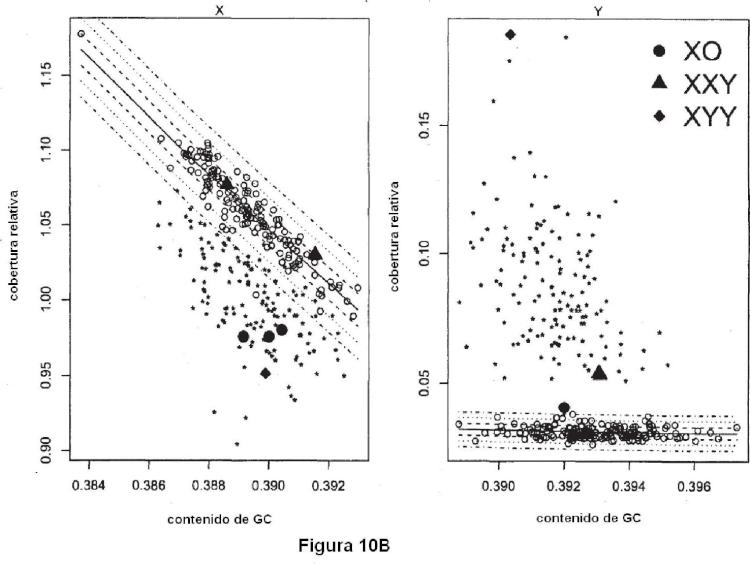
5. El método de acuerdo con la reivindicación 2 que comprende adlclonalmente estimar la fracción fetal, en donde la fracción fetal es calculada con base en la profundidad de la cobertura del cromosoma X y/o Y determinada como en la etapa (c) de la reivindicación 1 de acuerdo con una fórmula seleccionada de:
A / A A
fiV A = (Cn,Y -Cri,Yf)¡ (Cri,Ym ~Cri,Yf)
donde cr¡,Y es la profundidad de cobertura del cromosoma Y de la muestra /', cri,Yf~ %GC¡,Yf> es la profundidad de cobertura ajustada calculada a partir de la relación de la profundidad de la cobertura del cromosoma Y y el contenido
correspondiente de GC de las muestras de mujeres en embarazo con un feto femenino, y cri.Ym = AGC¡ym)
se refiere
a la profundidad de la cobertura ajustada calculada a partir de la relación de la profundidad de la cobertura del cromosoma Y y el contenido correspondiente de GC de sujetos masculinos; o
(ü) A = ~cn,xf)/(crUXm-criXf),
A
en donde cri}X es la profundidad de cobertura del cromosoma X de la muestra i, crw= es la profundidad de la
cobertura ajustada calculada a partir de la relación de la profundidad de cobertura del cromosoma X y el contenido
A
correspondiente de GC de las muestras de mujeres embarazadas con un feto femenino, crij<m = f(GCiXm) se refiere a la profundidad de la cobertura ajustada calculada a partir de la relación de la profundidad de cobertura del cromosoma X y el contenido correspondiente de GC de muestras de sujetos masculinos; o
(¡ü)
^. tyjff-Q-tO+cn,
m,,=aigmin(^^ A , ,
E<1) <Pxj (!-£)) +(.vx,m-s) (°iv'O-®))
i,júx & ctl,x) | (Aty ` g)+C/jfw £ CtJjr)
en donde crw ^GC¡,x¡> es la profundidad de la cobertura ajustada calculada a partir de la relación de la profundidad de cobertura del cromosoma X y el contenido correspondiente de GC de muestras de mujeres embarazadas con un
A
feto femenino, cr¡yt = i(GC¡,Yf> Se refiere a la profundidad de la cobertura ajustada calculada a partir de la relación de la profundidad de la cobertura del cromosoma Y y el contenido correspondiente de GC de muestras de mujeres
embarazadas con un feto femenino, cri,xm ~ %GCi,xm) Se refiere a la profundidad de la cobertura ajustada calculada a partir de la relación de la profundidad de cobertura del cromosoma X y el contenido correspondiente de GC de
muestras de sujetos masculinos, y cr¡,Yirt=^GGi,Ym> se refiere a la profundidad de cobertura ajustada calculada a partir de la relación de la profundidad de cobertura del cromosoma Y y el contenido correspondiente de GC de sujetos masculinos.
6. El método de acuerdo con la reivindicación 2, en donde la comparación de dicha profundidad de cobertura ajustada con la profundidad de cobertura del cromosoma determinada en la etapa (c) se lleva a cabo mediante una prueba de hipótesis estadística, en donde una hipótesis es que el feto es euploide (H) y la otra hipótesis es que el feto exhibe aneuploidía para dicho cromosoma (H1).
7. El método de la reivindicación 6, en donde la estadística t de Student se calcula para ambas hipótesis.
8. El método de la reivindicación 7, en donde la estadística t de Student se calcula para H y H1 de acuerdo con las fórmulas
th,j=(crij-cr¡tj)/stdj
y
t2ij =(«y -crij{Y + fxylJl2)))¡stdj
respectivamente, en donde fxy es la fracción fetal, i es el índice de muestra, j es el número de cromosomas,
crIJ=/(GC,J)+eIJ,j = l,2,-,22,X,Y
en donde f(GC¡,j) representa la función para la relación entre la profundidad de cobertura normalizada y el contenido correspondiente de GC de la muestra i, cromosoma j, e¡j representa el residual de la muestra i, cromosoma j,
cr,j =f(GCtJ)J = 1,2,-, 22, X, ¥
y representa la profundidad de cobertura ajustada, y stdj es la variación estándar de acuerdo con la fórmula
itd, = Je {cr,j -4 jfjins -1), j = 1,2,.... 22, X, Y
en donde ns representa el número de muestras de referencia.
9. El método de la reivindicación 8, en donde la relación de probabilidad logarítmica de t1 y t2 se calcula de acuerdo con la fórmula: Ly = log(p(t1y, grado |D))/log(p(t2y, grado|T)), en donde Ly es la relación de probabilidad logarítmica, en donde grado se refiere a un grado de distribución t, D se refiere a Diploidía, T se refiere a Trisomía, y p(t1y, grado |*),*=D, T representa la densidad de probabilidad condicional dada a un grado de distribución t, si la relación es más grande que 1 infiriéndose que el feto exhibe trisomía para dicho cromosoma.
1. El método de acuerdo con una cualquiera de las reivindicaciones 1 a 9 para uso en la determinación de aneuploidía en autosoma fetal.
11. El método de la reivindicación 1, en donde la aneuploidía fetal se selecciona del grupo consistente de trisomía 13, 18 y 21.
12. El método de acuerdo con la reivindicación 4 para uso en la determinación de aneuploidía de cromosoma sexual, tal como aneuploidía de cromosoma sexual seleccionada del grupo consistente de XO, XXX, XXY y XYY.
13. Un medio legible por ordenador que comprende una pluralidad de instrucciones adaptadas para ejecutar el método de una cualquiera de las reivindicaciones 1 a 12 cuando dichas instrucciones son suplementadas con información de secuencia obtenida como en la etapa (a) de la reivindicación 1.
14. Un sistema que comprende medios adaptados para ejecutar el método de una cualquiera de las reivindicaciones 1 a 12.
Patentes similares o relacionadas:
Método para analizar ácido nucleico molde, método para analizar sustancia objetivo, kit de análisis para ácido nucleico molde o sustancia objetivo y analizador para ácido nucleico molde o sustancia objetivo, del 29 de Julio de 2020, de Kabushiki Kaisha DNAFORM: Un método para analizar un ácido nucleico molde, que comprende las etapas de: fraccionar una muestra que comprende un ácido nucleico molde […]
MÉTODOS PARA EL DIAGNÓSTICO DE ENFERMOS ATÓPICOS SENSIBLES A COMPONENTES ALERGÉNICOS DEL POLEN DE OLEA EUROPAEA (OLIVO), del 23 de Julio de 2020, de SERVICIO ANDALUZ DE SALUD: Biomarcadores y método para el diagnostico, estratificación, seguimiento y pronostico de la evolución de la enfermedad alérgica a polen del olivo, kit […]
Detección de interacciones proteína a proteína, del 15 de Julio de 2020, de THE GOVERNING COUNCIL OF THE UNIVERSITY OF TORONTO: Un método para medir cuantitativamente la fuerza y la afinidad de una interacción entre una primera proteína de membrana o parte de la misma y una […]
Secuenciación dirigida y filtrado de UID, del 15 de Julio de 2020, de F. HOFFMANN-LA ROCHE AG: Un procedimiento para generar una biblioteca de polinucleótidos que comprende: (a) generar una primera secuencia del complemento (CS) de un polinucleótido diana a partir […]
Métodos para la recopilación, estabilización y conservación de muestras, del 8 de Julio de 2020, de Drawbridge Health, Inc: Un método para estabilizar uno o más componentes biológicos de una muestra biológica de un sujeto, comprendiendo el método obtener un […]
Evento de maíz DP-004114-3 y métodos para la detección del mismo, del 1 de Julio de 2020, de PIONEER HI-BRED INTERNATIONAL, INC.: Un amplicón que consiste en la secuencia de ácido nucleico de la SEQ ID NO: 32 o el complemento de longitud completa del mismo.
Composiciones para modular la expresión de SOD-1, del 24 de Junio de 2020, de Biogen MA Inc: Un compuesto antisentido según la siguiente fórmula: mCes Aeo Ges Geo Aes Tds Ads mCds Ads Tds Tds Tds mCds Tds Ads mCeo Aes Geo mCes Te (secuencia […]
Aislamiento de ácidos nucleicos, del 24 de Junio de 2020, de REVOLUGEN LIMITED: Un método de aislamiento de ácidos nucleicos que comprenden ADN de material biológico, comprendiendo el método las etapas que consisten en: (i) efectuar un lisado […]